+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | 1 ATP-bound V1EG of V/A-ATPase from Thermus thermophilus | ||||||||||||||||||
 マップデータ マップデータ | Masked map of 1 ATP-bound V1EG of V/A-ATPase | ||||||||||||||||||
 試料 試料 |
| ||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報proton-transporting two-sector ATPase complex, catalytic domain / proton-transporting ATP synthase complex / proton motive force-driven plasma membrane ATP synthesis /  ATP合成酵素 / proton-transporting ATPase activity, rotational mechanism / proton-transporting ATP synthase activity, rotational mechanism / ATP合成酵素 / proton-transporting ATPase activity, rotational mechanism / proton-transporting ATP synthase activity, rotational mechanism /  ATP binding / ATP binding /  metal ion binding metal ion binding類似検索 - 分子機能 | ||||||||||||||||||
| 生物種 |    Thermus thermophilus HB8 (サーマス・サーモフィルス) Thermus thermophilus HB8 (サーマス・サーモフィルス) | ||||||||||||||||||
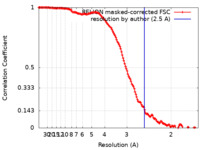
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 2.5 Å クライオ電子顕微鏡法 / 解像度: 2.5 Å | ||||||||||||||||||
 データ登録者 データ登録者 | Nakanishi A / Kishikawa J / Mitsuoka K / Yokoyama K | ||||||||||||||||||
| 資金援助 |  日本, 5件 日本, 5件
| ||||||||||||||||||
 引用 引用 |  ジャーナル: J Biol Chem / 年: 2023 ジャーナル: J Biol Chem / 年: 2023タイトル: Cryo-EM analysis of V/A-ATPase intermediates reveals the transition of the ground-state structure to steady-state structures by sequential ATP binding. 著者: Atsuko Nakanishi / Jun-Ichi Kishikawa / Kaoru Mitsuoka / Ken Yokoyama /  要旨: Vacuolar/archaeal-type ATPase (V/A-ATPase) is a rotary ATPase that shares a common rotary catalytic mechanism with FF ATP synthase. Structural images of V/A-ATPase obtained by single-particle cryo- ...Vacuolar/archaeal-type ATPase (V/A-ATPase) is a rotary ATPase that shares a common rotary catalytic mechanism with FF ATP synthase. Structural images of V/A-ATPase obtained by single-particle cryo-electron microscopy during ATP hydrolysis identified several intermediates, revealing the rotary mechanism under steady-state conditions. However, further characterization is needed to understand the transition from the ground state to the steady state. Here, we identified the cryo-electron microscopy structures of V/A-ATPase corresponding to short-lived initial intermediates during the activation of the ground state structure by time-resolving snapshot analysis. These intermediate structures provide insights into how the ground-state structure changes to the active, steady state through the sequential binding of ATP to its three catalytic sites. All the intermediate structures of V/A-ATPase adopt the same asymmetric structure, whereas the three catalytic dimers adopt different conformations. This is significantly different from the initial activation process of FF, where the overall structure of the F domain changes during the transition from a pseudo-symmetric to a canonical asymmetric structure (PNAS NEXUS, pgac116, 2022). In conclusion, our findings provide dynamical information that will enhance the future prospects for studying the initial activation processes of the enzymes, which have unknown intermediate structures in their functional pathway. | ||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_34362.map.gz emd_34362.map.gz | 20.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-34362-v30.xml emd-34362-v30.xml emd-34362.xml emd-34362.xml | 23.3 KB 23.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_34362_fsc.xml emd_34362_fsc.xml | 14.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_34362.png emd_34362.png | 106.1 KB | ||
| マスクデータ |  emd_34362_msk_1.map emd_34362_msk_1.map | 244.1 MB |  マスクマップ マスクマップ | |
| その他 |  emd_34362_half_map_1.map.gz emd_34362_half_map_1.map.gz emd_34362_half_map_2.map.gz emd_34362_half_map_2.map.gz | 194.2 MB 194.2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-34362 http://ftp.pdbj.org/pub/emdb/structures/EMD-34362 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-34362 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-34362 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_34362.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_34362.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Masked map of 1 ATP-bound V1EG of V/A-ATPase | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.83 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_34362_msk_1.map emd_34362_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Unfiltered half map of 1 ATP-bound V1EG of V/A-ATPase
| ファイル | emd_34362_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unfiltered half map of 1 ATP-bound V1EG of V/A-ATPase | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Unfiltered half map of 1 ATP-bound V1EG of V/A-ATPase
| ファイル | emd_34362_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unfiltered half map of 1 ATP-bound V1EG of V/A-ATPase | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : 1 ATP-bound V1EG of V/A-ATPase from Thermus thermophilus
| 全体 | 名称: 1 ATP-bound V1EG of V/A-ATPase from Thermus thermophilus |
|---|---|
| 要素 |
|
-超分子 #1: 1 ATP-bound V1EG of V/A-ATPase from Thermus thermophilus
| 超分子 | 名称: 1 ATP-bound V1EG of V/A-ATPase from Thermus thermophilus タイプ: complex / ID: 1 / キメラ: Yes / 親要素: 0 / 含まれる分子: #1-#6 |
|---|---|
| 由来(天然) | 生物種:    Thermus thermophilus HB8 (サーマス・サーモフィルス) Thermus thermophilus HB8 (サーマス・サーモフィルス) |
| 分子量 | 理論値: 600 KDa |
-分子 #1: V-type ATP synthase alpha chain
| 分子 | 名称: V-type ATP synthase alpha chain / タイプ: protein_or_peptide / ID: 1 詳細: Authors state the bacterium they used has two mutations in its genome (S232A and T235S introduced by homologous recombination with engineered template DNA). コピー数: 3 / 光学異性体: LEVO / EC番号:  ATP合成酵素 ATP合成酵素 |
|---|---|
| 由来(天然) | 生物種:    Thermus thermophilus HB8 (サーマス・サーモフィルス) Thermus thermophilus HB8 (サーマス・サーモフィルス)株: HB8 |
| 分子量 | 理論値: 63.669957 KDa |
| 配列 | 文字列: MIQGVIQKIA GPAVIAKGML GARMYDICKV GEEGLVGEII RLDGDTAFVQ VYEDTSGLKV GEPVVSTGLP LAVELGPGML NGIYDGIQR PLERIREKTG IYITRGVVVH ALDREKKWAW TPMVKPGDEV RGGMVLGTVP EFGFTHKILV PPDVRGRVKE V KPAGEYTV ...文字列: MIQGVIQKIA GPAVIAKGML GARMYDICKV GEEGLVGEII RLDGDTAFVQ VYEDTSGLKV GEPVVSTGLP LAVELGPGML NGIYDGIQR PLERIREKTG IYITRGVVVH ALDREKKWAW TPMVKPGDEV RGGMVLGTVP EFGFTHKILV PPDVRGRVKE V KPAGEYTV EEPVVVLEDG TELKMYHTWP VRRARPVQRK LDPNTPFLTG MRILDVLFPV AMGGTAAIPG PFGAGKSVTQ QS LAKWSNA DVVVYVGCGE RGNEMTDVLV EFPELTDPKT GGPLMHRTVL IANTSNMPVA AREASIYVGV TIAEYFRDQG FSV ALMADS TSRWAEALRE ISSRLEEMPA EEGYPPYLAA RLAAFYERAG KVITLGGEEG AVTIVGAVSP PGGDMSEPVT QSTL RIVGA FWRLDASLAF RRHFPAINWN GSYSLFTSAL DPWYRENVAE DYPELRDAIS ELLQREAGLQ EIVQLVGPDA LQDAE RLVI EVGRIIREDF LQQNAYHEVD AYCSMKKAYG IMKMILAFYK EAEAAIKRGV SIDEILQLPV LERIGRARYV SEEEFP AYF EEAMKEIQGA FKALA |
-分子 #2: V-type ATP synthase beta chain
| 分子 | 名称: V-type ATP synthase beta chain / タイプ: protein_or_peptide / ID: 2 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:    Thermus thermophilus HB8 (サーマス・サーモフィルス) Thermus thermophilus HB8 (サーマス・サーモフィルス)株: HB8 |
| 分子量 | 理論値: 53.2195 KDa |
| 配列 | 文字列: MDLLKKEYTG ITYISGPLLF VENAKDLAYG AIVDIKDGTG RVRGGQVIEV SEEYAVIQVF EETTGLDLAT TSVSLVEDVA RLGVSKEML GRRFNGIGKP IDGLPPITPE KRLPITGLPL NPVARRKPEQ FIQTGISTID VMNTLVRGQK LPIFSGSGLP A NEIAAQIA ...文字列: MDLLKKEYTG ITYISGPLLF VENAKDLAYG AIVDIKDGTG RVRGGQVIEV SEEYAVIQVF EETTGLDLAT TSVSLVEDVA RLGVSKEML GRRFNGIGKP IDGLPPITPE KRLPITGLPL NPVARRKPEQ FIQTGISTID VMNTLVRGQK LPIFSGSGLP A NEIAAQIA RQATVRPDLS GEGEKEEPFA VVFAAMGITQ RELSYFIQEF ERTGALSRSV LFLNKADDPT IERILTPRMA LT VAEYLAF EHDYHVLVIL TDMTNYCEAL REIGAAREEI PGRRGYPGYM YTDLATIYER AGVVEGKKGS VTQIPILSMP DDD RTHPIP DLTGYITEGQ IQLSRELHRK GIYPPIDPLP SLSRLMNNGV GKGKTREDHK QVSDQLYSAY ANGVDIRKLV AIIG EDALT ENDRRYLQFA DAFERFFINQ GQQNRSIEES LQIAWALLSM LPQGELKRIS KDHIGKYYGQ KLEEIWGAPQ ALD |
-分子 #3: V-type ATP synthase subunit D
| 分子 | 名称: V-type ATP synthase subunit D / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:    Thermus thermophilus HB8 (サーマス・サーモフィルス) Thermus thermophilus HB8 (サーマス・サーモフィルス)株: HB8 |
| 分子量 | 理論値: 24.715566 KDa |
| 配列 | 文字列: MSQVSPTRMN LLQRRGQLRL AQKGVDLLKK KRDALVAEFF GLVREAMEAR KALDQAAKEA YAALLLAQAF DGPEVVAGAA LGVPPLEGV EAEVENVWGS KVPRLKATFP DGALLSPVGT PAYTLEASRA FRRYAEALIR VANTETRLKK IGEEIKKTTR R VNALEQVV ...文字列: MSQVSPTRMN LLQRRGQLRL AQKGVDLLKK KRDALVAEFF GLVREAMEAR KALDQAAKEA YAALLLAQAF DGPEVVAGAA LGVPPLEGV EAEVENVWGS KVPRLKATFP DGALLSPVGT PAYTLEASRA FRRYAEALIR VANTETRLKK IGEEIKKTTR R VNALEQVV IPGIRAQIRF IQQVLEQRER EDTFRLKRIK GKIEAREAEE EGGRPNPQVE IGAGL |
-分子 #4: V-type ATP synthase subunit F
| 分子 | 名称: V-type ATP synthase subunit F / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:    Thermus thermophilus HB8 (サーマス・サーモフィルス) Thermus thermophilus HB8 (サーマス・サーモフィルス)株: HB8 |
| 分子量 | 理論値: 11.294904 KDa |
| 配列 | 文字列: MAVIADPETA QGFRLAGLEG YGASSAEEAQ SLLETLVERG GYALVAVDEA LLPDPERAVE RLMRGRDLPV LLPIAGLKEA FQGHDVEGY MRELVRKTIG FDIKL |
-分子 #5: V-type ATP synthase, subunit (VAPC-THERM)
| 分子 | 名称: V-type ATP synthase, subunit (VAPC-THERM) / タイプ: protein_or_peptide / ID: 5 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:    Thermus thermophilus HB8 (サーマス・サーモフィルス) Thermus thermophilus HB8 (サーマス・サーモフィルス)株: HB8 |
| 分子量 | 理論値: 13.166218 KDa |
| 配列 | 文字列: MTGGLVLNAI SRAGGAMGGL GLIKSLAEKE KQLLERLEAA KKEAEERVKR AEAEAKALLE EAEAKAKALE AQYRERERAE TEALLARYR ERAEAEAKAV REKAMARLDE AVALVLKEVL P |
-分子 #6: V-type ATP synthase subunit E
| 分子 | 名称: V-type ATP synthase subunit E / タイプ: protein_or_peptide / ID: 6 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:    Thermus thermophilus HB8 (サーマス・サーモフィルス) Thermus thermophilus HB8 (サーマス・サーモフィルス)株: HB8 |
| 分子量 | 理論値: 20.645582 KDa |
| 配列 | 文字列: MSKLEAILSQ EVEAEIQALL QEAEAKAEAV KREAEEKAKA LLQARERALE AQYRAALRRA ESAGELLVAT ARTQARGEVL EEVRRRVRE ALEALPQKPE WPEVVRKLAL EALEALPGAK ALVANPEDLP HLEALARERG VELQAEPALR LGVRAVGAEG K TQVENSLL ARLDRAWDAL SSKVAQALWG |
-分子 #7: SULFATE ION
| 分子 | 名称: SULFATE ION / タイプ: ligand / ID: 7 / コピー数: 2 / 式: SO4 |
|---|---|
| 分子量 | 理論値: 96.063 Da |
| Chemical component information |  ChemComp-SO4: |
-分子 #8: ADENOSINE-5'-TRIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-TRIPHOSPHATE / タイプ: ligand / ID: 8 / コピー数: 1 / 式: ATP |
|---|---|
| 分子量 | 理論値: 507.181 Da |
| Chemical component information |  ChemComp-ATP: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 4.0 mg/mL |
|---|---|
| 緩衝液 | pH: 8 |
| グリッド | モデル: Quantifoil / 材質: MOLYBDENUM / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 60 sec. |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 1.8 µm / 最小 デフォーカス(公称値): 0.8 µm Bright-field microscopy / 最大 デフォーカス(公称値): 1.8 µm / 最小 デフォーカス(公称値): 0.8 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 50.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー
















 Z
Z Y
Y X
X