+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of RNA polymerase in complex with P protein tetramer of Newcastle disease virus | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  cryo-EM (低温電子顕微鏡法) / L-P complex / Newcastle disease virus / cryo-EM (低温電子顕微鏡法) / L-P complex / Newcastle disease virus /  VIRUS (ウイルス) VIRUS (ウイルス) | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報GDP polyribonucleotidyltransferase /  加水分解酵素; 酸無水物に作用; リン含有酸無水物に作用 / 加水分解酵素; 酸無水物に作用; リン含有酸無水物に作用 /  転移酵素; 一炭素原子の基を移すもの; メチル基を移すもの / 転移酵素; 一炭素原子の基を移すもの; メチル基を移すもの /  virion component / mRNA 5'-cap (guanine-N7-)-methyltransferase activity / host cell cytoplasm / virion component / mRNA 5'-cap (guanine-N7-)-methyltransferase activity / host cell cytoplasm /  RNA依存性RNAポリメラーゼ / RNA依存性RNAポリメラーゼ /  RNA-dependent RNA polymerase activity / RNA-dependent RNA polymerase activity /  GTPase activity / GTPase activity /  ATP binding / ATP binding /  細胞質 細胞質類似検索 - 分子機能 | |||||||||
| 生物種 |   Avian orthoavulavirus 1 (ニューカッスル病) Avian orthoavulavirus 1 (ニューカッスル病) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.25 Å クライオ電子顕微鏡法 / 解像度: 3.25 Å | |||||||||
 データ登録者 データ登録者 | Chen Y / Jingyuan C / Xiaoying F | |||||||||
| 資金援助 |  中国, 1件 中国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2023 ジャーナル: Nat Commun / 年: 2023タイトル: Structure of the Newcastle Disease Virus L protein in complex with tetrameric phosphoprotein. 著者: Jingyuan Cong / Xiaoying Feng / Huiling Kang / Wangjun Fu / Lei Wang / Chenlong Wang / Xuemei Li / Yutao Chen / Zihe Rao /  要旨: Newcastle disease virus (NDV) belongs to Paramyxoviridae, which contains lethal human and animal pathogens. NDV RNA genome is replicated and transcribed by a multifunctional 250 kDa RNA-dependent ...Newcastle disease virus (NDV) belongs to Paramyxoviridae, which contains lethal human and animal pathogens. NDV RNA genome is replicated and transcribed by a multifunctional 250 kDa RNA-dependent RNA polymerase (L protein). To date, high-resolution structure of NDV L protein complexed with P protein remains to be elucidated, limiting our understanding of the molecular mechanisms of Paramyxoviridae replication/transcription. Here, we used cryo-EM and enzymatic assays to investigate the structure-function relationship of L-P complex. We found that C-terminal of CD-MTase-CTD module of the atomic-resolution L-P complex conformationally rearranges, and the priming/intrusion loops are likely in RNA elongation conformations different from previous structures. The P protein adopts a unique tetrameric organization and interacts with L protein. Our findings indicate that NDV L-P complex represents elongation state distinct from previous structures. Our work greatly advances the understanding of Paramyxoviridae RNA synthesis, revealing how initiation/elongation alternates, providing clues for identifying therapeutic targets against Paramyxoviridae. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_33988.map.gz emd_33988.map.gz | 96.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-33988-v30.xml emd-33988-v30.xml emd-33988.xml emd-33988.xml | 16.5 KB 16.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_33988.png emd_33988.png | 37.3 KB | ||
| その他 |  emd_33988_half_map_1.map.gz emd_33988_half_map_1.map.gz emd_33988_half_map_2.map.gz emd_33988_half_map_2.map.gz | 80.8 MB 80.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-33988 http://ftp.pdbj.org/pub/emdb/structures/EMD-33988 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-33988 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-33988 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_33988.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_33988.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.04 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_33988_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
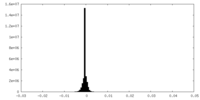
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_33988_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
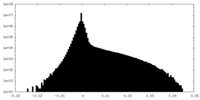
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : NDV L-P complex
| 全体 | 名称: NDV L-P complex |
|---|---|
| 要素 |
|
-超分子 #1: NDV L-P complex
| 超分子 | 名称: NDV L-P complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:   Avian orthoavulavirus 1 (ニューカッスル病) Avian orthoavulavirus 1 (ニューカッスル病) |
-分子 #1: NDV P protein
| 分子 | 名称: NDV P protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Avian orthoavulavirus 1 (ニューカッスル病) Avian orthoavulavirus 1 (ニューカッスル病) |
| 分子量 | 理論値: 41.738379 KDa |
| 組換発現 | 生物種:   Spodoptera frugiperda (ツマジロクサヨトウ) Spodoptera frugiperda (ツマジロクサヨトウ) |
| 配列 | 文字列: MATFTDAEID ELFETSGTVI DSIITAQGKP VETVGRSAIP QGKTKALSLA WEKHGNTNTP AAQESAGEQD QHGQNQASNS NRATPEEGP HSSQAQAATQ PQEDANESQL KTGASSSLLS MLDKLSNKSS NAKKGPPQSP PQQALHSKGS PAVEQTQHGA N QGRAQQET ...文字列: MATFTDAEID ELFETSGTVI DSIITAQGKP VETVGRSAIP QGKTKALSLA WEKHGNTNTP AAQESAGEQD QHGQNQASNS NRATPEEGP HSSQAQAATQ PQEDANESQL KTGASSSLLS MLDKLSNKSS NAKKGPPQSP PQQALHSKGS PAVEQTQHGA N QGRAQQET GHQAAPSPGP PGTGVNIAFP GQRGVSPQSV GATQPAPQSG QNQGSTPASA DHVQPPVDFV QAMMSMMEAI SQ RVSKIDY QLDLVLKQTS SIPTMRSEIQ QLKTSVAVME ANLGMMKILD PGCANVSSLS DLRAVAKSHP VLIAGPGDPS PYV TQGGEI ALNKLSQPVP HPSDLIKHAT SGGPDIGIER DTVRALILSR PMHPSSSSKL LSKLDSAGSV EEIRKIKRLA LNG UniProtKB:  Phosphoprotein Phosphoprotein |
-分子 #2: RNA-directed RNA polymerase L
| 分子 | 名称: RNA-directed RNA polymerase L / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO / EC番号:  RNA依存性RNAポリメラーゼ RNA依存性RNAポリメラーゼ |
|---|---|
| 由来(天然) | 生物種:   Avian orthoavulavirus 1 (ニューカッスル病) Avian orthoavulavirus 1 (ニューカッスル病) |
| 分子量 | 理論値: 249.183188 KDa |
| 組換発現 | 生物種:   Spodoptera frugiperda (ツマジロクサヨトウ) Spodoptera frugiperda (ツマジロクサヨトウ) |
| 配列 | 文字列: MAGSGSERAE HQIILPESHL SSPLVKHKLL YYWKLTGLPL PDECDFDHLI LSRQWKKILE SSTPDIERMI KLGRSVHQTL SHSSKLTGI LHPRCLEDLV GLDIPDSTNK FRRIEKKIQI HNTRYGEPFT RLCSYVEKKL LGSSWTHKIR RSEEFDSLRT D PAFWFHSS ...文字列: MAGSGSERAE HQIILPESHL SSPLVKHKLL YYWKLTGLPL PDECDFDHLI LSRQWKKILE SSTPDIERMI KLGRSVHQTL SHSSKLTGI LHPRCLEDLV GLDIPDSTNK FRRIEKKIQI HNTRYGEPFT RLCSYVEKKL LGSSWTHKIR RSEEFDSLRT D PAFWFHSS WSTAKFAWLH VKQIQRHLIV AARTRSASNK LVTLSHRSGQ VFITPELVIV THTNENKFTC LSQELVLMYA DM MEGRDMV NIISSTAVHL RCLAEKIDDI LRLVDALARD LGNQVYDVVA LMEGFAYGAV QLLEPSGTFA GDFFSFNLQE LRD TLICLL PQRIADSVTH AIANIFSGLE QNQAAEMLCL LRLWGHPLLE SRAAAKAVRA QMCAPKMVDF DMILQVLSFF KGTI INGYR KKNAGVWPRV KAHTIYGNVI AQLHADSAEI SHDIMLREYK NLSAIEFEAC IEYDPVTNLS MFLKDKAIAH PRNNW LASF RRNLLSEEQK KNVQDSTSTN RLLIEFLESN DFDPYKEMEY LTTLEYLRDD SVAVSYSLKE EEVKVNGRIF AKLTKK LRN CQVMAEGILA DQIAPFFQGN GVIQDSISLT KSMLAMSQLS YNSNRKRITD CKERVSSSRN HDLKGKHRRR VATFITT DL QKYCLNWRYQ TIKLFAHAIN QLMGLPHFFE WIHLRLMDTT MFVGDPFNPP SDPTDYDLTK VPNDDIYIVS ARGGIEGL C QKLWTMISIA AIQLAAARSH CRVACMVQGD NQVIAVTREV RPDDSPESVL TQLHEASDNF FRELIHVNHL IGHNLKDRE TIRSDTFFIY SKRIFKDGAI LSQVLKNSSK LVLVSGDLSE NTVMSCANIS STVARLCENG LPKDFCYYLN YLMSCIQTYF DSEFSITSS TQSGSNQSWI NDIPFIHSYV LTPAQLGGLS NLQYSRLYTR NIGDPGTTAF AEVKRLEAVG LLGPNIMTNI L TRPPGNGD WASLCNDPYS FNFESVASPS IVLKKHTQRV LFETCSNPLL SGVHTEDNEA EEKALAEYLL NQEVIHPRVA HA IMEASSV GRRKQIQGLV DTTNTVIKIA LSRKPLGIKR LARIINYSSM HAMLFRDDVF LSNRANHPLV SSDMCSLALA DYA RNRSWS PLTGGRKILG VSNPDTIELV EGEILSISGG CSKCDSGDEQ FTWFHLPSNI ELTDDTSKNP PMRVPYLGSK TQER RAASL AKIAHMSPHV KAALRASSVL IWAYGDNDIN WTAALKLARS RCNISSEYLR LLSPLPTAGN LQHRLDDGIT QMTFT PASL YRVSPYVHIS NDSQRLFTEE GVKEGNVVYQ QIMLLGLSLI ESLFPMTVTK TYDEITLHLH SKFSCCIREA PVAVPF ELT GVAPDLRVVA SNKFMYDPNP VAEGDFARLD LAIFKSYELN LESYSTVELM NILSISSGKL IGQSVVSYDE ETSIKND AI IVYDNTRNWI SEAQNSDVVR LFEYAALEVL LDCSYQLYYL RVRGLNNVVL YMSDLYKNMP GILLSNIAAT ISHPIIHS R LHTVGLISHD GSHQLADTDF IELSAKLLVS CTRRVVSGLY AGNKYDLLFP SVLDDNLNEK MLQLISRLCC LYTVLFATT REIPKIRGLP AEEKCAMLTE YLLSDAVRPL LSPEQVDSIT SPSIVTFPAN LYYMSRKSLN LIREREDRDS ILALMFPQEP LFEFPLVQD IGARVKDQLT MKPAAFLHEL DLSAPARYDA YTLEQARSDC ALADMGEDQL VRYLFRGVGT ASSSWYKASH L LSVPEIRC ARHGNSLYLA EGSGAIMSLL ELHIPHETIY YNTLFSNEMN PPQRHFGPTP TQFLNSVVYR NLQAEVPCKD GF VQEFRTL WRENTEESDL TSDKAVGYIT SVVPYRSVSL LHCDIEIPPG SNQSLLDQLA TNLSLIAMHS VREGGVVIVK ILY SMGYYF HLLVNLFTPC SVKGYVLSNG YACRGDMECY VVFVMGYLGG PTFVNEVVRM AKTLIQRHGT LLAKSDETAL MALF TSQKQ RVDNILSSPL PRLAKLLRRN IDTALIEAGG QPVRPFCAES LVNTLSDITQ TTQVIASHID TVIRSVIYME AEGDL ADTV FLFTPYNLSI DGKKRTSLKQ CTRQILEVTI LGLGPEDLNR VGDIISLILR GTISLEDLIP LRTYLKMSTC PKYLKS VLG LTKLREMFSD GSMLYLTRAQ QKFYMKTVGN AVKGYYNSSK NENLYFQG UniProtKB: RNA-directed RNA polymerase L |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | 3D array |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.2 µm Bright-field microscopy / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.2 µm |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 60.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: NONE |
|---|---|
| 初期 角度割当 | タイプ: ANGULAR RECONSTITUTION |
| 最終 角度割当 | タイプ: ANGULAR RECONSTITUTION |
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.25 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 153000 |
 ムービー
ムービー コントローラー
コントローラー










 Z
Z Y
Y X
X