+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of SbCas7-11 in complex with crRNA and target RNA | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 | CRISPR type III-associated protein / RAMP superfamily / defense response to virus /  RNA binding / RAMP superfamily protein RNA binding / RAMP superfamily protein 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |   Candidatus Scalindua brodae (バクテリア) Candidatus Scalindua brodae (バクテリア) | |||||||||
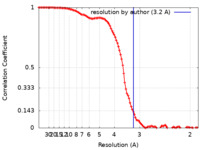
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.2 Å クライオ電子顕微鏡法 / 解像度: 3.2 Å | |||||||||
 データ登録者 データ登録者 | Yu G / Wang X / Deng Z / Zhang H | |||||||||
| 資金援助 |  中国, 1件 中国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Microbiol / 年: 2022 ジャーナル: Nat Microbiol / 年: 2022タイトル: Structure and function of a bacterial type III-E CRISPR-Cas7-11 complex. 著者: Guimei Yu / Xiaoshen Wang / Yi Zhang / Qiyin An / Yanan Wen / Xuzichao Li / Hang Yin / Zengqin Deng / Heng Zhang /  要旨: The type III-E CRISPR-Cas system uses a single multidomain effector called Cas7-11 (also named gRAMP) to cleave RNA and associate with a caspase-like protease Csx29, showing promising potential for ...The type III-E CRISPR-Cas system uses a single multidomain effector called Cas7-11 (also named gRAMP) to cleave RNA and associate with a caspase-like protease Csx29, showing promising potential for RNA-targeting applications. The structural and molecular mechanisms of the type III-E CRISPR-Cas system remain poorly understood. Here we report four cryo-electron microscopy structures of Cas7-11 at different functional states. Cas7-11 has four Cas7-like domains, which assemble into a helical filament to accommodate CRISPR RNA (crRNA), and a Cas11-like domain facilitating crRNA-target RNA duplex formation. The Cas7.1 domain is critical for crRNA maturation, whereas Cas7.2 and Cas7.3 are responsible for target RNA cleavage. Target RNA binding induces the structural arrangements of Csx29, potentially exposing the catalytic site of Csx29. These results delineate the molecular mechanisms underlying pre-crRNA processing, target RNA recognition and cleavage for Cas7-11, and provide a structural framework to understand the role of Csx29 in type III-E CRISPR system. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_33040.map.gz emd_33040.map.gz | 94.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-33040-v30.xml emd-33040-v30.xml emd-33040.xml emd-33040.xml | 16.2 KB 16.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_33040_fsc.xml emd_33040_fsc.xml | 13.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_33040.png emd_33040.png | 111.1 KB | ||
| その他 |  emd_33040_half_map_1.map.gz emd_33040_half_map_1.map.gz emd_33040_half_map_2.map.gz emd_33040_half_map_2.map.gz | 95.6 MB 95.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-33040 http://ftp.pdbj.org/pub/emdb/structures/EMD-33040 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-33040 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-33040 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_33040.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_33040.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 0.95 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: unmasked
| ファイル | emd_33040_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | unmasked | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: unmasked
| ファイル | emd_33040_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | unmasked | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : binary complex
| 全体 | 名称: binary complex |
|---|---|
| 要素 |
|
-超分子 #1: binary complex
| 超分子 | 名称: binary complex / タイプ: complex / キメラ: Yes / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:   Candidatus Scalindua brodae (バクテリア) Candidatus Scalindua brodae (バクテリア) |
-分子 #1: RAMP superfamily protein
| 分子 | 名称: RAMP superfamily protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Candidatus Scalindua brodae (バクテリア) Candidatus Scalindua brodae (バクテリア) |
| 分子量 | 理論値: 197.823797 KDa |
| 組換発現 | 生物種:   Escherichia coli BL21(DE3) (大腸菌) Escherichia coli BL21(DE3) (大腸菌) |
| 配列 | 文字列: MKSNDMNITV ELTFFEPYRL VEWFDWDARK KSHSAMRGQA FAQWTWKGKG RTAGKSFITG TLVRSAVIKA VEELLSLNNG KWEGVPCCN GSFQTDESKG KKPSFLRKRH TLQWQANNKN ICDKEEACPF CILLGRFDNA GKVHERNKDY DIHFSNFDLD H KQEKNDLR ...文字列: MKSNDMNITV ELTFFEPYRL VEWFDWDARK KSHSAMRGQA FAQWTWKGKG RTAGKSFITG TLVRSAVIKA VEELLSLNNG KWEGVPCCN GSFQTDESKG KKPSFLRKRH TLQWQANNKN ICDKEEACPF CILLGRFDNA GKVHERNKDY DIHFSNFDLD H KQEKNDLR LVDIASGRIL NRVDFDTGKA KDYFRTWEAD YETYGTYTGR ITLRNEHAKK LLLASLGFVD KLCGALCRIE VI KKSESPL PSDTKEQSYT KDDTVEVLSE DHNDELRKQA EVIVEAFKQN DKLEKIRILA DAIRTLRLHG EGVIEKDELP DGK EERDKG HHLWDIKVQG TALRTKLKEL WQSNKDIGWR KFTEMLGSNL YLIYKKETGG VSTRFRILGD TEYYSKAHDS EGSD LFIPV TPPEGIETKE WIIVGRLKAA TPFYFGVQQP SDSIPGKEKK SEDSLVINEH TSFNILLDKE NRYRIPRSAL RGALR RDLR TAFGSGCNVS LGGQILCNCK VCIEMRRITL KDSVSDFSEP PEIRYRIAKN PGTATVEDGS LFDIEVGPEG LTFPFV LRY RGHKFPEQLS SVIRYWEEND GKNGMAWLGG LDSTGKGRFA LKDIKIFEWD LNQKINEYIK ERGMRGKEKE LLEMGES SL PDGLIPYKFF EERECLFPYK ENLKPQWSEV QYTIEVGSPL LTADTISALT EPGNRDAIAY KKRVYNDGNN AIEPEPRF A VKSETHRGIF RTAVGRRTGD LGKEDHEDCT CDMCIIFGNE HESSKIRFED LELINGNEFE KLEKHIDHVA IDRFTGGAL DKAKFDTYPL AGSPKKPLKL KGRFWIKKGF SGDHKLLITT ALSDIRDGLY PLGSKGGVGY GWVAGISIDD NVPDDFKEMI NKTEMPLPE EVEESNNGPI NNDYVHPGHQ SPKQDHKNKN IYYPHYFLDS GSKVYREKDI ITHEEFTEEL LSGKINCKLE T LTPLIIPD TSDENGLKLQ GNKPGHKNYK FFNINGELMI PGSELRGMLR THFEALTKSC FAIFGEDSTL SWRMNADEKD YK IDSNSIR KMESQRNPKY RIPDELQKEL RNSGNGLFNR LYTSERRFWS DVSNKFENSI DYKREILRCA GRPKNYKGGI IRQ RKDSLM AEELKVHRLP LYDNFDIPDS AYKANDHCRK SATCSTSRGC RERFTCGIKV RDKNRVFLNA ANNNRQYLNN IKKS NHDLY LQYLKGEKKI RFNSKVITGS ERSPIDVIAE LNERGRQTGF IKLSGLNNSN KSQGNTGTTF NSGWDRFELN ILLDD LETR PSKSDYPRPR LLFTKDQYEY NITKRCERVF EIDKGNKTGY PVDDQIKKNY EDILDSYDGI KDQEVAERFD TFTRGS KLK VGDLVYFHID GDNKIDSLIP VRISRKCASK TLGGKLDKAL HPCTGLSDGL CPGCHLFGTT DYKGRVKFGF AKYENGP EW LITRGNNPER SLTLGVLESP RPAFSIPDDE SEIPGRKFYL HHNGWRIIRQ KQLEIRETVQ PERNVTTEVM DKGNVFSF D VRFENLREWE LGLLLQSLDP GKNIAHKLGK GKPYGFGSVK IKIDSLHTFK INSNNDKIKR VPQSDIREYI NKGYQKLIE WSGNNSIQKG NVLPQWHVIP HIDKLYKLLW VPFLNDSKLE PDVRYPVLNE ESKGYIEGSD YTYKKLGDKD NLPYKTRVKG LTTPWSPWN PFQVIAEHEE QEVNVTGSRP SVTDKIERDG KMV |
-分子 #2: RNA (33-MER)
| 分子 | 名称: RNA (33-MER) / タイプ: rna / ID: 2 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種:   Candidatus Scalindua brodae (バクテリア) Candidatus Scalindua brodae (バクテリア) |
| 分子量 | 理論値: 10.404171 KDa |
| 配列 | 文字列: GACUUAAUGU CACGGUACCC AAUUUUCUGC CCC |
-分子 #3: ZINC ION
| 分子 | 名称: ZINC ION / タイプ: ligand / ID: 3 / コピー数: 4 / 式: ZN |
|---|---|
| 分子量 | 理論値: 65.409 Da |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.6 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / 装置: FEI VITROBOT MARK IV |
| 詳細 | No further treatment. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | JEOL CRYO ARM 300 |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 0.5 µm Bright-field microscopy / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 0.5 µm |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 40.0 e/Å2 |
 ムービー
ムービー コントローラー
コントローラー











 Z
Z Y
Y X
X