+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
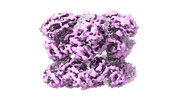
| タイトル | Cryo-EM structure of substrate engaged Drg1 hexamer | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |   Saccharomyces cerevisiae (パン酵母) / Saccharomyces cerevisiae (パン酵母) /   Escherichia coli (大腸菌) Escherichia coli (大腸菌) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.6 Å クライオ電子顕微鏡法 / 解像度: 3.6 Å | |||||||||
 データ登録者 データ登録者 | Ma CY / Wu DM / Chen Q / Gao N | |||||||||
| 資金援助 | 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2022 ジャーナル: Nat Commun / 年: 2022タイトル: Structural dynamics of AAA + ATPase Drg1 and mechanism of benzo-diazaborine inhibition. 著者: Chengying Ma / Damu Wu / Qian Chen / Ning Gao /  要旨: The type II AAA + ATPase Drg1 is a ribosome assembly factor, functioning to release Rlp24 from the pre-60S particle just exported from nucleus, and its activity in can be inhibited by a drug ...The type II AAA + ATPase Drg1 is a ribosome assembly factor, functioning to release Rlp24 from the pre-60S particle just exported from nucleus, and its activity in can be inhibited by a drug molecule diazaborine. However, molecular mechanisms of Drg1-mediated Rlp24 removal and diazaborine-mediated inhibition are not fully understood. Here, we report Drg1 structures in different nucleotide-binding and benzo-diazaborine treated states. Drg1 hexamers transits between two extreme conformations (planar or helical arrangement of protomers). By forming covalent adducts with ATP molecules in both ATPase domain, benzo-diazaborine locks Drg1 hexamers in a symmetric and non-productive conformation to inhibits both inter-protomer and inter-ring communication of Drg1 hexamers. We also obtained a substrate-engaged mutant Drg1 structure, in which conserved pore-loops form a spiral staircase to interact with the polypeptide through a sequence-independent manner. Structure-based mutagenesis data highlight the functional importance of the pore-loop, the D1-D2 linker and the inter-subunit signaling motif of Drg1, which share similar regulatory mechanisms with p97. Our results suggest that Drg1 may function as an unfoldase that threads a substrate protein within the pre-60S particle. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_32396.map.gz emd_32396.map.gz | 7.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-32396-v30.xml emd-32396-v30.xml emd-32396.xml emd-32396.xml | 10.9 KB 10.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_32396.png emd_32396.png | 65.5 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-32396 http://ftp.pdbj.org/pub/emdb/structures/EMD-32396 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-32396 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-32396 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_32396.map.gz / 形式: CCP4 / 大きさ: 52.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_32396.map.gz / 形式: CCP4 / 大きさ: 52.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.057 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Double mutant Drg1 in the presence of ATP with substrate engaged
| 全体 | 名称: Double mutant Drg1 in the presence of ATP with substrate engaged |
|---|---|
| 要素 |
|
-超分子 #1: Double mutant Drg1 in the presence of ATP with substrate engaged
| 超分子 | 名称: Double mutant Drg1 in the presence of ATP with substrate engaged タイプ: complex / ID: 1 / キメラ: Yes / 親要素: 0 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母) |
-分子 #1: AFG2 isoform 1
| 分子 | 名称: AFG2 isoform 1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 6 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母) |
| 分子量 | 理論値: 84.848742 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: MAPKSSSSGS KKKSSASSNS ADAKASKFKL PAEFITRPHP SKDHGKETCT AYIHPNVLSS LEINPGSFCT VGKIGENGIL VIARAGDEE VHPVNVITLS TTIRSVGNLI LGDRLELKKA QVQPPYATKV TVGSLQGYNI LECMEEKVIQ KLLDDSGVIM P GMIFQNLK ...文字列: MAPKSSSSGS KKKSSASSNS ADAKASKFKL PAEFITRPHP SKDHGKETCT AYIHPNVLSS LEINPGSFCT VGKIGENGIL VIARAGDEE VHPVNVITLS TTIRSVGNLI LGDRLELKKA QVQPPYATKV TVGSLQGYNI LECMEEKVIQ KLLDDSGVIM P GMIFQNLK TKAGDESIDV VITDASDDSL PDVSQLDLNM DDMYGGLDNL FYLSPPFIFR KGSTHITFSK ETQANRKYNL PE PLSYAAV GGLDKEIESL KSAIEIPLHQ PTLFSSFGVS PPRGILLHGP PGTGKTMLLR VVANTSNAHV LTINGPSIVS KYL GETEAA LRDIFNEARK YQPSIIFIDQ IDSIAPNRAN DDSGEVESRV VATLLTLMDG MGAAGKVVVI AATNRPNSVD PALR RPGRF DQEVEIGIPD VDARFDILTK QFSRMSSDRH VLDSEAIKYI ASKTHGYVGA DLTALCRESV MKTIQRGLGT DANID KFSL KVTLKDVESA MVDIRPSAMR EIFLEMPKVY WSDIGGQEEL KTKMKEMIQL PLEASETFAR LGISAPKGVL LYGPPG CSK TLTAKALATE SGINFLAVKG PEIFNKYVGE SERAIREIFR KARSAAPSII FFDQIDALSP DRDGSSTSAA NHVLTSL LN EIDGVEELKG VVIVAATNRP DEIDAALLRP GRLDRHIYVG PPDVNARLEI LKKCTKKFNT EESGVDLHEL ADRTEGYS G AEVVLLCQEA GLAAIMEDLD VAKVELRHFE KAFKGIARGI TPEMLSYYEE FALRSGSSS |
-分子 #2: substrate
| 分子 | 名称: substrate / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 分子量 | 理論値: 1.975426 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: (UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK)(UNK)(UNK) (UNK)(UNK)(UNK) (UNK)(UNK)(UNK)(UNK) |
-分子 #3: ADENOSINE-5'-TRIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-TRIPHOSPHATE / タイプ: ligand / ID: 3 / コピー数: 11 / 式: ATP |
|---|---|
| 分子量 | 理論値: 507.181 Da |
| Chemical component information |  ChemComp-ATP: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.5 µm Bright-field microscopy / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.5 µm |
| 撮影 | フィルム・検出器のモデル: GATAN K2 QUANTUM (4k x 4k) 平均電子線量: 8.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
|---|---|
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.6 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 152973 |
 ムービー
ムービー コントローラー
コントローラー