+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of glutamate dehydrogenase frozen at various temperature | |||||||||
 マップデータ マップデータ | The EM map of Glutamate dehydrogenase (GDH) embedded in vitreous ice frozen at -183 celsius degree using standard procedure. The resolution of EM map is 2.7 angstrom | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報glutamate dehydrogenase [NAD(P)+] activity / glutamate catabolic process / tricarboxylic acid metabolic process / glutamate dehydrogenase [NAD(P)+] /  glutamate dehydrogenase (NADP+) activity / glutamate dehydrogenase (NAD+) activity / glutamine metabolic process / glutamate dehydrogenase (NADP+) activity / glutamate dehydrogenase (NAD+) activity / glutamine metabolic process /  ミトコンドリア内膜 / GTP binding / ミトコンドリア内膜 / GTP binding /  小胞体 ...glutamate dehydrogenase [NAD(P)+] activity / glutamate catabolic process / tricarboxylic acid metabolic process / glutamate dehydrogenase [NAD(P)+] / 小胞体 ...glutamate dehydrogenase [NAD(P)+] activity / glutamate catabolic process / tricarboxylic acid metabolic process / glutamate dehydrogenase [NAD(P)+] /  glutamate dehydrogenase (NADP+) activity / glutamate dehydrogenase (NAD+) activity / glutamine metabolic process / glutamate dehydrogenase (NADP+) activity / glutamate dehydrogenase (NAD+) activity / glutamine metabolic process /  ミトコンドリア内膜 / GTP binding / ミトコンドリア内膜 / GTP binding /  小胞体 / 小胞体 /  ミトコンドリア / ミトコンドリア /  ATP binding / identical protein binding ATP binding / identical protein binding類似検索 - 分子機能 | |||||||||
| 生物種 |   Bos taurus (ウシ) / Bos taurus (ウシ) /   cattle (ウシ) cattle (ウシ) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 2.7 Å クライオ電子顕微鏡法 / 解像度: 2.7 Å | |||||||||
 データ登録者 データ登録者 | Shi H / Wu C / Zhang X | |||||||||
| 資金援助 |  中国, 1件 中国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Structure / 年: 2023 ジャーナル: Structure / 年: 2023タイトル: Addressing compressive deformation of proteins embedded in crystalline ice. 著者: Huigang Shi / Chunling Wu / Xinzheng Zhang /  要旨: For cryoelectron microscopy (cryo-EM), high cooling rates have been required for preparation of protein samples to vitrify the surrounding water and avoid formation of damaging crystalline ice. ...For cryoelectron microscopy (cryo-EM), high cooling rates have been required for preparation of protein samples to vitrify the surrounding water and avoid formation of damaging crystalline ice. Whether and how crystalline ice affects single-particle cryo-EM is still unclear. Here, single-particle cryo-EM was used to analyze three-dimensional structures of various proteins and viruses embedded in crystalline ice formed at various cooling rates. Low cooling rates led to shrinkage deformation and density distortions on samples having loose structures. Higher cooling rates reduced deformations. Deformation-free proteins in crystalline ice were obtained by modifying the freezing conditions, and reconstructions from these samples revealed a marked improvement over vitreous ice. This procedure also increased the efficiency of cryo-EM structure determinations and was essential for high-resolution reconstructions. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_28639.map.gz emd_28639.map.gz | 59.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-28639-v30.xml emd-28639-v30.xml emd-28639.xml emd-28639.xml | 45.2 KB 45.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_28639.png emd_28639.png | 36.4 KB | ||
| マスクデータ |  emd_28639_msk_1.map emd_28639_msk_1.map | 64 MB |  マスクマップ マスクマップ | |
| その他 |  emd_28639_additional_1.map.gz emd_28639_additional_1.map.gz emd_28639_additional_10.map.gz emd_28639_additional_10.map.gz emd_28639_additional_11.map.gz emd_28639_additional_11.map.gz emd_28639_additional_12.map.gz emd_28639_additional_12.map.gz emd_28639_additional_13.map.gz emd_28639_additional_13.map.gz emd_28639_additional_2.map.gz emd_28639_additional_2.map.gz emd_28639_additional_3.map.gz emd_28639_additional_3.map.gz emd_28639_additional_4.map.gz emd_28639_additional_4.map.gz emd_28639_additional_5.map.gz emd_28639_additional_5.map.gz emd_28639_additional_6.map.gz emd_28639_additional_6.map.gz emd_28639_additional_7.map.gz emd_28639_additional_7.map.gz emd_28639_additional_8.map.gz emd_28639_additional_8.map.gz emd_28639_additional_9.map.gz emd_28639_additional_9.map.gz emd_28639_half_map_1.map.gz emd_28639_half_map_1.map.gz emd_28639_half_map_2.map.gz emd_28639_half_map_2.map.gz | 59.9 MB 49.6 MB 49.6 MB 59.8 MB 49.5 MB 60 MB 49 MB 49 MB 59.9 MB 49.5 MB 49.6 MB 59 MB 59.8 MB 49 MB 49 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-28639 http://ftp.pdbj.org/pub/emdb/structures/EMD-28639 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-28639 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-28639 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8ew0MC  8bqnC  8ew2C  8f49C  8f7yC  8hhsC  8hi2C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_28639.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_28639.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | The EM map of Glutamate dehydrogenase (GDH) embedded in vitreous ice frozen at -183 celsius degree using standard procedure. The resolution of EM map is 2.7 angstrom | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.82 Å | ||||||||||||||||||||
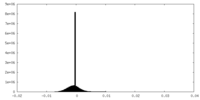
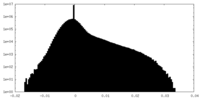
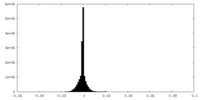
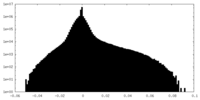
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
+マスク #1
+追加マップ: The EM map of Glutamate dehydrogenase (GDH) embedded...
+追加マップ: The half map of Glutamate dehydrogenase (GDH) embedded...
+追加マップ: The half map of Glutamate dehydrogenase (GDH) embedded...
+追加マップ: The EM map of Glutamate dehydrogenase (GDH) embedded...
+追加マップ: The half map of Glutamate dehydrogenase (GDH) embedded...
+追加マップ: The EM map of Glutamate dehydrogenase (GDH) embedded...
+追加マップ: The half map of Glutamate dehydrogenase (GDH) embedded...
+追加マップ: The half map of Glutamate dehydrogenase (GDH) embedded...
+追加マップ: The EM map of Glutamate dehydrogenase (GDH) embedded...
+追加マップ: The half map of Glutamate dehydrogenase (GDH) embedded...
+追加マップ: The half map of Glutamate dehydrogenase (GDH) embedded...
+追加マップ: The EM map of Glutamate dehydrogenase (GDH) embedded...
+追加マップ: The EM map of Glutamate dehydrogenase (GDH) embedded...
+ハーフマップ: The half map of Glutamate dehydrogenase (GDH) embedded...
+ハーフマップ: The EM map of Glutamate dehydrogenase (GDH) embedded...
- 試料の構成要素
試料の構成要素
-全体 : Glutamate Dehydrogenase
| 全体 | 名称: Glutamate Dehydrogenase グルタミン酸デヒドロゲナーゼ グルタミン酸デヒドロゲナーゼ |
|---|---|
| 要素 |
|
-超分子 #1: Glutamate Dehydrogenase
| 超分子 | 名称: Glutamate Dehydrogenase / タイプ: complex / ID: 1 / キメラ: Yes / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:   Bos taurus (ウシ) Bos taurus (ウシ) |
-分子 #1: Glutamate dehydrogenase 1, mitochondrial
| 分子 | 名称: Glutamate dehydrogenase 1, mitochondrial / タイプ: protein_or_peptide / ID: 1 / コピー数: 6 / 光学異性体: LEVO / EC番号: glutamate dehydrogenase [NAD(P)+] |
|---|---|
| 由来(天然) | 生物種:   cattle (ウシ) cattle (ウシ) |
| 分子量 | 理論値: 61.60891 KDa |
| 配列 | 文字列: MYRYLGEALL LSRAGPAALG SASADSAALL GWARGQPAAA PQPGLVPPAR RHYSEAAADR EDDPNFFKMV EGFFDRGASI VEDKLVEDL KTRETEEQKR NRVRSILRII KPCNHVLSLS FPIRRDDGSW EVIEGYRAQH SQHRTPCKGG IRYSTDVSVD E VKALASLM ...文字列: MYRYLGEALL LSRAGPAALG SASADSAALL GWARGQPAAA PQPGLVPPAR RHYSEAAADR EDDPNFFKMV EGFFDRGASI VEDKLVEDL KTRETEEQKR NRVRSILRII KPCNHVLSLS FPIRRDDGSW EVIEGYRAQH SQHRTPCKGG IRYSTDVSVD E VKALASLM TYKCAVVDVP FGGAKAGVKI NPKNYTDNEL EKITRRFTME LAKKGFIGPG VDVPAPDMST GEREMSWIAD TY ASTIGHY DINAHACVTG KPISQGGIHG RISATGRGVF HGIENFINEA SYMSILGMTP GFGDKTFVVQ GFGNVGLHSM RYL HRFGAK CITVGESDGS IWNPDGIDPK ELEDFKLQHG TILGFPKAKI YEGSILEVDC DILIPAASEK QLTKSNAPRV KAKI IAEGA NGPTTPEADK IFLERNIMVI PDLYLNAGGV TVSYFEWLKN LNHVSYGRLT FKYERDSNYH LLMSVQESLE RKFGK HGGT IPIVPTAEFQ DRISGASEKD IVHSGLAYTM ERSARQIMRT AMKYNLGLDL RTAAYVNAIE KVFRVYNEAG VTFT |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 6.8 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 3.5 µm / 最小 デフォーカス(公称値): 1.0 µm Bright-field microscopy / 最大 デフォーカス(公称値): 3.5 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K2 QUANTUM (4k x 4k) 検出モード: SUPER-RESOLUTION / 平均電子線量: 60.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD / ソフトウェア - 名称: RELION |
|---|---|
| 最終 3次元分類 | ソフトウェア - 名称: RELION |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD / ソフトウェア - 名称: RELION |
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 2.7 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 122399 |
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
|---|---|
| 得られたモデル |  PDB-8ew0: |
 ムービー
ムービー コントローラー
コントローラー










 Z
Z Y
Y X
X