+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | 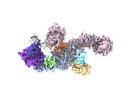 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
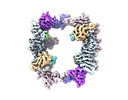
| タイトル | EM map of the human UBR5 HECT-type E3 ubiquitin ligase in a C2 symmetric dimeric form | |||||||||
 マップデータ マップデータ | Final composite map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | HECT /  E3 ligase (ユビキチンリガーゼ) / Dimer / E3 ligase (ユビキチンリガーゼ) / Dimer /  LIGASE (リガーゼ) LIGASE (リガーゼ) | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報heterochromatin boundary formation / HECT-type E3 ubiquitin transferase / DNA repair-dependent chromatin remodeling / ubiquitin-ubiquitin ligase activity / progesterone receptor signaling pathway / protein K48-linked ubiquitination /  ubiquitin binding / positive regulation of protein import into nucleus / protein polyubiquitination / ubiquitin binding / positive regulation of protein import into nucleus / protein polyubiquitination /  ubiquitin protein ligase activity ...heterochromatin boundary formation / HECT-type E3 ubiquitin transferase / DNA repair-dependent chromatin remodeling / ubiquitin-ubiquitin ligase activity / progesterone receptor signaling pathway / protein K48-linked ubiquitination / ubiquitin protein ligase activity ...heterochromatin boundary formation / HECT-type E3 ubiquitin transferase / DNA repair-dependent chromatin remodeling / ubiquitin-ubiquitin ligase activity / progesterone receptor signaling pathway / protein K48-linked ubiquitination /  ubiquitin binding / positive regulation of protein import into nucleus / protein polyubiquitination / ubiquitin binding / positive regulation of protein import into nucleus / protein polyubiquitination /  ubiquitin protein ligase activity / positive regulation of canonical Wnt signaling pathway / ubiquitin protein ligase activity / positive regulation of canonical Wnt signaling pathway /  DNA修復 / DNA damage response / positive regulation of gene expression / perinuclear region of cytoplasm / protein-containing complex / DNA修復 / DNA damage response / positive regulation of gene expression / perinuclear region of cytoplasm / protein-containing complex /  RNA binding / zinc ion binding / RNA binding / zinc ion binding /  核質 / 核質 /  生体膜 / 生体膜 /  細胞核 / 細胞核 /  細胞質基質 / 細胞質基質 /  細胞質 細胞質類似検索 - 分子機能 | |||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 2.66 Å クライオ電子顕微鏡法 / 解像度: 2.66 Å | |||||||||
 データ登録者 データ登録者 | Wang F / He Q / Lin G / Li H | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Structure / 年: 2023 ジャーナル: Structure / 年: 2023タイトル: Structure of the human UBR5 E3 ubiquitin ligase. 著者: Feng Wang / Qing He / Wenhu Zhan / Ziqi Yu / Efrat Finkin-Groner / Xiaojing Ma / Gang Lin / Huilin Li /  要旨: The human UBR5 is a single polypeptide chain homology to E6AP C terminus (HECT)-type E3 ubiquitin ligase essential for embryonic development in mammals. Dysregulated UBR5 functions like an ...The human UBR5 is a single polypeptide chain homology to E6AP C terminus (HECT)-type E3 ubiquitin ligase essential for embryonic development in mammals. Dysregulated UBR5 functions like an oncoprotein to promote cancer growth and metastasis. Here, we report that UBR5 assembles into a dimer and a tetramer. Our cryoelectron microscopy (cryo-EM) structures reveal that two crescent-shaped UBR5 monomers assemble head to tail to form the dimer, and two dimers bind face to face to form the cage-like tetramer with all four catalytic HECT domains facing the central cavity. Importantly, the N-terminal region of one subunit and the HECT of the other form an "intermolecular jaw" in the dimer. We show the jaw-lining residues are important for function, suggesting that the intermolecular jaw functions to recruit ubiquitin-loaded E2 to UBR5. Further work is needed to understand how oligomerization regulates UBR5 ligase activity. This work provides a framework for structure-based anticancer drug development and contributes to a growing appreciation of E3 ligase diversity. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_27822.map.gz emd_27822.map.gz | 217.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-27822-v30.xml emd-27822-v30.xml emd-27822.xml emd-27822.xml | 23.9 KB 23.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_27822.png emd_27822.png | 65.7 KB | ||
| Filedesc metadata |  emd-27822.cif.gz emd-27822.cif.gz | 7.6 KB | ||
| その他 |  emd_27822_additional_1.map.gz emd_27822_additional_1.map.gz emd_27822_additional_2.map.gz emd_27822_additional_2.map.gz emd_27822_additional_3.map.gz emd_27822_additional_3.map.gz emd_27822_additional_4.map.gz emd_27822_additional_4.map.gz | 216.1 MB 216.2 MB 216.7 MB 118.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-27822 http://ftp.pdbj.org/pub/emdb/structures/EMD-27822 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27822 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27822 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_27822.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_27822.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Final composite map | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.828 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: Focused on refinment map of UBR5-HECT
| ファイル | emd_27822_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Focused on refinment map of UBR5-HECT | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
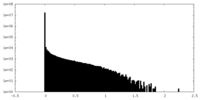
| 密度ヒストグラム |
-追加マップ: Focused on refinment map of UBR5-NTR
| ファイル | emd_27822_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Focused on refinment map of UBR5-NTR | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
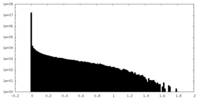
| 密度ヒストグラム |
-追加マップ: sharpen map of UBR5 in C2 symmetry
| ファイル | emd_27822_additional_3.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | sharpen map of UBR5 in C2 symmetry | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
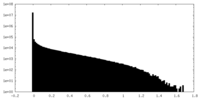
| 密度ヒストグラム |
-追加マップ: Raw EM map of UBR5 in C2 symmetry
| ファイル | emd_27822_additional_4.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Raw EM map of UBR5 in C2 symmetry | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
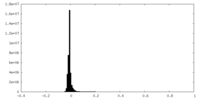
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Homodimer of human E3 ligase UBR5
| 全体 | 名称: Homodimer of human E3 ligase UBR5 |
|---|---|
| 要素 |
|
-超分子 #1: Homodimer of human E3 ligase UBR5
| 超分子 | 名称: Homodimer of human E3 ligase UBR5 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 / 詳細: UBR5 expressed in insect cell |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 610 KDa |
-分子 #1: E3 ubiquitin-protein ligase UBR5
| 分子 | 名称: E3 ubiquitin-protein ligase UBR5 / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO / EC番号: HECT-type E3 ubiquitin transferase |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 310.719719 KDa |
| 組換発現 | 生物種: Insect cell expression vector pTIE1 (その他) |
| 配列 | 文字列: DYKDDDDKMT SIHFVVHPLP GTEDQLNDRL REVSEKLNKY NLNSHPPLNV LEQATIKQCV VGPNHAAFLL EDGRVCRIGF SVQPDRLEL GKPDNNDGSK LNSNSGAGRT SRPGRTSDSP WFLSGSETLG RLAGNTLGSR WSSGVGGSGG GSSGRSSAGA R DSRRQTRV ...文字列: DYKDDDDKMT SIHFVVHPLP GTEDQLNDRL REVSEKLNKY NLNSHPPLNV LEQATIKQCV VGPNHAAFLL EDGRVCRIGF SVQPDRLEL GKPDNNDGSK LNSNSGAGRT SRPGRTSDSP WFLSGSETLG RLAGNTLGSR WSSGVGGSGG GSSGRSSAGA R DSRRQTRV IRTGRDRGSG LLGSQPQPVI PASVIPEELI SQAQVVLQGK SRSVIIRELQ RTNLDVNLAV NNLLSRDDED GD DGDDTAS ESYLPGEDLM SLLDADIHSA HPSVIIDADA MFSEDISYFG YPSFRRSSLS RLGSSRVLLL PLERDSELLR ERE SVLRLR ERRWLDGASF DNERGSTSKE GEPNLDKKNT PVQSPVSLGE DLQWWPDKDG TKFICIGALY SELLAVSSKG ELYQ WKWSE SEPYRNAQNP SLHHPRATFL GLTNEKIVLL SANSIRATVA TENNKVATWV DETLSSVASK LEHTAQTYSE LQGER IVSL HCCALYTCAQ LENSLYWWGV VPFSQRKKML EKARAKNKKP KSSAGISSMP NITVGTQVCL RNNPLYHAGA VAFSIS AGI PKVGVLMESV WNMNDSCRFQ LRSPESLKNM EKASKTTEAK PESKQEPVKT EMGPPPSPAS TCSDASSIAS SASMPYK RR RSTPAPKEEE KVNEEQWSLR EVVFVEDVKN VPVGKVLKVD GAYVAVKFPG TSSNTNCQNS SGPDADPSSL LQDCRLLR I DELQVVKTGG TPKVPDCFQR TPKKLCIPEK TEILAVNVDS KGVHAVLKTG NWVRYCIFDL ATGKAEQENN FPTSSIAFL GQNERNVAIF TAGQESPIIL RDGNGTIYPM AKDCMGGIRD PDWLDLPPIS SLGMGVHSLI NLPANSTIKK KAAVIIMAVE KQTLMQHIL RCDYEACRQY LMNLEQAVVL EQNLQMLQTF ISHRCDGNRN ILHACVSVCF PTSNKETKEE EEAERSERNT F AERLSAVE AIANAISVVS SNGPGNRAGS SSSRSLRLRE MMRRSLRAAG LGRHEAGASS SDHQDPVSPP IAPPSWVPDP PA MDPDGDI DFILAPAVGS LTTAATGTGQ GPSTSTIPGP STEPSVVESK DRKANAHFIL KLLCDSVVLQ PYLRELLSAK DAR GMTPFM SAVSGRAYPA AITILETAQK IAKAEISSSE KEEDVFMGMV CPSGTNPDDS PLYVLCCNDT CSFTWTGAEH INQD IFECR TCGLLESLCC CTECARVCHK GHDCKLKRTS PTAYCDCWEK CKCKTLIAGQ KSARLDLLYR LLTATNLVTL PNSRG EHLL LFLVQTVARQ TVEHCQYRPP RIREDRNRKT ASPEDSDMPD HDLEPPRFAQ LALERVLQDW NALKSMIMFG SQENKD PLS ASSRIGHLLP EEQVYLNQQS GTIRLDCFTH CLIVKCTADI LLLDTLLGTL VKELQNKYTP GRREEAIAVT MRFLRSV AR VFVILSVEMA SSKKKNNFIP QPIGKCKRVF QALLPYAVEE LCNVAESLIV PVRMGIARPT APFTLASTSI DAMQGSEE L FSVEPLPPRP SSDQSSSSSQ SQSSYIIRNP QQRRISQSQP VRGRDEEQDD IVSADVEEVE VVEGVAGEED HHDEQEEHG EENAEAEGQH DEHDEDGSDM ELDLLAAAET ESDSESNHSN QDNASGRRSV VTAATAGSEA GASSVPAFFS EDDSQSNDSS DSDSSSSQS DDIEQETFML DEPLERTTNS SHANGAAQAP RSMQWAVRNT QHQRAASTAP SSTSTPAASS AGLIYIDPSN L RRSGTIST SAAAAAAALE ASNASSYLTS ASSLARAYSI VIRQISDLMG LIPKYNHLVY SQIPAAVKLT YQDAVNLQNY VE EKLIPTW NWMVSIMDST EAQLRYGSAL ASAGDPGHPN HPLHASQNSA RRERMTAREE ASLRTLEGRR RATLLSARQG MMS ARGDFL NYALSLMRSH NDEHSDVLPV LDVCSLKHVA YVFQALIYWI KAMNQQTTLD TPQLERKRTR ELLELGIDNE DSEH ENDDD TNQSATLNDK DDDSLPAETG QNHPFFRRSD SMTFLGCIPP NPFEVPLAEA IPLADQPHLL QPNARKEDLF GRPSQ GLYS SSASSGKCLM EVTVDRNCLE VLPTKMSYAA NLKNVMNMQN RQKKEGEEQP VLPEETESSK PGPSAHDLAA QLKSSL LAE IGLTESEGPP LTSFRPQCSF MGMVISHDML LGRWRLSLEL FGRVFMEDVG AEPGSILTEL GGFEVKESKF RREMEKL RN QQSRDLSLEV DRDRDLLIQQ TMRQLNNHFG RRCATTPMAV HRVKVTFKDE PGEGSGVARS FYTAIAQAFL SNEKLPNL E CIQNANKGTH TSLMQRLRNR GERDRERERE REMRRSSGLR AGSRRDRDRD FRRQLSIDTR PFRPASEGNP SDDPEPLPA HRQALGERLY PRVQAMQPAF ASKITGMLLE LSPAQLLLLL ASEDSLRARV DEAMELIIAH GRENGADSIL DLGLVDSSEK VQQENRKRH GSSRSVVDMD LDDTDDGDDN APLFYQPGKR GFYTPRPGKN TEARLNCFRN IGRILGLCLL QNELCPITLN R HVIKVLLG RKVNWHDFAF FDPVMYESLR QLILASQSSD ADAVFSAMDL AFAIDLCKEE GGGQVELIPN GVNIPVTPQN VY EYVRKYA EHRMLVVAEQ PLHAMRKGLL DVLPKNSLED LTAEDFRLLV NGCGEVNVQM LISFTSFNDE SGENAEKLLQ FKR WFWSIV EKMSMTERQD LVYFWTSSPS LPASEEGFQP MPSITIRPPD DQHLPTANTC ISRLYVPLYS SKQILKQKLL LAIK TKNFG FV UniProtKB: E3 ubiquitin-protein ligase UBR5 |
-分子 #2: ZINC ION
| 分子 | 名称: ZINC ION / タイプ: ligand / ID: 2 / コピー数: 6 / 式: ZN |
|---|---|
| 分子量 | 理論値: 65.409 Da |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.7 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| ||||||||||||
| グリッド | モデル: Quantifoil R2/1 / 支持フィルム - 材質: GOLD / 支持フィルム - トポロジー: CONTINUOUS | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 279 K / 装置: FEI VITROBOT MARK IV / 詳細: blot 2S, blot forth 2. | ||||||||||||
| 詳細 | freshly purified UBR5 |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 105000 Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 105000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 温度 | 最低: 193.0 K / 最高: 193.0 K |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 65.0 e/Å2 詳細: Images were collected in movie-mode at 75 frames per second |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: NONE |
|---|---|
| 初期 角度割当 | タイプ: ANGULAR RECONSTITUTION |
| 最終 3次元分類 | クラス数: 3 / 平均メンバー数/クラス: 80 |
| 最終 角度割当 | タイプ: ANGULAR RECONSTITUTION |
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 2.66 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: cryoSPARC (ver. 3.2.0) / 使用した粒子像数: 844403 |
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT / 温度因子: 119 / 当てはまり具合の基準: Correlation coefficient |
|---|---|
| 得られたモデル |  PDB-8e0q: |
 ムービー
ムービー コントローラー
コントローラー






 Z
Z Y
Y X
X