+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | TBOA-bound GltPh RSMR mutant in OFS state | |||||||||
 マップデータ マップデータ | TBOA-bound RSMR mutant in OFS | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報amino acid:sodium symporter activity / L-aspartate transmembrane transport / L-aspartate transmembrane transporter activity / L-aspartate import across plasma membrane / chloride transmembrane transporter activity / protein homotrimerization / chloride transmembrane transport / identical protein binding /  metal ion binding / metal ion binding /  細胞膜 細胞膜類似検索 - 分子機能 | |||||||||
| 生物種 |    Pyrococcus horikoshii (古細菌) Pyrococcus horikoshii (古細菌) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 2.81 Å クライオ電子顕微鏡法 / 解像度: 2.81 Å | |||||||||
 データ登録者 データ登録者 | Huang Y / Boudker O | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: J.Am.Chem.Soc. / 年: 2023 ジャーナル: J.Am.Chem.Soc. / 年: 2023タイトル: Environmentally Ultrasensitive Fluorine Probe to Resolve Protein Conformational Ensembles by 19F NMR and Cryo-EM 著者: Huang Y / Reddy KD / Bracken C / Qiu B / Zhan W / Eliezer D / Boudker O | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
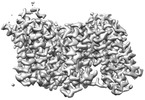
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_26489.map.gz emd_26489.map.gz | 108.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-26489-v30.xml emd-26489-v30.xml emd-26489.xml emd-26489.xml | 16.4 KB 16.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_26489_fsc.xml emd_26489_fsc.xml | 13.3 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_26489.png emd_26489.png | 105.4 KB | ||
| その他 |  emd_26489_half_map_1.map.gz emd_26489_half_map_1.map.gz emd_26489_half_map_2.map.gz emd_26489_half_map_2.map.gz | 200.2 MB 200.2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-26489 http://ftp.pdbj.org/pub/emdb/structures/EMD-26489 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26489 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26489 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_26489.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_26489.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | TBOA-bound RSMR mutant in OFS | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.852 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: Half Map 1
| ファイル | emd_26489_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half Map 1 | ||||||||||||
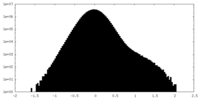
| 投影像・断面図 |
| ||||||||||||
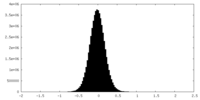
| 密度ヒストグラム |
-ハーフマップ: Half Map 2
| ファイル | emd_26489_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half Map 2 | ||||||||||||
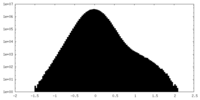
| 投影像・断面図 |
| ||||||||||||
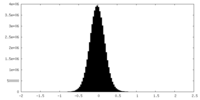
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : TBOA-bound GltPh RSMR mutant in OFS state
| 全体 | 名称: TBOA-bound GltPh RSMR mutant in OFS state |
|---|---|
| 要素 |
|
-超分子 #1: TBOA-bound GltPh RSMR mutant in OFS state
| 超分子 | 名称: TBOA-bound GltPh RSMR mutant in OFS state / タイプ: complex / ID: 1 / キメラ: Yes / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:    Pyrococcus horikoshii (古細菌) Pyrococcus horikoshii (古細菌) |
-分子 #1: Glutamate transporter homolog
| 分子 | 名称: Glutamate transporter homolog / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:    Pyrococcus horikoshii (古細菌) Pyrococcus horikoshii (古細菌)株: ATCC 700860 / DSM 12428 / JCM 9974 / NBRC 100139 / OT-3 |
| 分子量 | 理論値: 44.13323 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: MGLYRKYIEY PVLQKILIGL ILGAIVGLIL GHYGYAHAVH TYVKPFGDLF VRLLKMLVMP IVFASLVVGA ASISPARLGR VGVKIVVYY LLTSAFAVTL GIIMARLFNP GAGIHLAVGG QQFQPHQAPP LVHILLDIVP TNPFGALANG QVLPTIFFAI I LGIAITYL ...文字列: MGLYRKYIEY PVLQKILIGL ILGAIVGLIL GHYGYAHAVH TYVKPFGDLF VRLLKMLVMP IVFASLVVGA ASISPARLGR VGVKIVVYY LLTSAFAVTL GIIMARLFNP GAGIHLAVGG QQFQPHQAPP LVHILLDIVP TNPFGALANG QVLPTIFFAI I LGIAITYL MNSENEKVRK SAETLLDAIN GLAEAMYKIV NGVMQYAPIG VFALIAHVMA HQGVHVVGEL AKVTAAVYVG LT LQILLVY FVLLKIYGID PISFIKHAKD AMLTAFVTSS SSGTLPVTMR VAKEMGISEG IYSFTLPLGA TINMDGTALY QGV ATFFIA NALGSHLTVG QQLTIVLTAV LASIGTAGVP GAGAIMLAMV LHSVGLPLTD PNVAAAYA(EFC)I LGIDAILDRG RTMVNVTGD LTGTAIVAKT EGT |
-分子 #2: (3S)-3-(BENZYLOXY)-L-ASPARTIC ACID
| 分子 | 名称: (3S)-3-(BENZYLOXY)-L-ASPARTIC ACID / タイプ: ligand / ID: 2 / コピー数: 1 / 式: TB1 |
|---|---|
| 分子量 | 理論値: 239.225 Da |
| Chemical component information | 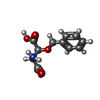 ChemComp-TB1: |
-分子 #3: SODIUM ION
| 分子 | 名称: SODIUM ION / タイプ: ligand / ID: 3 / コピー数: 2 |
|---|---|
| 分子量 | 理論値: 22.99 Da |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 4.5 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 298 K |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.3 µm Bright-field microscopy / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.3 µm |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 52.88 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー















 Z
Z Y
Y X
X