+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Mouse retromer (VPS26/VPS35 E615A/D616A/E617A mutant/VPS29) 3KE particle | |||||||||
 マップデータ マップデータ | 3KE mutant | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |   Mus musculus (ハツカネズミ) Mus musculus (ハツカネズミ) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 7.1 Å クライオ電子顕微鏡法 / 解像度: 7.1 Å | |||||||||
 データ登録者 データ登録者 | Kendall AK / Jackson LP | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: J Biol Chem / 年: 2022 ジャーナル: J Biol Chem / 年: 2022タイトル: Improved mammalian retromer cryo-EM structures reveal a new assembly interface. 著者: Amy K Kendall / Mintu Chandra / Boyang Xie / William Wan / Lauren P Jackson /  要旨: Retromer (VPS26/VPS35/VPS29 subunits) assembles with multiple sorting nexin proteins on membranes to mediate endosomal recycling of transmembrane protein cargoes. Retromer has been implicated in ...Retromer (VPS26/VPS35/VPS29 subunits) assembles with multiple sorting nexin proteins on membranes to mediate endosomal recycling of transmembrane protein cargoes. Retromer has been implicated in other cellular processes, including mitochondrial homeostasis, nutrient sensing, autophagy, and fission events. Mechanisms for mammalian retromer assembly remain undefined, and retromer engages multiple sorting nexin proteins to sort cargoes to different destinations. Published structures demonstrate mammalian retromer forms oligomers in vitro, but several structures were poorly resolved. We report here improved retromer oligomer structures using single-particle cryo-EM by combining data collected from tilted specimens with multiple advancements in data processing, including using a 3D starting model for enhanced automated particle picking in RELION. We used a retromer mutant (3KE retromer) that breaks VPS35-mediated interfaces to determine a structure of a new assembly interface formed by the VPS26A and VPS35 N-termini. The interface reveals how an N-terminal VPS26A arrestin saddle can link retromer chains by engaging a neighboring VPS35 N- terminus, on the opposite side from the well-characterized C-VPS26/N-VPS35 interaction observed within heterotrimers. The new interaction interface exhibits substantial buried surface area (∼7000 Å) and further suggests that metazoan retromer may serve as an adaptable scaffold. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_26341.map.gz emd_26341.map.gz | 9.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-26341-v30.xml emd-26341-v30.xml emd-26341.xml emd-26341.xml | 16 KB 16 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_26341.png emd_26341.png | 45.3 KB | ||
| その他 |  emd_26341_half_map_1.map.gz emd_26341_half_map_1.map.gz emd_26341_half_map_2.map.gz emd_26341_half_map_2.map.gz | 98.8 MB 98.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-26341 http://ftp.pdbj.org/pub/emdb/structures/EMD-26341 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26341 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26341 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_26341.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_26341.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | 3KE mutant | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.3622 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: 3KE mutant, half map 2
| ファイル | emd_26341_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | 3KE mutant, half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
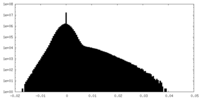
| 密度ヒストグラム |
-ハーフマップ: 3KE mutant, half map 1
| ファイル | emd_26341_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | 3KE mutant, half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
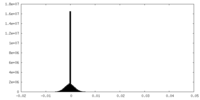
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Mouse retromer (VPS26/VPS35 E615A/D616A/E617A mutant/VPS29) 3KE p...
| 全体 | 名称: Mouse retromer (VPS26/VPS35 E615A/D616A/E617A mutant/VPS29) 3KE particle |
|---|---|
| 要素 |
|
-超分子 #1: Mouse retromer (VPS26/VPS35 E615A/D616A/E617A mutant/VPS29) 3KE p...
| 超分子 | 名称: Mouse retromer (VPS26/VPS35 E615A/D616A/E617A mutant/VPS29) 3KE particle タイプ: complex / キメラ: Yes / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:   Mus musculus (ハツカネズミ) Mus musculus (ハツカネズミ) |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
-分子 #1: Vacuolar protein sorting-associated protein 35 (E615A/D616A/E617A)
| 分子 | 名称: Vacuolar protein sorting-associated protein 35 (E615A/D616A/E617A) タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Mus musculus (ハツカネズミ) Mus musculus (ハツカネズミ) |
| 配列 | 文字列: MPTTQQSPQD EQEKLLDEA I QAVKVQSF QM KRCLDKN KLM DALKHA SNML GELRT SMLSP KSYY ELYMA ISDE LHYLEV YLT DEFAKGR KV ADLYELVQ Y AGNIIPRLY LLITVGVVYV K SFPQSRK D ILKDLVEM CR GVQHPLR GLF LRNYLL ...文字列: MPTTQQSPQD EQEKLLDEA I QAVKVQSF QM KRCLDKN KLM DALKHA SNML GELRT SMLSP KSYY ELYMA ISDE LHYLEV YLT DEFAKGR KV ADLYELVQ Y AGNIIPRLY LLITVGVVYV K SFPQSRK D ILKDLVEM CR GVQHPLR GLF LRNYLL QCTR NILPD EGEPT DEET TGDISD SM DFVLLNF AE MNKLWVRM Q HQGHSRDRE KRERERQELR ILVGTNLVR L SQLEGVNV ER YKQ IVL TGI LEQVVN CRDA LAQEY LMECI IQVF PDEFHL QTL NPFLRAC AE LHQNVNVK N III ALIDR LALFAHREDG PGIPAEIKL F DIFSQQVA TV IQSRQDM PSE DVVSLQ VSLI NLAMK CYPD RVDY VDKVLE TTV EIFNKLN LE HIATSSAV S KELTRLLKI PVDTYNNILT VLKLKH FH P LFEYFDYE SR KSMSCYV LSN VLDYNT EIVS QDQVD SIMNL VSTL IQDQPD QPV EDPD PE DF ADEQSLVG R FIHLLRSDD PDQQYLILNT ARKHFGAGG N QRIRFTLP PL VFAAYQL A F RYKENS QMDD KWEKK CQKIF SFAH QTISAL IKA ELAELPL RL FLQGALAA G EIGFENHE T VAYEFMSQA FSLYEDEISD SKAQLAAIT L IIGTFERM KC FSEENHE PLR TQCALA ASEL LEEPD QGRA VSTCA HLFWS GRNT DKNGEE LHG GKRVMEC LK KALKIANQ C MDPSLQVQL F IEILNRYI YFYEKENDA V TIQVLNQL IQ KIREDLP NLE SSEETE QINK HFHNT LEHLR SR R ESPESE GPI YEGLIL |
-分子 #2: Vacuolar protein sorting-associated protein 26A
| 分子 | 名称: Vacuolar protein sorting-associated protein 26A / タイプ: protein_or_peptide / ID: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Mus musculus (ハツカネズミ) Mus musculus (ハツカネズミ) |
| 配列 | 文字列: MSFLGGFFGP ICEIDVALN D GETRKMAE MK TEDGKVE KHY LFYDGE SVSG KVNLA FKQPG KRLE HQGIR IEFV GQIELF NDK SNTHEFV NL VKELALPG E LTQSRSYDF EFMQVEKPYE S YIGANVR L RYFLKVTI VR RLTDLVK EYD LIVHQL ...文字列: MSFLGGFFGP ICEIDVALN D GETRKMAE MK TEDGKVE KHY LFYDGE SVSG KVNLA FKQPG KRLE HQGIR IEFV GQIELF NDK SNTHEFV NL VKELALPG E LTQSRSYDF EFMQVEKPYE S YIGANVR L RYFLKVTI VR RLTDLVK EYD LIVHQL ATYP DVNNS IKMEV GIED CLHIEF EY NKSKYHL KD VIVGKIYF L LVRIKIQHM ELQLIKKEIT GIGPSTTTE T ETIAKYEI MD GAP VKG ESI PIRLFL AGYD PTPTM RDVNK KFSV RYFLNL VLV DEEDRRY FK QQEIILWR K APE KLRKQ RTNFHQRFES PDSQASAEQ P EM |
-分子 #3: vacuolar protein sorting-associated protein 29
| 分子 | 名称: vacuolar protein sorting-associated protein 29 / タイプ: protein_or_peptide / ID: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Mus musculus (ハツカネズミ) Mus musculus (ハツカネズミ) |
| 配列 | 文字列: MLVLVLGDLH IPHRCNSLP A KFKKLLVP GK IQHILCT GNL CTKESY DYLK TLAGD VHIVR GDFD ENLNY PEQK VVTVGQ FKI GLIHGHQ VI PWGDMASL A LLQRQFDVD ILISGHTHKF E AFEHENK F YINPGSAT GA YNALET |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 2.6 µm / 最小 デフォーカス(公称値): 0.8 µm Bright-field microscopy / 最大 デフォーカス(公称値): 2.6 µm / 最小 デフォーカス(公称値): 0.8 µm |
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 平均電子線量: 50.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
|---|---|
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 7.1 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 26641 |
 ムービー
ムービー コントローラー
コントローラー









 Z
Z Y
Y X
X