+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-20923 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | GltPh in complex with L-aspartate and sodium ions in intermediate outward-facing state | |||||||||
 マップデータ マップデータ | GltPh iOFS map | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報amino acid:sodium symporter activity / L-aspartate transmembrane transport / L-aspartate transmembrane transporter activity / L-aspartate import across plasma membrane / chloride transmembrane transporter activity / protein homotrimerization / chloride transmembrane transport / identical protein binding /  metal ion binding / metal ion binding /  細胞膜 細胞膜類似検索 - 分子機能 | |||||||||
| 生物種 |    Pyrococcus horikoshii (古細菌) Pyrococcus horikoshii (古細菌) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.62 Å クライオ電子顕微鏡法 / 解像度: 3.62 Å | |||||||||
 データ登録者 データ登録者 | Wang X / Boudker O | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Chem Biol / 年: 2020 ジャーナル: Nat Chem Biol / 年: 2020タイトル: Use of paramagnetic F NMR to monitor domain movement in a glutamate transporter homolog. 著者: Yun Huang / Xiaoyu Wang / Guohua Lv / Asghar M Razavi / Gerard H M Huysmans / Harel Weinstein / Clay Bracken / David Eliezer / Olga Boudker /   要旨: In proteins where conformational changes are functionally important, the number of accessible states and their dynamics are often difficult to establish. Here we describe a novel F-NMR spectroscopy ...In proteins where conformational changes are functionally important, the number of accessible states and their dynamics are often difficult to establish. Here we describe a novel F-NMR spectroscopy approach to probe dynamics of large membrane proteins. We labeled a glutamate transporter homolog with a F probe via cysteine chemistry and with a Ni ion via chelation by a di-histidine motif. We used distance-dependent enhancement of the longitudinal relaxation of F nuclei by the paramagnetic metal to assign the observed resonances. We identified one inward- and two outward-facing states of the transporter, in which the substrate-binding site is near the extracellular and intracellular solutions, respectively. We then resolved the structure of the unanticipated second outward-facing state by cryo-EM. Finally, we showed that the rates of the conformational exchange are accessible from measurements of the metal-enhanced longitudinal relaxation of F nuclei. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_20923.map.gz emd_20923.map.gz | 3.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-20923-v30.xml emd-20923-v30.xml emd-20923.xml emd-20923.xml | 11.2 KB 11.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_20923.png emd_20923.png | 24.8 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-20923 http://ftp.pdbj.org/pub/emdb/structures/EMD-20923 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20923 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20923 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_20923.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_20923.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | GltPh iOFS map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.06 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Complex of GltPh with L-aspartate and sodium ions in MSP1E3 nanodisc
| 全体 | 名称: Complex of GltPh with L-aspartate and sodium ions in MSP1E3 nanodisc |
|---|---|
| 要素 |
|
-超分子 #1: Complex of GltPh with L-aspartate and sodium ions in MSP1E3 nanodisc
| 超分子 | 名称: Complex of GltPh with L-aspartate and sodium ions in MSP1E3 nanodisc タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:    Pyrococcus horikoshii (古細菌) Pyrococcus horikoshii (古細菌) |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 分子量 | 理論値: 134 KDa |
-分子 #1: Glutamate transporter homolog
| 分子 | 名称: Glutamate transporter homolog / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:    Pyrococcus horikoshii (古細菌) Pyrococcus horikoshii (古細菌) |
| 分子量 | 理論値: 44.669957 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: (FME)GLYRKYIEY PVLQKILIGL ILGAIVGLIL GHYGYAHAVH TYVKPFGDLF VRLLKMLVMP IVFASLVVGA ASISPA RLG RVGVKIVVYY LLTSAFAVTL GIIMARLFNP GAGIHLAVGG QQFQPHQAPP LVHILLDIVP TNPFGALANG QVLPTIF FA ...文字列: (FME)GLYRKYIEY PVLQKILIGL ILGAIVGLIL GHYGYAHAVH TYVKPFGDLF VRLLKMLVMP IVFASLVVGA ASISPA RLG RVGVKIVVYY LLTSAFAVTL GIIMARLFNP GAGIHLAVGG QQFQPHQAPP LVHILLDIVP TNPFGALANG QVLPTIF FA IILGIAITYL MNSENEKVRK SAETLLDAIN GLAEAMYKIV NGVMQYAPIG VFALIAYVMA EQGVHVVGEL AKVTAAVY V GLTLQILLVY FVLLKIYGID PISFIKHAKD AMLTAFVTRS SSGTLPVTMR VAKEMGISEG IYSFTLPLGA TINMDGTAL YQGVCTFFIA NALGSHLTVG QQLTIVLTAV LASIGTAGVP GAGAIMLAMV LHSVGLPLTD PNVAAAYAMI LGIDAILDMG RTMVNVTGD LTGTAIVAKT EGTLVPR |
-分子 #2: ASPARTIC ACID
| 分子 | 名称: ASPARTIC ACID / タイプ: ligand / ID: 2 / コピー数: 1 / 式: ASP |
|---|---|
| 分子量 | 理論値: 133.103 Da |
| Chemical component information |  ChemComp-ASP: |
-分子 #3: [(2~{R})-1-[2-azanylethoxy(oxidanyl)phosphoryl]oxy-3-hexadecanoyl...
| 分子 | 名称: [(2~{R})-1-[2-azanylethoxy(oxidanyl)phosphoryl]oxy-3-hexadecanoyloxy-propan-2-yl] (~{Z})-octadec-9-enoate タイプ: ligand / ID: 3 / コピー数: 1 / 式: 6OU |
|---|---|
| 分子量 | 理論値: 717.996 Da |
| Chemical component information | 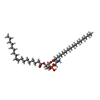 ChemComp-6OU: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| 糖包埋 | 材質: ice |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy |
| 撮影 | #0 - Image recording ID: 1 #0 - フィルム・検出器のモデル: GATAN K3 (6k x 4k) #0 - 平均電子線量: 50.1615 e/Å2 / #1 - Image recording ID: 2 #1 - フィルム・検出器のモデル: GATAN K3 (6k x 4k) #1 - 平均電子線量: 50.1615 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期 角度割当 | タイプ: ANGULAR RECONSTITUTION |
|---|---|
| 最終 角度割当 | タイプ: ANGULAR RECONSTITUTION |
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.62 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 120280 |
| Image recording ID | 1 |
 ムービー
ムービー コントローラー
コントローラー


















