[日本語] English
 万見
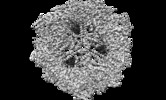
万見- EMDB-20657: Electron cryomicroscopy Structure of C. albicans FAS in the Apo state -
+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-20657 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Electron cryomicroscopy Structure of C. albicans FAS in the Apo state | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報mitotic nuclear membrane biogenesis / palmitic acid biosynthetic process / : / fatty-acyl-CoA synthase system / : /  fatty-acyl-CoA synthase activity / fatty-acyl-CoA synthase activity /  fatty acid synthase complex / [acyl-carrier-protein] S-acetyltransferase / fatty acid synthase complex / [acyl-carrier-protein] S-acetyltransferase /  palmitoyltransferase activity / [acyl-carrier-protein] S-acetyltransferase activity ...mitotic nuclear membrane biogenesis / palmitic acid biosynthetic process / : / fatty-acyl-CoA synthase system / : / palmitoyltransferase activity / [acyl-carrier-protein] S-acetyltransferase activity ...mitotic nuclear membrane biogenesis / palmitic acid biosynthetic process / : / fatty-acyl-CoA synthase system / : /  fatty-acyl-CoA synthase activity / fatty-acyl-CoA synthase activity /  fatty acid synthase complex / [acyl-carrier-protein] S-acetyltransferase / fatty acid synthase complex / [acyl-carrier-protein] S-acetyltransferase /  palmitoyltransferase activity / [acyl-carrier-protein] S-acetyltransferase activity / oleoyl-[acyl-carrier-protein] hydrolase / (3R)-3-hydroxypalmitoyl-[acyl-carrier-protein] dehydratase activity / (3R)-3-hydroxymyristoyl-[acyl-carrier-protein] dehydratase activity / holo-[acyl-carrier-protein] synthase activity / [acyl-carrier-protein] S-malonyltransferase / [acyl-carrier-protein] S-malonyltransferase activity / 3-hydroxyacyl-[acyl-carrier-protein] dehydratase / beta-ketoacyl-[acyl-carrier-protein] synthase I / 3-oxoacyl-[acyl-carrier-protein] reductase / 3-oxoacyl-[acyl-carrier-protein] reductase (NADPH) activity / palmitoyltransferase activity / [acyl-carrier-protein] S-acetyltransferase activity / oleoyl-[acyl-carrier-protein] hydrolase / (3R)-3-hydroxypalmitoyl-[acyl-carrier-protein] dehydratase activity / (3R)-3-hydroxymyristoyl-[acyl-carrier-protein] dehydratase activity / holo-[acyl-carrier-protein] synthase activity / [acyl-carrier-protein] S-malonyltransferase / [acyl-carrier-protein] S-malonyltransferase activity / 3-hydroxyacyl-[acyl-carrier-protein] dehydratase / beta-ketoacyl-[acyl-carrier-protein] synthase I / 3-oxoacyl-[acyl-carrier-protein] reductase / 3-oxoacyl-[acyl-carrier-protein] reductase (NADPH) activity /  fatty acid synthase activity / enoyl-[acyl-carrier-protein] reductase (NADH) / long-chain fatty acid biosynthetic process / enoyl-[acyl-carrier-protein] reductase (NADH) activity / 3-oxoacyl-[acyl-carrier-protein] synthase activity / fatty acid synthase activity / enoyl-[acyl-carrier-protein] reductase (NADH) / long-chain fatty acid biosynthetic process / enoyl-[acyl-carrier-protein] reductase (NADH) activity / 3-oxoacyl-[acyl-carrier-protein] synthase activity /  lipid droplet / magnesium ion binding / lipid droplet / magnesium ion binding /  細胞質基質 細胞質基質類似検索 - 分子機能 | |||||||||
| 生物種 |   Candida albicans (酵母) / Candida albicans (酵母) /   Yeast (酵母) Yeast (酵母) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 2.8 Å クライオ電子顕微鏡法 / 解像度: 2.8 Å | |||||||||
 データ登録者 データ登録者 | Lou JW / Mazhab-Jafari MT | |||||||||
 引用 引用 |  ジャーナル: Sci Rep / 年: 2019 ジャーナル: Sci Rep / 年: 2019タイトル: Electron cryomicroscopy observation of acyl carrier protein translocation in type I fungal fatty acid synthase. 著者: Jennifer W Lou / Kali R Iyer / S M Naimul Hasan / Leah E Cowen / Mohammad T Mazhab-Jafari /  要旨: During fatty acid biosynthesis, acyl carrier proteins (ACPs) from type I fungal fatty acid synthase (FAS) shuttle substrates and intermediates within a reaction chamber that hosts multiple spatially- ...During fatty acid biosynthesis, acyl carrier proteins (ACPs) from type I fungal fatty acid synthase (FAS) shuttle substrates and intermediates within a reaction chamber that hosts multiple spatially-fixed catalytic centers. A major challenge in understanding the mechanism of ACP-mediated substrate shuttling is experimental observation of its transient interaction landscape within the reaction chamber. Here, we have shown that ACP spatial distribution is sensitive to the presence of substrates in a catalytically inhibited state, which enables high-resolution investigation of the ACP-dependent conformational transitions within the enoyl reductase (ER) reaction site. In two fungal FASs with distinct ACP localization, the shuttling domain is targeted to the ketoacyl-synthase (KS) domain and away from other catalytic centers, such as acetyl-transferase (AT) and ER domains by steric blockage of the KS active site followed by addition of substrates. These studies strongly suggest that acylation of phosphopantetheine arm of ACP may be an integral part of the substrate shuttling mechanism in type I fungal FAS. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_20657.map.gz emd_20657.map.gz | 83.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-20657-v30.xml emd-20657-v30.xml emd-20657.xml emd-20657.xml | 15.6 KB 15.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_20657.png emd_20657.png | 290.9 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-20657 http://ftp.pdbj.org/pub/emdb/structures/EMD-20657 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20657 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20657 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_20657.map.gz / 形式: CCP4 / 大きさ: 166.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_20657.map.gz / 形式: CCP4 / 大きさ: 166.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.06 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Fatty acid synthase
| 全体 | 名称: Fatty acid synthase 脂肪酸合成酵素 脂肪酸合成酵素 |
|---|---|
| 要素 |
|
-超分子 #1: Fatty acid synthase
| 超分子 | 名称: Fatty acid synthase / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #2 |
|---|---|
| 由来(天然) | 生物種:   Candida albicans (酵母) Candida albicans (酵母) |
| 分子量 | 理論値: 2.6 MDa |
-分子 #1: Fatty acid synthase subunit alpha
| 分子 | 名称: Fatty acid synthase subunit alpha / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO / EC番号: fatty-acyl-CoA synthase system |
|---|---|
| 由来(天然) | 生物種:   Yeast (酵母) Yeast (酵母) |
| 分子量 | 理論値: 207.703453 KDa |
| 配列 | 文字列: MKPEIEQELS HTLLTELLAY QFASPVRWIE TQDVFLKQHN TERIIEIGPS PTLAGMANRT IKAKYESYDA ALSLQRQVLC YSKDAKEIY YKPDPADLAP KETPKQEEST PSAPAAATPT PAAAAAPTPA PAPASAGPVE SIPDEPVKAN LLIHVLVAQK L KKPLDAVP ...文字列: MKPEIEQELS HTLLTELLAY QFASPVRWIE TQDVFLKQHN TERIIEIGPS PTLAGMANRT IKAKYESYDA ALSLQRQVLC YSKDAKEIY YKPDPADLAP KETPKQEEST PSAPAAATPT PAAAAAPTPA PAPASAGPVE SIPDEPVKAN LLIHVLVAQK L KKPLDAVP MTKAIKDLVN GKSTVQNEIL GDLGKEFGST PEKPEDTPLE ELAEQFQDSF SGQLGKTSTS LIGRLMSSKM PG GFSITTA RKYLESRFGL GAGRQDSVLL MALTNEPANR LGSEADAKTF FDGIAQKYAS SAGISLSSGA GSGAGAANSG GAV VDSAAL DALTAENKKL AKQQLEVLAR YLQVDLNKGS AKSFIKEKEA SAVLQKELDL WEAEHGEFYA KGIQPTFSAL KSRT YDSYW NWARQDVLSM YFDIIFGKLT SVDRETINQC IQIMNRANPT LIKFMQYHID HCPEYKGETY KLAKRLGQQL IDNCK QVLT EDPVYKDVSR ITGPKTKVSA KGNIEYEETQ KDSVRKFEQY VYEMAQGGAM TKVSQPTIQE DLARVYKAIS KQASKD SKL ELQRVYEDLL KVVESSKEIE TEQLTKDILQ AATVPTTPTE EVDDPCTPSS DDEIASLPDK TSIIQPVSST IPSQTIP FL HIQKKTKDGW EYNKKLSSLY LDGLESAAIN GLTFKDKYVL VTGAGAGSIG AEILQGLISG GAKVIVTTSR FSKKVTEY Y QNMYARYGAA GSTLIVVPFN QGSKQDVDAL VQYIYDEPKK GGLGWDLDAI IPFAAIPENG NGLDNIDSKS EFAHRIMLT NLLRLLGAVK SKKTTDTRPA QCILPLSPNH GTFGFDGLYS ESKISLETLF NRWYSEDWGS KLTVCGAVIG WTRGTGLMSA NNIIAEGIE KLGVRTFSQK EMAFNILGLL TPEIVQLCQE EPVMADLNGG LQFIDNLKDF TSKLRTDLLE TADIRRAVSI E SAIEQKVV NGDNVDANYS KVMVEPRANM KFDFPTLKSY DEIKQIAPEL EGMLDLENVV VVTGFAEVGP WGNSRTRWEM EA YGEFSLE GAIEMAWIMG FIKYHNGNLK GKPYSGWVDA KTQTPIDEKD IKSKYEEEIL EHSGIRLIEP ELFNGYDPKK KQM IQEVVV QHDLEPFECS KETAEQYKHE HGEKCEIFEI EESGEYTVRI LKGATLYVPK ALRFDRLVAG QIPTGWDART YGIP EDTIS QVDPITLYVL VATVEALLSA GITDPYEFYK YVHVSEVGNC SGSGMGGVSA LRGMFKDRYA DKPVQNDILQ ESFIN TMSA WVNMLLLSSS GPIKTPVGAC ATAVESVDIG IETILSGKAK VVLVGGYDDF QEEGSYEFAN MNATSNSIEE FKHGRT PKE MSRPTTTTRN GFMEAQGSGI QVIMTADLAL KMGVPIHAVL AMTATATDKI GRSVPAPGKG ILTTAREHHG NLKYPSP LL NIEYRKRQLN KRLEQIKSWE ETELSYLQEE AELAKEEFGD EFSMHEFLKE RTEEVYRESK RQVSDAKKQW GNSFYKSD P RIAPLRGALA AFNLTIDDIG VASFHGTSTV ANDKNESATI NNMMKHLGRS EGNPVFGVFQ KYLTGHPKGA AGAWMLNGA IQILESGLVP GNRNADNVDK LLEQYEYVLY PSRSIQTDGI KAVSVTSFGF GQKGAQAVVV HPDYLFAVLD RSTYEEYATK VSARNKKTY RYMHNAITRN TMFVAKDKAP YSDELEQPVY LDPLARVEEN KKKLVFSDKT IQSSQSYVGE VAQKTAKALS T LNKSSKGV GVDVELLSAI NIDNETFIER NFTGNEVEYC LNTAHPQASF TGTWSAKEAV FKALGVESKG AGASLIDIEI TR DVNGAPK VILHGEAKKA AAKAGVKNVN ISISHDDFQA TAVALSEF |
-分子 #2: Fatty acid synthase subunit beta
| 分子 | 名称: Fatty acid synthase subunit beta / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO / EC番号: fatty-acyl-CoA synthase system |
|---|---|
| 由来(天然) | 生物種:   Yeast (酵母) Yeast (酵母) |
| 分子量 | 理論値: 228.177609 KDa |
| 配列 | 文字列: MSTHRPFQLT HGSIEHTLLV PNDLFFNYSQ LKDEFIKTLP EPTEGFAGDD EPSSPAELYG KFIGFISNAQ FPQIVELSLK DFESRFLDN NNDNIHSFAV KLLDDETYPT TIAKVKENIV KNYYKAVKSI NKVESNLLYH CKHDAKLVAI FGGQGNTDDY F EELRELYT ...文字列: MSTHRPFQLT HGSIEHTLLV PNDLFFNYSQ LKDEFIKTLP EPTEGFAGDD EPSSPAELYG KFIGFISNAQ FPQIVELSLK DFESRFLDN NNDNIHSFAV KLLDDETYPT TIAKVKENIV KNYYKAVKSI NKVESNLLYH CKHDAKLVAI FGGQGNTDDY F EELRELYT LYQGLIEDLL VSIAEKLNQL HPSFDKIYTQ GLNILSWLKH PETTPDQDYL LSVPVSCPVI CVIQLCHYTI TC KVLGLTP GEFRNSLKWS TGHSQGLVTA VTIAASDSWD SFLKNSLTAV SLLLFIGSRC LSTYPRTSLP PTMLQDSLDN GEG RPSPML SVRDLSIKQV EKFIEQTNSH LPREKHIAIS LINGARNLVL SGPPESLYGF NLNLRNQKAP MGLDQSRVPF SERK LKCSN RFLPIFAPFH SHLLADATEL ILDDVKEHGL SFEGLKIPVY DTFDGSDFQA LKEPIIDRVV KLITELPVHW EEATN HKAT HILDFGPGGV SGLGVLTHRN KEGTGARIIL AGTLDSNPID DEYGFKHEIF QTSADKAIKW APDWLKELRP TLVKNS EGK IYVKTKFSQL LGRAPLMVAG MTPTTVNTDI VSASLNAGYH IELAGGGYFS PVMMTRAIDD IVSRIKPGYG LGINLIY VN PFMLQWGIPL IKDLREKGYP IQSLTIGAGV PSIEVATEYI EDLGLTHLGL KPGSVDAISQ VIAIAKAHPT FPIVLQWT G GRGGGHHSFE DFHQPIIQMY SKIRRCSNIV LVAGSGFGSD EDTYPYLSGY WSEKFNYPPM PFDGVLFGSR VMTSKESHT SLAAKKLIVE CKGVPDQQWE QTYKKPTGGI ITVRSEMGEP IHKIATRGVM FWKELDDTIF NLPKNKLLDA LNKKRDHIIK KLNNDFQKP WFGKNANGVC DLQEMTYKEV ANRLVELMYV KKSHRWIDVS LRNMYGDFLR RVEERFTSSA GTVSLLQNFN Q LNEPEQFT ADFFEKFPQA GKQLISEEDC DYFLMLAARP GQKPVPFVPV LDERFEFFFK KDSLWQSEDL ESVVDEDVQR TC ILHGPVA SQYTSKVDEP IGDILNSIHE GHIARLIKEE YAGDESKIPV VEYFGGKKPA SVSATSVNII DGNQVVYEID SEL PNKQEW LDLLAGTELN WLQAFISTDR IVQGSKHVSN PLHDILTPAK HSKVTIDKKT KKLTAFENIK GDLLPVVEIE LVKP NTIQL SLIEHRTADT NPVALPFLYK YNPADGFAPI LEIMEDRNER IKEFYWKLWF GSSVPYSNDI NVEKAILGDE ITISS QTIS EFTHAIGNKC DAFVDRPGKA TLAPMDFAIV IGWKAIIKAI FPKSVDGDLL KLVHLSNGYK MITGAAPLKK GDVVST KAE IKAVLNQPSG KLVEVVGTIY REGKPVMEVT SQFLYRGEYN DYCNTFQKVT ETPVQVAFKS AKDLAVLRSK EWFHLEK DV QFDVLTFRCE STYKFKSANV YSSIKTTGQV LLELPTKEVI QVGSVDYEAG TSYGNPVTDY LSRNGKTIEE SVIFENAI P LSSGEELTSK APGTNEPYAI VSGDYNPIHV SRVFAAYAKL PGTITHGMYS SASIRALVEE WAANNVAARV RAFKCDFVG MVLPNDTLQT TMEHVGMING RKIIKVETRN VETELPVLIG EAEIEQPTTT YVFTGQGSQE QGMGMELYNS SEVAREVWDK ADRHFVNNY GFSILDIVQN NPNELTIHFG GAKGRAIRDN YIGMMFETIG EDGALKSEKI FKDIDETTTS YTFVSPTGLL S ATQFTQPA LTLMEKAAYE DIKSKGLIPS DIMFAGHSLG EYSALSSLAN VMPIESLVDV VFYRGMTMQV AVPRDELGRS NY GMVAVNP SRVSATFDDS ALRFVVDEVA NKTKWLLEIV NYNVENQQYV AAGDLRALDT LTNVLNVLKI NKIDIVKLQE QMS IEKVKE HLYEIVDEVA AKSLAKPQPI DLERGFAVIP LKGISVPFHS SYLMSGVKPF QRFLCKKIPK SSVKPQDLIG KYIP NLTAK PFELTKEYFQ SVYDLTKSEK IKSILDNWEQ YE |
-分子 #3: 4'-PHOSPHOPANTETHEINE
| 分子 | 名称: 4'-PHOSPHOPANTETHEINE / タイプ: ligand / ID: 3 / コピー数: 1 / 式: PNS |
|---|---|
| 分子量 | 理論値: 358.348 Da |
| Chemical component information |  ChemComp-PNS: |
-分子 #4: FLAVIN MONONUCLEOTIDE
| 分子 | 名称: FLAVIN MONONUCLEOTIDE / タイプ: ligand / ID: 4 / コピー数: 1 / 式: FMN |
|---|---|
| 分子量 | 理論値: 456.344 Da |
| Chemical component information |  ChemComp-FMN: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.15 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy |
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 平均電子線量: 43.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| CTF補正 | ソフトウェア - 名称: CTFFIND (ver. 4) / 詳細: CTFFIND4 within cryoSPARC2 |
|---|---|
| 初期モデル | モデルのタイプ: PDB ENTRY PDBモデル - PDB ID: |
| 初期 角度割当 | タイプ: OTHER / ソフトウェア - 名称: cryoSPARC (ver. 2) / 詳細: cryoSPARC2 |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD / ソフトウェア - 名称: cryoSPARC (ver. 2) |
| 最終 再構成 | 使用したクラス数: 1 想定した対称性 - 点群: D3 (2回x3回 2面回転対称  ) )解像度のタイプ: BY AUTHOR / 解像度: 2.8 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: cryoSPARC (ver. 2) / 使用した粒子像数: 92958 |
 ムービー
ムービー コントローラー
コントローラー