+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of C-terminally truncated Apoptosis signal-regulating kinase 1 (ASK1) | |||||||||
 マップデータ マップデータ | main map, sharpened in phenix.autosharpen | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  ASK1 (MAP3K5) / ASK1 (MAP3K5) /  MAP3K (MAPキナーゼキナーゼキナーゼ) / MAPK signaling / MAP3K (MAPキナーゼキナーゼキナーゼ) / MAPK signaling /  thioredoxin (チオレドキシン) / thioredoxin (チオレドキシン) /  APOPTOSIS (アポトーシス) APOPTOSIS (アポトーシス) | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報cellular response to reactive nitrogen species / neuron intrinsic apoptotic signaling pathway in response to oxidative stress / IRE1-TRAF2-ASK1 complex /  protein kinase complex / protein kinase complex /  MAPキナーゼキナーゼキナーゼ / programmed necrotic cell death / JUN kinase kinase kinase activity / endothelial cell apoptotic process / positive regulation of p38MAPK cascade / intrinsic apoptotic signaling pathway in response to oxidative stress ...cellular response to reactive nitrogen species / neuron intrinsic apoptotic signaling pathway in response to oxidative stress / IRE1-TRAF2-ASK1 complex / MAPキナーゼキナーゼキナーゼ / programmed necrotic cell death / JUN kinase kinase kinase activity / endothelial cell apoptotic process / positive regulation of p38MAPK cascade / intrinsic apoptotic signaling pathway in response to oxidative stress ...cellular response to reactive nitrogen species / neuron intrinsic apoptotic signaling pathway in response to oxidative stress / IRE1-TRAF2-ASK1 complex /  protein kinase complex / protein kinase complex /  MAPキナーゼキナーゼキナーゼ / programmed necrotic cell death / JUN kinase kinase kinase activity / endothelial cell apoptotic process / positive regulation of p38MAPK cascade / intrinsic apoptotic signaling pathway in response to oxidative stress / positive regulation of cardiac muscle cell apoptotic process / p38MAPK cascade / positive regulation of cysteine-type endopeptidase activity involved in apoptotic process / MAPキナーゼキナーゼキナーゼ / programmed necrotic cell death / JUN kinase kinase kinase activity / endothelial cell apoptotic process / positive regulation of p38MAPK cascade / intrinsic apoptotic signaling pathway in response to oxidative stress / positive regulation of cardiac muscle cell apoptotic process / p38MAPK cascade / positive regulation of cysteine-type endopeptidase activity involved in apoptotic process /  MAP kinase kinase kinase activity / intrinsic apoptotic signaling pathway in response to endoplasmic reticulum stress / positive regulation of protein kinase activity / positive regulation of myoblast differentiation / stress-activated MAPK cascade / positive regulation of JUN kinase activity / JNK cascade / positive regulation of vascular associated smooth muscle cell proliferation / cellular response to amino acid starvation / response to endoplasmic reticulum stress / response to ischemia / apoptotic signaling pathway / positive regulation of JNK cascade / cellular response to hydrogen peroxide / MAP kinase kinase kinase activity / intrinsic apoptotic signaling pathway in response to endoplasmic reticulum stress / positive regulation of protein kinase activity / positive regulation of myoblast differentiation / stress-activated MAPK cascade / positive regulation of JUN kinase activity / JNK cascade / positive regulation of vascular associated smooth muscle cell proliferation / cellular response to amino acid starvation / response to endoplasmic reticulum stress / response to ischemia / apoptotic signaling pathway / positive regulation of JNK cascade / cellular response to hydrogen peroxide /  分裂促進因子活性化タンパク質キナーゼ / 分裂促進因子活性化タンパク質キナーゼ /  細胞老化 / cellular response to tumor necrosis factor / 細胞老化 / cellular response to tumor necrosis factor /  protein phosphatase binding / Oxidative Stress Induced Senescence / neuron apoptotic process / protein phosphatase binding / Oxidative Stress Induced Senescence / neuron apoptotic process /  protein kinase activity / positive regulation of apoptotic process / protein domain specific binding / external side of plasma membrane / protein kinase activity / positive regulation of apoptotic process / protein domain specific binding / external side of plasma membrane /  protein phosphorylation / protein serine kinase activity / protein phosphorylation / protein serine kinase activity /  自然免疫系 / protein serine/threonine kinase activity / 自然免疫系 / protein serine/threonine kinase activity /  protein kinase binding / positive regulation of DNA-templated transcription / magnesium ion binding / protein homodimerization activity / protein-containing complex / protein kinase binding / positive regulation of DNA-templated transcription / magnesium ion binding / protein homodimerization activity / protein-containing complex /  ATP binding / identical protein binding / ATP binding / identical protein binding /  細胞質基質 / 細胞質基質 /  細胞質 細胞質類似検索 - 分子機能 | |||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.71 Å クライオ電子顕微鏡法 / 解像度: 3.71 Å | |||||||||
 データ登録者 データ登録者 | Kosek D / Honzejkova K / Obsilova V / Obsil T | |||||||||
| 資金援助 |  チェコ, 2件 チェコ, 2件
| |||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2024 ジャーナル: Elife / 年: 2024タイトル: The cryo-EM structure of ASK1 reveals an asymmetric architecture allosterically modulated by TRX1. 著者: Karolina Honzejkova / Dalibor Kosek / Veronika Obsilova / Tomas Obsil /  要旨: Apoptosis signal-regulating kinase 1 (ASK1) is a crucial stress sensor, directing cells toward apoptosis, differentiation, and senescence via the p38 and JNK signaling pathways. ASK1 dysregulation ...Apoptosis signal-regulating kinase 1 (ASK1) is a crucial stress sensor, directing cells toward apoptosis, differentiation, and senescence via the p38 and JNK signaling pathways. ASK1 dysregulation has been associated with cancer and inflammatory, cardiovascular, and neurodegenerative diseases, among others. However, our limited knowledge of the underlying structural mechanism of ASK1 regulation hampers our ability to target this member of the MAP3K protein family towards developing therapeutic interventions for these disorders. Nevertheless, as a multidomain Ser/Thr protein kinase, ASK1 is regulated by a complex mechanism involving dimerization and interactions with several other proteins, including thioredoxin 1 (TRX1). Thus, the present study aims at structurally characterizing ASK1 and its complex with TRX1 using several biophysical techniques. As shown by cryo-EM analysis, in a state close to its active form, ASK1 is a compact and asymmetric dimer, which enables extensive interdomain and interchain interactions. These interactions stabilize the active conformation of the ASK1 kinase domain. In turn, TRX1 functions as a negative allosteric effector of ASK1, modifying the structure of the TRX1-binding domain and changing its interaction with the tetratricopeptide repeats domain. Consequently, TRX1 reduces access to the activation segment of the kinase domain. Overall, our findings not only clarify the role of ASK1 dimerization and inter-domain contacts but also provide key mechanistic insights into its regulation, thereby highlighting the potential of ASK1 protein-protein interactions as targets for anti-inflammatory therapy. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_18396.map.gz emd_18396.map.gz | 138.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-18396-v30.xml emd-18396-v30.xml emd-18396.xml emd-18396.xml | 20.5 KB 20.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
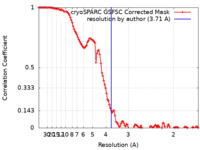
| FSC (解像度算出) |  emd_18396_fsc.xml emd_18396_fsc.xml | 11.3 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_18396.png emd_18396.png | 88.2 KB | ||
| マスクデータ |  emd_18396_msk_1.map emd_18396_msk_1.map | 149.9 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-18396.cif.gz emd-18396.cif.gz | 6.5 KB | ||
| その他 |  emd_18396_additional_1.map.gz emd_18396_additional_1.map.gz emd_18396_half_map_1.map.gz emd_18396_half_map_1.map.gz emd_18396_half_map_2.map.gz emd_18396_half_map_2.map.gz | 74 MB 139.1 MB 139.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-18396 http://ftp.pdbj.org/pub/emdb/structures/EMD-18396 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18396 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18396 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8qgyMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_18396.map.gz / 形式: CCP4 / 大きさ: 149.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_18396.map.gz / 形式: CCP4 / 大きさ: 149.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | main map, sharpened in phenix.autosharpen | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.8336 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_18396_msk_1.map emd_18396_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
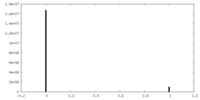
| 投影像・断面図 |
| ||||||||||||
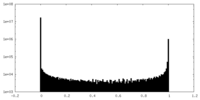
| 密度ヒストグラム |
-追加マップ: mask
| ファイル | emd_18396_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | mask | ||||||||||||
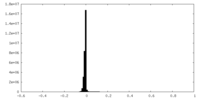
| 投影像・断面図 |
| ||||||||||||
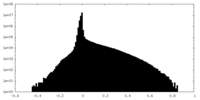
| 密度ヒストグラム |
-ハーフマップ: halfmapA
| ファイル | emd_18396_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | halfmapA | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: halfmapB
| ファイル | emd_18396_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | halfmapB | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : C-terminally truncated Apoptosis signal-regulating kinase 1
| 全体 | 名称: C-terminally truncated Apoptosis signal-regulating kinase 1 |
|---|---|
| 要素 |
|
-超分子 #1: C-terminally truncated Apoptosis signal-regulating kinase 1
| 超分子 | 名称: C-terminally truncated Apoptosis signal-regulating kinase 1 タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 203.5 KDa |
-分子 #1: Mitogen-activated protein kinase kinase kinase 5
| 分子 | 名称: Mitogen-activated protein kinase kinase kinase 5 / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 101.908625 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: MSRRTTVAYV INEASQGQLV VAESEALQSL REACETVGAT LETLHFGKLD FGETTVLDRF YNADIAVVEM SDAFRQPSLF YHLGVRESF SMANNIILYC DTNSDSLQSL KEIICQKNTM CTGNYTFVPY MITPHNKVYC CDSSFMKGLT ELMQPNFELL L GPICLPLV ...文字列: MSRRTTVAYV INEASQGQLV VAESEALQSL REACETVGAT LETLHFGKLD FGETTVLDRF YNADIAVVEM SDAFRQPSLF YHLGVRESF SMANNIILYC DTNSDSLQSL KEIICQKNTM CTGNYTFVPY MITPHNKVYC CDSSFMKGLT ELMQPNFELL L GPICLPLV DRFIQLLKVA QASSSQYFRE SILNDIRKAR NLYTGKELAA ELARIRQRVD NIEVLTADIV INLLLSYRDI QD YDSIVKL VETLEKLPTF DLASHHHVKF HYAFALNRRN LPGDRAKALD IMIPMVQSEG QVASDMYCLV GRIYKDMFLD SNF TDTESR DHGASWFKKA FESEPTLQSG INYAVLLLAA GHQFESSFEL RKVGVKLSSL LGKKGNLEKL QSYWEVGFFL GASV LANDH MRVIQASEKL FKLKTPAWYL KSIVETILIY KHFVKLTTEQ PVAKQELVDF WMDFLVEATK TDVTVVRFPV LILEP TKIY QPSYLSINNE VEEKTISIWH VLPDDKKGIH EWNFSASSVR GVSISKFEER CCFLYVLHNS DDFQIYFCTE LHCKKF FEM VNTITEEKGR STEEGDCESD LLEYDYEYDE NGDRVVLGKG TYGIVYAGRD LSNQVRIAIK EIPERDSRYS QPLHEEI AL HKHLKHKNIV QYLGSFSENG FIKIFMEQVP GGSLSALLRS KWGPLKDNEQ TIGFYTKQIL EGLKYLHDNQ IVHRDIKG D NVLINTYSGV LKISDFGTSK RLAGINPCTE TFTGTLQYMA PEIIDKGPRG YGKAADIWSL GCTIIEMATG KPPFYELGE PQAAMFKVGM FKVHPEIPES MSAEAKAFIL KCFEPDPDKR ACANDLLVDE FLKVSSKKKK TQPKLSALSA GSNEYLRRIS LPVPVLVNS SHHHHHH UniProtKB: Mitogen-activated protein kinase kinase kinase 5 |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.7 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 詳細: 20 mM Tris-HCl 7.5 150 mM NaCl 2 mM 2-mercaptoethanol 3.8 mM CHAPSO |
| グリッド | モデル: UltrAuFoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 前処理 - タイプ: PLASMA CLEANING / 前処理 - 時間: 45 sec. / 前処理 - 雰囲気: OTHER / 詳細: Gatan Solarus II 955 |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 298 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy最大 デフォーカス(公称値): 2.8000000000000003 µm 最小 デフォーカス(公称値): 0.7000000000000001 µm 倍率(公称値): 105000 |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 撮影したグリッド数: 2 / 実像数: 5781 / 平均電子線量: 40.0 e/Å2 詳細: 40 frames 2704 micrographs with 0 degree tilt, 8691 micrographs with 40 degree tilt, 5781 selected for analysis |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 初期モデル |
| ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT | ||||||||||||
| 得られたモデル |  PDB-8qgy: |
 ムービー
ムービー コントローラー
コントローラー




 Z
Z Y
Y X
X