+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | 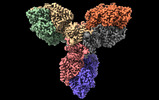 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Single particle cryo-EM structure of homohexameric 2-oxoglutarate dehydrogenase OdhA from Corynebacterium glutamicum with Coenzyme A bound to the E2o domain | |||||||||
 マップデータ マップデータ | OdhA_CoA main map (dens_mod) | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  2-oxoglutarate dehydrogenase / ODH / 2-oxoglutarate dehydrogenase / ODH /  OXIDOREDUCTASE (酸化還元酵素) OXIDOREDUCTASE (酸化還元酵素) | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 oxoglutarate dehydrogenase (succinyl-transferring) / oxoglutarate dehydrogenase (succinyl-transferring) /  oxoglutarate dehydrogenase (succinyl-transferring) activity / oxoglutarate dehydrogenase (succinyl-transferring) activity /  dihydrolipoyllysine-residue succinyltransferase / dihydrolipoyllysine-residue succinyltransferase /  dihydrolipoyllysine-residue succinyltransferase activity / dihydrolipoyllysine-residue succinyltransferase activity /  thiamine pyrophosphate binding / thiamine pyrophosphate binding /  クエン酸回路 / magnesium ion binding クエン酸回路 / magnesium ion binding類似検索 - 分子機能 | |||||||||
| 生物種 |   Corynebacterium glutamicum ATCC 13032 (バクテリア) Corynebacterium glutamicum ATCC 13032 (バクテリア) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 2.17 Å クライオ電子顕微鏡法 / 解像度: 2.17 Å | |||||||||
 データ登録者 データ登録者 | Yang L / Mechaly AM / Bellinzoni M | |||||||||
| 資金援助 |  フランス, 2件 フランス, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2023 ジャーナル: Nat Commun / 年: 2023タイトル: High resolution cryo-EM and crystallographic snapshots of the actinobacterial two-in-one 2-oxoglutarate dehydrogenase. 著者: Lu Yang / Tristan Wagner / Ariel Mechaly / Alexandra Boyko / Eduardo M Bruch / Daniela Megrian / Francesca Gubellini / Pedro M Alzari / Marco Bellinzoni /     要旨: Actinobacteria possess unique ways to regulate the oxoglutarate metabolic node. Contrary to most organisms in which three enzymes compose the 2-oxoglutarate dehydrogenase complex (ODH), ...Actinobacteria possess unique ways to regulate the oxoglutarate metabolic node. Contrary to most organisms in which three enzymes compose the 2-oxoglutarate dehydrogenase complex (ODH), actinobacteria rely on a two-in-one protein (OdhA) in which both the oxidative decarboxylation and succinyl transferase steps are carried out by the same polypeptide. Here we describe high-resolution cryo-EM and crystallographic snapshots of representative enzymes from Mycobacterium smegmatis and Corynebacterium glutamicum, showing that OdhA is an 800-kDa homohexamer that assembles into a three-blade propeller shape. The obligate trimeric and dimeric states of the acyltransferase and dehydrogenase domains, respectively, are critical for maintaining the overall assembly, where both domains interact via subtle readjustments of their interfaces. Complexes obtained with substrate analogues, reaction products and allosteric regulators illustrate how these domains operate. Furthermore, we provide additional insights into the phosphorylation-dependent regulation of this enzymatic machinery by the signalling protein OdhI. #1:  ジャーナル: Biorxiv / 年: 2023 ジャーナル: Biorxiv / 年: 2023タイトル: High resolution cryo-EM and crystallographic snapshots of the large actinobacterial 2-oxoglutarate dehydrogenase: an all-in-one fusion with unique properties 著者: Yang L / Wagner T / Mechaly A / Boyko A / Bruch EM / Megrian D / Gubellini F / Alzari PM / Bellinzoni M | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_17453.map.gz emd_17453.map.gz | 149 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-17453-v30.xml emd-17453-v30.xml emd-17453.xml emd-17453.xml | 20.7 KB 20.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_17453_fsc.xml emd_17453_fsc.xml | 14.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_17453.png emd_17453.png | 79 KB | ||
| その他 |  emd_17453_half_map_1.map.gz emd_17453_half_map_1.map.gz emd_17453_half_map_2.map.gz emd_17453_half_map_2.map.gz | 199.9 MB 199.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-17453 http://ftp.pdbj.org/pub/emdb/structures/EMD-17453 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-17453 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-17453 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8p5uMC  8p5rC  8p5sC  8p5tC  8p5vC  8p5wC  8p5xC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_17453.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_17453.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | OdhA_CoA main map (dens_mod) | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.86 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: OdhA CoA P18 J100 half map A flipped
| ファイル | emd_17453_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | OdhA_CoA_P18_J100_half_map_A_flipped | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: OdhA CoA P18 J100 half map B flipped
| ファイル | emd_17453_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | OdhA_CoA_P18_J100_half_map_B_flipped | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Homohexameric 2-oxoglutarate dehydrogenase
| 全体 | 名称: Homohexameric 2-oxoglutarate dehydrogenase |
|---|---|
| 要素 |
|
-超分子 #1: Homohexameric 2-oxoglutarate dehydrogenase
| 超分子 | 名称: Homohexameric 2-oxoglutarate dehydrogenase / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:   Corynebacterium glutamicum ATCC 13032 (バクテリア) Corynebacterium glutamicum ATCC 13032 (バクテリア) |
| 分子量 | 理論値: 808 KDa |
-分子 #1: 2-oxoglutarate dehydrogenase E1/E2 component
| 分子 | 名称: 2-oxoglutarate dehydrogenase E1/E2 component / タイプ: protein_or_peptide / ID: 1 / コピー数: 6 / 光学異性体: LEVO EC番号:  oxoglutarate dehydrogenase (succinyl-transferring) oxoglutarate dehydrogenase (succinyl-transferring) |
|---|---|
| 由来(天然) | 生物種:   Corynebacterium glutamicum ATCC 13032 (バクテリア) Corynebacterium glutamicum ATCC 13032 (バクテリア) |
| 分子量 | 理論値: 134.972078 KDa |
| 組換発現 | 生物種:   Escherichia coli BL21(DE3) (大腸菌) Escherichia coli BL21(DE3) (大腸菌) |
| 配列 | 文字列: GSMSSASTFG QNAWLVDEMF QQFQKDPKSV DKEWRELFEA QGGPNTTPAT TEAQPSAPKE SAKPAPKAAP AAKAAPRVET KPADKTAPK AKESSVPQQP KLPEPGQTPI RGIFKSIAKN MDISLEIPTA TSVRDMPARL MFENRAMVND QLKRTRGGKI S FTHIIGYA ...文字列: GSMSSASTFG QNAWLVDEMF QQFQKDPKSV DKEWRELFEA QGGPNTTPAT TEAQPSAPKE SAKPAPKAAP AAKAAPRVET KPADKTAPK AKESSVPQQP KLPEPGQTPI RGIFKSIAKN MDISLEIPTA TSVRDMPARL MFENRAMVND QLKRTRGGKI S FTHIIGYA MVKAVMAHPD MNNSYDVIDG KPTLIVPEHI NLGLAIDLPQ KDGSRALVVA AIKETEKMNF SEFLAAYEDI VA RSRKGKL TMDDYQGVTV SLTNPGGIGT RHSVPRLTKG QGTIIGVGSM DYPAEFQGAS EDRLAELGVG KLVTITSTYD HRV IQGAVS GEFLRTMSRL LTDDSFWDEI FDAMNVPYTP MRWAQDVPNT GVDKNTRVMQ LIEAYRSRGH LIADTNPLSW VQPG MPVPD HRDLDIETHN LTIWDLDRTF NVGGFGGKET MTLREVLSRL RAAYTLKVGS EYTHILDRDE RTWLQDRLEA GMPKP TQAE QKYILQKLNA AEAFENFLQT KYVGQKRFSL EGAEALIPLM DSAIDTAAGQ GLDEVVIGMP HRGRLNVLFN IVGKPL ASI FNEFEGQMEQ GQIGGSGDVK YHLGSEGQHL QMFGDGEIKV SLTANPSHLE AVNPVMEGIV RAKQDYLDKG VDGKTVV PL LLHGDAAFAG LGIVPETINL AKLRGYDVGG TIHIVVNNQI GFTTTPDSSR SMHYATDYAK AFGCPVFHVN GDDPEAVV W VGQLATEYRR RFGKDVFIDL VCYRLRGHNE ADDPSMTQPK MYELITGRET VRAQYTEDLL GRGDLSNEDA EAVVRDFHD QMESVFNEVK EGGKKQAEAQ TGITGSQKLP HGLETNISRE ELLELGQAFA NTPEGFNYHP RVAPVAKKRV SSVTEGGIDW AWGELLAFG SLANSGRLVR LAGEDSRRGT FTQRHAVAID PATAEEFNPL HELAQSKGNN GKFLVYNSAL TEYAGMGFEY G YSVGNEDS IVAWEAQFGD FANGAQTIID EYVSSGEAKW GQTSKLILLL PHGYEGQGPD HSSARIERFL QLCAEGSMTV AQ PSTPANH FHLLRRHALS DLKRPLVIFT PKSMLRNKAA ASAPEDFTEV TKFQSVINDP NVADAAKVKK VMLVSGKLYY ELA KRKEKD GRDDIAIVRI EMLHPIPFNR ISEALAGYPN AEEVLFVQDE PANQGPWPFY QEHLPELIPN MPKMRRVSRR AQSS TATGV AKVHQLEEKQ LIDEAFEA UniProtKB: 2-oxoglutarate dehydrogenase E1/E2 component |
-分子 #2: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 2 / コピー数: 6 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-分子 #3: COENZYME A
| 分子 | 名称: COENZYME A / タイプ: ligand / ID: 3 / コピー数: 6 / 式: COA |
|---|---|
| 分子量 | 理論値: 767.534 Da |
| Chemical component information |  ChemComp-COA: |
-分子 #4: ACETYL COENZYME *A
| 分子 | 名称: ACETYL COENZYME *A / タイプ: ligand / ID: 4 / コピー数: 6 / 式: ACO |
|---|---|
| 分子量 | 理論値: 809.571 Da |
| Chemical component information |  ChemComp-ACO: |
-分子 #5: THIAMINE DIPHOSPHATE
| 分子 | 名称: THIAMINE DIPHOSPHATE / タイプ: ligand / ID: 5 / コピー数: 6 / 式: TPP |
|---|---|
| 分子量 | 理論値: 425.314 Da |
| Chemical component information |  ChemComp-TPP: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 構成要素:
| ||||||
|---|---|---|---|---|---|---|---|
| グリッド | モデル: UltrAuFoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 前処理 - タイプ: PLASMA CLEANING / 前処理 - 時間: 25 sec. / 前処理 - 雰囲気: OTHER | ||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 105000 Bright-field microscopy / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 105000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 撮影したグリッド数: 1 / 実像数: 12202 / 平均露光時間: 1.82 sec. / 平均電子線量: 40.0 e/Å2 / 詳細: Each image was composed of 40 frames |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー









 Z
Z Y
Y X
X


















