+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of complement C3 bound to Trypanosoma brucei ISG65 | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  Complement system (補体) / parasite virulence / trypanosome surface protein / host-pathogen complex / Complement system (補体) / parasite virulence / trypanosome surface protein / host-pathogen complex /  IMMUNE SYSTEM (免疫系) IMMUNE SYSTEM (免疫系) | |||||||||
| 生物種 |   Homo sapiens (ヒト) / Homo sapiens (ヒト) /   Trypanosoma brucei brucei (ブルーストリパノソーマ) Trypanosoma brucei brucei (ブルーストリパノソーマ) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.3 Å クライオ電子顕微鏡法 / 解像度: 3.3 Å | |||||||||
 データ登録者 データ登録者 | Cook AD / Higgins MK | |||||||||
| 資金援助 |  英国, 1件 英国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2024 ジャーナル: Elife / 年: 2024タイトル: Molecular mechanism of complement inhibition by the trypanosome receptor ISG65. 著者: Alexander D Cook / Mark Carrington / Matthew K Higgins /  要旨: African trypanosomes replicate within infected mammals where they are exposed to the complement system. This system centres around complement C3, which is present in a soluble form in serum but ...African trypanosomes replicate within infected mammals where they are exposed to the complement system. This system centres around complement C3, which is present in a soluble form in serum but becomes covalently deposited onto the surfaces of pathogens after proteolytic cleavage to C3b. Membrane-associated C3b triggers different complement-mediated effectors which promote pathogen clearance. To counter complement-mediated clearance, African trypanosomes have a cell surface receptor, ISG65, which binds to C3b and which decreases the rate of trypanosome clearance in an infection model. However, the mechanism by which ISG65 reduces C3b function has not been determined. We reveal through cryogenic electron microscopy that ISG65 has two distinct binding sites for C3b, only one of which is available in C3 and C3d. We show that ISG65 does not block the formation of C3b or the function of the C3 convertase which catalyses the surface deposition of C3b. However, we show that ISG65 forms a specific conjugate with C3b, perhaps acting as a decoy. ISG65 also occludes the binding sites for complement receptors 2 and 3, which may disrupt recruitment of immune cells, including B cells, phagocytes, and granulocytes. This suggests that ISG65 protects trypanosomes by combining multiple approaches to dampen the complement cascade. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_17273.map.gz emd_17273.map.gz | 122.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-17273-v30.xml emd-17273-v30.xml emd-17273.xml emd-17273.xml | 17.5 KB 17.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_17273.png emd_17273.png | 112.5 KB | ||
| Filedesc metadata |  emd-17273.cif.gz emd-17273.cif.gz | 4.6 KB | ||
| その他 |  emd_17273_half_map_1.map.gz emd_17273_half_map_1.map.gz emd_17273_half_map_2.map.gz emd_17273_half_map_2.map.gz | 134.5 MB 134.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-17273 http://ftp.pdbj.org/pub/emdb/structures/EMD-17273 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-17273 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-17273 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_17273.map.gz / 形式: CCP4 / 大きさ: 144.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_17273.map.gz / 形式: CCP4 / 大きさ: 144.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 0.832 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
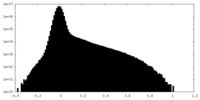
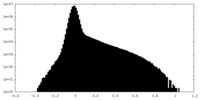
-ハーフマップ: #1
| ファイル | emd_17273_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
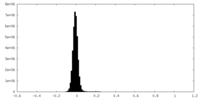
-ハーフマップ: #2
| ファイル | emd_17273_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Complex of Human Complement C3b and T. brucei ISG65
| 全体 | 名称: Complex of Human Complement C3b and T. brucei ISG65 |
|---|---|
| 要素 |
|
-超分子 #1: Complex of Human Complement C3b and T. brucei ISG65
| 超分子 | 名称: Complex of Human Complement C3b and T. brucei ISG65 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) / 組織: Blood Homo sapiens (ヒト) / 組織: Blood |
| 分子量 | 理論値: 44.2 KDa |
-超分子 #2: Human Complement C3b
| 超分子 | 名称: Human Complement C3b / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1-#2 詳細: C3 purified from human serum, C3b generated from C3 by limited trypsin proteolysis. |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) / 組織: Blood Homo sapiens (ヒト) / 組織: Blood |
-超分子 #3: Trypanosoma brucei ISG65
| 超分子 | 名称: Trypanosoma brucei ISG65 / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #3 |
|---|---|
| 由来(天然) | 生物種:   Trypanosoma brucei brucei (ブルーストリパノソーマ) Trypanosoma brucei brucei (ブルーストリパノソーマ) |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2.2 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.4 構成要素:
| |||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY | |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 278.15 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.0 µm Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 特殊光学系 | エネルギーフィルター - 名称: GIF Quantum LS / エネルギーフィルター - スリット幅: 20 eV |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 撮影したグリッド数: 1 / 実像数: 14339 / 平均露光時間: 3.03 sec. / 平均電子線量: 49.9 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 粒子像選択 | 選択した数: 3824878 |
|---|---|
| 初期モデル | モデルのタイプ: INSILICO MODEL / In silico モデル: CryoSPARC Ab initio model |
| 初期 角度割当 | タイプ: PROJECTION MATCHING ソフトウェア: (名称: SIMPLE (ver. 3), cryoSPARC (ver. 3)) |
| 最終 3次元分類 | クラス数: 5 / ソフトウェア - 名称: cryoSPARC (ver. 3) |
| 最終 角度割当 | タイプ: PROJECTION MATCHING / ソフトウェア - 名称: cryoSPARC |
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.3 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: cryoSPARC (ver. 3) / 使用した粒子像数: 481606 |
-原子モデル構築 1
| 初期モデル |
| ||||||
|---|---|---|---|---|---|---|---|
| 精密化 | 空間: REAL / プロトコル: OTHER |
 ムービー
ムービー コントローラー
コントローラー









 Z
Z Y
Y X
X