+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the Tb ADAT2/3 deaminase in complex with tRNA | |||||||||
 マップデータ マップデータ | Final non b factor sharpened cryosparc map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  ADAT (アダット) / ADAT (アダット) /  inosine (イノシン) / tRNA modification / inosine (イノシン) / tRNA modification /  deaminase (脱アミノ) / cryo-EM structure / deaminase (脱アミノ) / cryo-EM structure /  Trypanosoma brucei (ブルーストリパノソーマ) / Trypanosoma brucei (ブルーストリパノソーマ) /  RNA BINDING PROTEIN (RNA結合タンパク質) RNA BINDING PROTEIN (RNA結合タンパク質) | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報tRNA wobble adenosine to inosine editing / tRNA-specific adenosine-34 deaminase activity /  加水分解酵素; ペプチド以外のCN結合加水分解酵素; 環状アミジンに作用 / 加水分解酵素; ペプチド以外のCN結合加水分解酵素; 環状アミジンに作用 /  hydrolase activity / zinc ion binding / hydrolase activity / zinc ion binding /  核質 / 核質 /  細胞質 細胞質類似検索 - 分子機能 | |||||||||
| 生物種 |   Trypanosoma brucei brucei (ブルーストリパノソーマ) / Trypanosoma brucei brucei (ブルーストリパノソーマ) /   Trypanosoma brucei (トリパノソーマ) Trypanosoma brucei (トリパノソーマ) | |||||||||
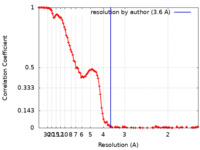
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.6 Å クライオ電子顕微鏡法 / 解像度: 3.6 Å | |||||||||
 データ登録者 データ登録者 | Dolce LG / Tengo L / Weis F / Kowalinski E | |||||||||
| 資金援助 |  フランス, 1件 フランス, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2022 ジャーナル: Nat Commun / 年: 2022タイトル: Structural basis for sequence-independent substrate selection by eukaryotic wobble base tRNA deaminase ADAT2/3. 著者: Luciano G Dolce / Aubree A Zimmer / Laura Tengo / Félix Weis / Mary Anne T Rubio / Juan D Alfonzo / Eva Kowalinski /    要旨: The essential deamination of adenosine A to inosine at the wobble base is the individual tRNA modification with the greatest effects on mRNA decoding, empowering a single tRNA to translate three ...The essential deamination of adenosine A to inosine at the wobble base is the individual tRNA modification with the greatest effects on mRNA decoding, empowering a single tRNA to translate three different codons. To date, many aspects of how eukaryotic deaminases specifically select their multiple substrates remain unclear. Here, using cryo-EM, we present the structure of a eukaryotic ADAT2/3 deaminase bound to a full-length tRNA, revealing that the enzyme distorts the anticodon loop, but in contrast to the bacterial enzymes, selects its substrate via sequence-independent contacts of eukaryote-acquired flexible or intrinsically unfolded motifs distal from the conserved catalytic core. A gating mechanism for substrate entry to the active site is identified. Our multi-step tRNA recognition model yields insights into how RNA editing by A deamination evolved, shaped the genetic code, and directly impacts the eukaryotic proteome. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_15690.map.gz emd_15690.map.gz | 50.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-15690-v30.xml emd-15690-v30.xml emd-15690.xml emd-15690.xml | 18.7 KB 18.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_15690_fsc.xml emd_15690_fsc.xml | 13.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_15690.png emd_15690.png | 51.3 KB | ||
| Filedesc metadata |  emd-15690.cif.gz emd-15690.cif.gz | 6.2 KB | ||
| その他 |  emd_15690_additional_1.map.gz emd_15690_additional_1.map.gz emd_15690_half_map_1.map.gz emd_15690_half_map_1.map.gz emd_15690_half_map_2.map.gz emd_15690_half_map_2.map.gz | 90.3 MB 95.7 MB 95.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-15690 http://ftp.pdbj.org/pub/emdb/structures/EMD-15690 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15690 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15690 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8aw3MC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_15690.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_15690.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Final non b factor sharpened cryosparc map | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.81 Å | ||||||||||||||||||||||||||||||||||||
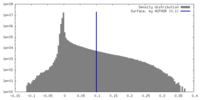
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: deepEMhancer post processed map
| ファイル | emd_15690_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | deepEMhancer post processed map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half map A
| ファイル | emd_15690_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
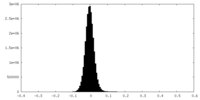
| 密度ヒストグラム |
-ハーフマップ: half map B
| ファイル | emd_15690_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
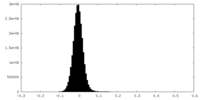
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : ADAT2/3 in complex with tRNA
| 全体 | 名称: ADAT2/3 in complex with tRNA |
|---|---|
| 要素 |
|
-超分子 #1: ADAT2/3 in complex with tRNA
| 超分子 | 名称: ADAT2/3 in complex with tRNA / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 |
|---|---|
| 由来(天然) | 生物種:   Trypanosoma brucei brucei (ブルーストリパノソーマ) Trypanosoma brucei brucei (ブルーストリパノソーマ) |
| 分子量 | 理論値: 90 KDa |
-分子 #1: RNA (75-MER)
| 分子 | 名称: RNA (75-MER) / タイプ: rna / ID: 1 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種:   Trypanosoma brucei (トリパノソーマ) Trypanosoma brucei (トリパノソーマ) |
| 分子量 | 理論値: 24.132311 KDa |
| 配列 | 文字列: GGCCGCUUAG CACAGUGGCA GUGCACCACU CUCGUAAAGU GGGGGUCGCG AGUUCGAUUC UCGCAGUGGC CUCCA GENBANK:  GENBANK: AC091702.3 GENBANK: AC091702.3 |
-分子 #2: Deaminase, putative
| 分子 | 名称: Deaminase, putative / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO EC番号:  加水分解酵素; ペプチド以外のCN結合加水分解酵素; 環状アミジンに作用 加水分解酵素; ペプチド以外のCN結合加水分解酵素; 環状アミジンに作用 |
|---|---|
| 由来(天然) | 生物種:   Trypanosoma brucei brucei (ブルーストリパノソーマ) Trypanosoma brucei brucei (ブルーストリパノソーマ)株: 927/4 GUTat10.1 |
| 分子量 | 理論値: 23.547535 KDa |
| 組換発現 | 生物種:   Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MVQDTGKDTN LKGTAEANES VVYCDVFMQA ALKEATCALE EGEVPVGCVL VKADSSTAAQ AQAGDDLALQ KLIVARGRNA TNRKGHGLA HAEFVAVEEL LRQATAGTSE NIGGGGNSGA VSQDLADYVL YVVVEPCIMC AAMLLYNRVR KVYFGCTNPR F GGNGTVLS ...文字列: MVQDTGKDTN LKGTAEANES VVYCDVFMQA ALKEATCALE EGEVPVGCVL VKADSSTAAQ AQAGDDLALQ KLIVARGRNA TNRKGHGLA HAEFVAVEEL LRQATAGTSE NIGGGGNSGA VSQDLADYVL YVVVEPCIMC AAMLLYNRVR KVYFGCTNPR F GGNGTVLS VHNSYKGCSG EDAALIGYES CGGYRAEEAV VLLQQFYRRE NTNAPLGKRK RKD UniProtKB:  Deaminase, putative Deaminase, putative |
-分子 #3: Deaminase, putative
| 分子 | 名称: Deaminase, putative / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Trypanosoma brucei brucei (ブルーストリパノソーマ) Trypanosoma brucei brucei (ブルーストリパノソーマ)株: 927/4 GUTat10.1 |
| 分子量 | 理論値: 40.600254 KDa |
| 組換発現 | 生物種:   Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: GPDSMEEVVV PEEPPKLVSA LATYVQQERL CTMFLSIANK LLPLKPHACH LKRIRRSSAT RVATAPMDGF AGGVICDKRD SSVATSTIS DGCERNSAAL GTPAAEKSHV LELLLSVGGP VDSSALKELE SAADTTVAVH RVWVPDRAPR SSAEEWTKWC Q IWPFATPK ...文字列: GPDSMEEVVV PEEPPKLVSA LATYVQQERL CTMFLSIANK LLPLKPHACH LKRIRRSSAT RVATAPMDGF AGGVICDKRD SSVATSTIS DGCERNSAAL GTPAAEKSHV LELLLSVGGP VDSSALKELE SAADTTVAVH RVWVPDRAPR SSAEEWTKWC Q IWPFATPK PRVPTQLSEC EVGSIQRIFR TVVMPLAKRL RTDETLGIAA VLVDPSDGYR VLVSSGEEHA LKRGNSAACL GY VSNSGCR KSNRVVLDHP VTFVLKEVTR KQCKDREVEG DASYLANGMD MFVSHEPCVM CSMALVHSRV RRVFYCFPNP VHG GLGSTV SIHAIQELNH HFRVFRCDSR WLSDPEGVSS DHDNPYWEDL TVP UniProtKB:  Deaminase, putative Deaminase, putative |
-分子 #4: ZINC ION
| 分子 | 名称: ZINC ION / タイプ: ligand / ID: 4 / コピー数: 2 / 式: ZN |
|---|---|
| 分子量 | 理論値: 65.409 Da |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.07 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 |
| グリッド | モデル: UltrAuFoil R1.2/1.3 / 材質: GOLD |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 1.8 µm / 最小 デフォーカス(公称値): 1.0 µm Bright-field microscopy / 最大 デフォーカス(公称値): 1.8 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 平均電子線量: 55.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー




 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)