+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of dimeric yeast RNA polymerase II bound to a transcription bubble (consensus map) | |||||||||
 マップデータ マップデータ | RNA Polymerase II dimer loaded with a DNA-RNA scaffold | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  transcription regulation / RNA 3' end processing / transcription termination / transcription regulation / RNA 3' end processing / transcription termination /  GENE REGULATION (遺伝子発現の調節) GENE REGULATION (遺伝子発現の調節) | |||||||||
| 生物種 |   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 4.6 Å クライオ電子顕微鏡法 / 解像度: 4.6 Å | |||||||||
 データ登録者 データ登録者 | Carminati M / Manav MC / Bellini D / Passmore LA | |||||||||
| 資金援助 | European Union, 2件
| |||||||||
 引用 引用 |  ジャーナル: Mol Cell / 年: 2023 ジャーナル: Mol Cell / 年: 2023タイトル: A direct interaction between CPF and RNA Pol II links RNA 3' end processing to transcription. 著者: Manuel Carminati / Juan B Rodríguez-Molina / M Cemre Manav / Dom Bellini / Lori A Passmore /  要旨: Transcription termination by RNA polymerase II (RNA Pol II) is linked to RNA 3' end processing by the cleavage and polyadenylation factor (CPF or CPSF). CPF contains endonuclease, poly(A) polymerase, ...Transcription termination by RNA polymerase II (RNA Pol II) is linked to RNA 3' end processing by the cleavage and polyadenylation factor (CPF or CPSF). CPF contains endonuclease, poly(A) polymerase, and protein phosphatase activities, which cleave and polyadenylate pre-mRNAs and dephosphorylate RNA Pol II to control transcription. Exactly how the RNA 3' end processing machinery is coupled to transcription remains unclear. Here, we combine in vitro reconstitution, structural studies, and genome-wide analyses to show that yeast CPF physically and functionally interacts with RNA Pol II. Surprisingly, CPF-mediated dephosphorylation promotes the formation of an RNA Pol II stalk-to-stalk homodimer in vitro. This dimer is compatible with transcription but not with the binding of transcription elongation factors. Disruption of the dimerization interface in cells causes transcription defects, including altered RNA Pol II abundance on protein-coding genes, tRNA genes, and intergenic regions. We hypothesize that RNA Pol II dimerization may provide a mechanistic basis for the allosteric model of transcription termination. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_15358.map.gz emd_15358.map.gz | 427.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-15358-v30.xml emd-15358-v30.xml emd-15358.xml emd-15358.xml | 19.7 KB 19.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_15358_fsc.xml emd_15358_fsc.xml | 17.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_15358.png emd_15358.png | 50.5 KB | ||
| Filedesc metadata |  emd-15358.cif.gz emd-15358.cif.gz | 5.1 KB | ||
| その他 |  emd_15358_half_map_1.map.gz emd_15358_half_map_1.map.gz emd_15358_half_map_2.map.gz emd_15358_half_map_2.map.gz | 380.4 MB 380.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-15358 http://ftp.pdbj.org/pub/emdb/structures/EMD-15358 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15358 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15358 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_15358.map.gz / 形式: CCP4 / 大きさ: 476.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_15358.map.gz / 形式: CCP4 / 大きさ: 476.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | RNA Polymerase II dimer loaded with a DNA-RNA scaffold | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.83 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #1
| ファイル | emd_15358_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
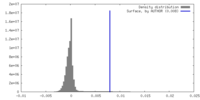
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_15358_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
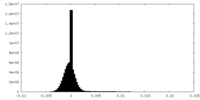
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : S. cerevisiae RNA polymerase II
| 全体 | 名称: S. cerevisiae RNA polymerase II |
|---|---|
| 要素 |
|
-超分子 #1: S. cerevisiae RNA polymerase II
| 超分子 | 名称: S. cerevisiae RNA polymerase II / タイプ: complex / ID: 1 / 親要素: 0 詳細: RNA polymerase 'stalk-to-stalk' homodimer loaded with a DNA-RNA scaffold mimicking a transcription bubble |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母) |
| 分子量 | 理論値: 552 KDa |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.1 mg/mL | ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
詳細: 0.005 % v/v Tween-20 was added to the sample just before vitrification to prevent preferred orientation problems. | ||||||||
| グリッド | モデル: UltrAuFoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 前処理 - タイプ: PLASMA CLEANING / 前処理 - 時間: 45 sec. | ||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.15 K / 装置: FEI VITROBOT MARK IV / 詳細: blot for 4 seconds (force -12) before plunging. | ||||||||
| 詳細 | The vitrified sample contained Pol II (with transcription bubble) bound to Ref2:Glc7:Swd2. We observed a dimeric Pol II population (~10 % of particles) which is reported in the present deposition. |
- 電子顕微鏡法 #1
電子顕微鏡法 #1
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.7 µm / 最小 デフォーカス(公称値): 1.5 µm / 倍率(公称値): 105000 Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.7 µm / 最小 デフォーカス(公称値): 1.5 µm / 倍率(公称値): 105000 |
| 特殊光学系 | エネルギーフィルター - スリット幅: 20 eV |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| Microscopy ID | 1 |
| 詳細 | The movies were collected without tilt or with a tilt angle over a 33-45 degrees range. |
| 撮影 | Image recording ID: 1 フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 撮影したグリッド数: 6 / 実像数: 25411 / 平均電子線量: 40.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 電子顕微鏡法 #1~
電子顕微鏡法 #1~
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.7 µm / 最小 デフォーカス(公称値): 1.5 µm / 倍率(公称値): 75000 Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.7 µm / 最小 デフォーカス(公称値): 1.5 µm / 倍率(公称値): 75000 |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| Microscopy ID | 1 |
| 詳細 | The movies were collected without tilt or with a tilt angle over a 30-40 degrees range. |
| 撮影 | Image recording ID: 2 フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: COUNTING / 撮影したグリッド数: 2 / 実像数: 2035 / 平均電子線量: 40.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - Source name: PDB / Chain - Initial model type: experimental model |
|---|---|
| 詳細 | Two copies of the monomeric Pol II structure (PDB: 5C4X) were rigid fit into the dimeric Pol II EM density |
| 精密化 | プロトコル: RIGID BODY FIT |
 ムービー
ムービー コントローラー
コントローラー







 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)