+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-12861 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Apo-structure of Lassa virus L protein (well-resolved alpha ribbon) [APO-RIBBON] | |||||||||
 マップデータ マップデータ | Apo-structure of Lassa virus L protein (well-resolved alpha ribbon) [APO-RIBBON] | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Lassa virus RNA-dependent RNA polymerase viral RNA /  VIRAL PROTEIN (ウイルスタンパク質) VIRAL PROTEIN (ウイルスタンパク質) | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報negative stranded viral RNA replication /  キャップスナッチング / キャップスナッチング /  virion component / host cell cytoplasm / virion component / host cell cytoplasm /  加水分解酵素; エステル加水分解酵素 / 加水分解酵素; エステル加水分解酵素 /  hydrolase activity / hydrolase activity /  RNA依存性RNAポリメラーゼ / RNA依存性RNAポリメラーゼ /  nucleotide binding / nucleotide binding /  RNA-dependent RNA polymerase activity / RNA-dependent RNA polymerase activity /  metal ion binding metal ion binding類似検索 - 分子機能 | |||||||||
| 生物種 |  Lassa mammarenavirus (ウイルス) Lassa mammarenavirus (ウイルス) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.73 Å クライオ電子顕微鏡法 / 解像度: 3.73 Å | |||||||||
 データ登録者 データ登録者 | Kouba T / Vogel D | |||||||||
| 資金援助 |  ドイツ, 2件 ドイツ, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2021 ジャーナル: Nat Commun / 年: 2021タイトル: Conformational changes in Lassa virus L protein associated with promoter binding and RNA synthesis activity. 著者: Tomas Kouba / Dominik Vogel / Sigurdur R Thorkelsson / Emmanuelle R J Quemin / Harry M Williams / Morlin Milewski / Carola Busch / Stephan Günther / Kay Grünewald / Maria Rosenthal / Stephen Cusack /   要旨: Lassa virus is endemic in West Africa and can cause severe hemorrhagic fever. The viral L protein transcribes and replicates the RNA genome via its RNA-dependent RNA polymerase activity. Here, we ...Lassa virus is endemic in West Africa and can cause severe hemorrhagic fever. The viral L protein transcribes and replicates the RNA genome via its RNA-dependent RNA polymerase activity. Here, we present nine cryo-EM structures of the L protein in the apo-, promoter-bound pre-initiation and active RNA synthesis states. We characterize distinct binding pockets for the conserved 3' and 5' promoter RNAs and show how full-promoter binding induces a distinct pre-initiation conformation. In the apo- and early elongation states, the endonuclease is inhibited by two distinct L protein peptides, whereas in the pre-initiation state it is uninhibited. In the early elongation state, a template-product duplex is bound in the active site cavity together with an incoming non-hydrolysable nucleotide and the full C-terminal region of the L protein, including the putative cap-binding domain, is well-ordered. These data advance our mechanistic understanding of how this flexible and multifunctional molecular machine is activated. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_12861.map.gz emd_12861.map.gz | 51 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-12861-v30.xml emd-12861-v30.xml emd-12861.xml emd-12861.xml | 18.5 KB 18.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_12861_fsc.xml emd_12861_fsc.xml | 10.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_12861.png emd_12861.png | 106.4 KB | ||
| Filedesc metadata |  emd-12861.cif.gz emd-12861.cif.gz | 6.8 KB | ||
| その他 |  emd_12861_additional_1.map.gz emd_12861_additional_1.map.gz emd_12861_half_map_1.map.gz emd_12861_half_map_1.map.gz emd_12861_half_map_2.map.gz emd_12861_half_map_2.map.gz | 96.2 MB 80.9 MB 80.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-12861 http://ftp.pdbj.org/pub/emdb/structures/EMD-12861 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-12861 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-12861 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_12861.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_12861.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Apo-structure of Lassa virus L protein (well-resolved alpha ribbon) [APO-RIBBON] | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.87 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: #1
| ファイル | emd_12861_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
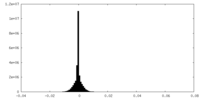
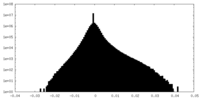
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_12861_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
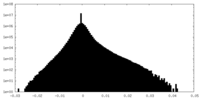
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_12861_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
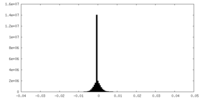
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : RNA-directed RNA polymerase L Lassa mammarenavirus
| 全体 | 名称: RNA-directed RNA polymerase L Lassa mammarenavirus |
|---|---|
| 要素 |
|
-超分子 #1: RNA-directed RNA polymerase L Lassa mammarenavirus
| 超分子 | 名称: RNA-directed RNA polymerase L Lassa mammarenavirus / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Lassa mammarenavirus (ウイルス) Lassa mammarenavirus (ウイルス) |
| 分子量 | 理論値: 253.346 KDa |
-分子 #1: RNA-directed RNA polymerase L
| 分子 | 名称: RNA-directed RNA polymerase L / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO / EC番号:  RNA依存性RNAポリメラーゼ RNA依存性RNAポリメラーゼ |
|---|---|
| 由来(天然) | 生物種:  Lassa mammarenavirus (ウイルス) Lassa mammarenavirus (ウイルス) |
| 分子量 | 理論値: 253.656938 KDa |
| 組換発現 | 生物種:   Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MEDDMACVKD LVSKYLADNE RLSRQKLAFL VQTEPRMLLM EGLKLLSLCI EIDSCNANGC EHNSEDKSVE RILHDHGILT PSLCFVVPD GYKLTGNVLI LLECFVRSSP ANFEQKYIED FKKLEQLKED LKSVDINLIP LIDGRTSFYN EQIPDWVNDK L RDTLFSLL ...文字列: MEDDMACVKD LVSKYLADNE RLSRQKLAFL VQTEPRMLLM EGLKLLSLCI EIDSCNANGC EHNSEDKSVE RILHDHGILT PSLCFVVPD GYKLTGNVLI LLECFVRSSP ANFEQKYIED FKKLEQLKED LKSVDINLIP LIDGRTSFYN EQIPDWVNDK L RDTLFSLL RYAQESNSLF EESEYSRLCE SLSMTSGRLS GVESLNVLLD NRSSHYEEII ASCHQGINNK LTAHEVKLQI EE EYQVFRN RLRKGEITGQ FLKVDKSRLL NDFNNLYVDE VTATKDNIEH LIYQFKRASP ILRFLYANIG EGNGEERHHT IKE CQMQYW RSFLNKVKSL RILNTRRKLL LIFDALILLA SIHDQTRHKC SKGWLGSCFI SVNDRLVSLE STKRDLEKWV GRRQ QSERS NTIQPPDKNQ ILISMFQKTI LKATAALKDV GISVEHYKIN MEVICPDSYD LILNFDVSGV VPTISYQRTE DEKFP FIMG GVELLESTDL ERLSSLSLAL VNSMKTSSTV KLRQNEFGPA RYQVVRCKEA YCQEFLLSGA EFQLIYQKTG ECSKCY AIN DNRVGEICSF YADPKRYFPA IFSAEVLQTT VSTMISWVKD CSELEEQLCN INSLTKMILV LILAHPSKRS QKLLQNL RY FIMAYVSDYH HKDLIDKLRE ELITDVEFLL YRLVRALVNL ILSEDVKSMM TNRFKFILNI SYMCHFITKE TPDRLTDQ I KCFEKFLEPK LEFGHVSINP ADVATEEELD DMVYNAKKFL SKEGCTSIKG PDYKKPGVSK RFLSLLTSSF NNGSLFKES EVKREIKDPL VTSGCATALD LASNKSVVVN KYTDGSRVLN YDFNKLTALA VSQLTEVFSR KGKHLLNKQD YDYKVQQAMS NLVLGPRQN KVGADEADLD EILLDGGASV YFDQLKETVE RIIDQYREPV KPGSNPNGGD QPSVNDLDEV VPNKFYIRLI K GELSNHMV EDFDYDVLPG NFYEEFCDAV YKNNKLKERY FYCGQMSQCP IGELTKAVAT RTYFDQEYFQ CFKSILLIMN AN TLMGRYT HYKSRNLNFK FDMGRLSDDV RISERESNSE ALSKALSLTN CTTAMLKNLC FYSQESPQSY SSTGPDTGRL KFS LSYKEQ VGGNRELYIG DLRTKMFTRL IEDYFEALSL QLSGSCLNNE REFENAILSM KLNVSLAHVS YSMDHSKWGP MMCP FLFLA TLQNLIFLSK DLQADIKGRD YLSTLLTWHM HKMVEIPFNV VSAMMKSFIK AQLGLKKKTT QSITEDFFYS NFQIG VVPS HVSSILDMGQ GILHNTSDFY ALISERFINY AISCICGGTI DAYTSSDDQI SLFDQVLTEL MQRDPEEFKT LIEFHY YMS DQLNKFVSPK SVIGRFVAEF KSRFYVWGDE VPLLTKFVAA ALHNIKCKEP HQLAETIDTI IDQSVANGVP VHLCNLI QK RTLSLLQYAR YPIDPFLLNC ETDVRDWVDG NRSYRIMRQI ERLIPDACGR IRSMLRKLYN KLKTGQLHEE FTTNYLSS E HLSSLSNLCE LLGVEPPSES DLEFSWLNLA AHHPLRMVLR QKIIYSGAVN LDDEKVPTIV KTIQNKLSST FTRGAQKLL SEAINKSAFQ SSIASGFVGL CRTLGSKCVR GPNKESLYIK SIQSLISDIQ GIEPLIDSHG VQYWRVPLNI RDGNEGVISY FRPLLWDYM CISLSTAIEL GAWVLGEPKK VRVLEFFKHN PCDYFPLKPA ASKLLEDRVG LNHIIHSLRR LYPSVFEKHI L PFMSDLAS TKMKWSPRIK FLDLCVALDV NCEALSLVSH IVKWKREEHY IVLSSELRLS HTRTHEPMVE ERVVSTSDAV DN FMRQIYF ESYVRSFVAT TRTLGSFTWF PHKTSVPEGE GLQRLGPFSS FVEKAIHKGI ERPMFKHDLM MGYAWIDFDI EPA RFNHNQ LIASGLVGPR FDSLEDFFDA VESLPPGSAK LSQTVRFRIK SQDASFKESF AIHLDYTGSI NQQTKYLVHE VSAM YSGAV SPCVLSDCWR LVLSGPTFKG KSAWYVDTEI VNEFLTDTNQ LGHVTPVEIV VDMEKLQFTE YDFVLVGPCV EPVPL VVHR GGLWECDKKL ASFTPVVQDQ DLEMFVKEVG DSSLDLLIGA LSAMILDRLK LRMQWSEVDI VSMLKAAMPS NSVKVL NAV LEAVDDWVDF KGYALCYSKS RKKVMVHSSG GKLRLKGRTC EELVKEDEGI EDIE UniProtKB: RNA-directed RNA polymerase L |
-分子 #2: ZINC ION
| 分子 | 名称: ZINC ION / タイプ: ligand / ID: 2 / コピー数: 1 / 式: ZN |
|---|---|
| 分子量 | 理論値: 65.409 Da |
-分子 #3: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 3 / コピー数: 1 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 49.5 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー





























 Z
Z Y
Y X
X