+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-11677 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of W107R after heme uptake (1heme molecule) KatG from M. tuberculosis | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報oxidoreductase activity, acting on a heme group of donors, nitrogenous group as acceptor / Tolerance of reactive oxygen produced by macrophages /  catalase-peroxidase / catalase-peroxidase /  NADH binding / NADH binding /  catalase activity / catalase activity /  NADPH binding / positive regulation of DNA repair / peptidoglycan-based cell wall / hydrogen peroxide catabolic process / NADPH binding / positive regulation of DNA repair / peptidoglycan-based cell wall / hydrogen peroxide catabolic process /  peroxidase activity ...oxidoreductase activity, acting on a heme group of donors, nitrogenous group as acceptor / Tolerance of reactive oxygen produced by macrophages / peroxidase activity ...oxidoreductase activity, acting on a heme group of donors, nitrogenous group as acceptor / Tolerance of reactive oxygen produced by macrophages /  catalase-peroxidase / catalase-peroxidase /  NADH binding / NADH binding /  catalase activity / catalase activity /  NADPH binding / positive regulation of DNA repair / peptidoglycan-based cell wall / hydrogen peroxide catabolic process / NADPH binding / positive regulation of DNA repair / peptidoglycan-based cell wall / hydrogen peroxide catabolic process /  peroxidase activity / cellular response to hydrogen peroxide / response to oxidative stress / response to antibiotic / peroxidase activity / cellular response to hydrogen peroxide / response to oxidative stress / response to antibiotic /  heme binding / extracellular region / heme binding / extracellular region /  metal ion binding / metal ion binding /  細胞膜 / 細胞膜 /  細胞質基質 細胞質基質類似検索 - 分子機能 | |||||||||
| 生物種 |   Mycobacterium tuberculosis (結核菌) Mycobacterium tuberculosis (結核菌) | |||||||||
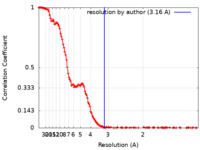
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.16 Å クライオ電子顕微鏡法 / 解像度: 3.16 Å | |||||||||
 データ登録者 データ登録者 | Blundell TL / Chaplin AK / Munir A | |||||||||
| 資金援助 |  英国, 1件 英国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Structure / 年: 2021 ジャーナル: Structure / 年: 2021タイトル: Using cryo-EM to understand antimycobacterial resistance in the catalase-peroxidase (KatG) from Mycobacterium tuberculosis. 著者: Asma Munir / Michael T Wilson / Steven W Hardwick / Dimitri Y Chirgadze / Jonathan A R Worrall / Tom L Blundell / Amanda K Chaplin /  要旨: Resolution advances in cryoelectron microscopy (cryo-EM) now offer the possibility to visualize structural effects of naturally occurring resistance mutations in proteins and also of understanding ...Resolution advances in cryoelectron microscopy (cryo-EM) now offer the possibility to visualize structural effects of naturally occurring resistance mutations in proteins and also of understanding the binding mechanisms of small drug molecules. In Mycobacterium tuberculosis the multifunctional heme enzyme KatG is indispensable for activation of isoniazid (INH), a first-line pro-drug for treatment of tuberculosis. We present a cryo-EM methodology for structural and functional characterization of KatG and INH resistance variants. The cryo-EM structure of the 161 kDa KatG dimer in the presence of INH is reported to 2.7 Å resolution allowing the observation of potential INH binding sites. In addition, cryo-EM structures of two INH resistance variants, identified from clinical isolates, W107R and T275P, are reported. In combination with electronic absorbance spectroscopy our cryo-EM approach reveals how these resistance variants cause disorder in the heme environment preventing heme uptake and retention, providing insight into INH resistance. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_11677.map.gz emd_11677.map.gz | 197.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-11677-v30.xml emd-11677-v30.xml emd-11677.xml emd-11677.xml | 15.9 KB 15.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_11677_fsc.xml emd_11677_fsc.xml | 17.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_11677.png emd_11677.png | 11.8 KB | ||
| その他 |  emd_11677_half_map_1.map.gz emd_11677_half_map_1.map.gz emd_11677_half_map_2.map.gz emd_11677_half_map_2.map.gz | 194.4 MB 194.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-11677 http://ftp.pdbj.org/pub/emdb/structures/EMD-11677 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11677 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11677 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_11677.map.gz / 形式: CCP4 / 大きさ: 209.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_11677.map.gz / 形式: CCP4 / 大きさ: 209.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 0.652 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-ハーフマップ: #1
| ファイル | emd_11677_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_11677_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Cryo-EM structure of W107R KatG after heme uptake from M. tuberculosis
| 全体 | 名称: Cryo-EM structure of W107R KatG after heme uptake from M. tuberculosis |
|---|---|
| 要素 |
|
-超分子 #1: Cryo-EM structure of W107R KatG after heme uptake from M. tuberculosis
| 超分子 | 名称: Cryo-EM structure of W107R KatG after heme uptake from M. tuberculosis タイプ: organelle_or_cellular_component / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:   Mycobacterium tuberculosis (結核菌) Mycobacterium tuberculosis (結核菌) |
| 分子量 | 実験値: 161.1 kDa/nm |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
-分子 #1: Catalase-peroxidase
| 分子 | 名称: Catalase-peroxidase / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO / EC番号:  catalase-peroxidase catalase-peroxidase |
|---|---|
| 由来(天然) | 生物種:   Mycobacterium tuberculosis (結核菌) Mycobacterium tuberculosis (結核菌) |
| 分子量 | 理論値: 80.658594 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: MPEQHPPITE TTTGAASNGC PVVGHMKYPV EGGGNQDWWP NRLNLKVLHQ NPAVADPMGA AFDYAAEVAT IDVDALTRDI EEVMTTSQP WWPADYGHYG PLFIRMARHA AGTYRIHDGR GGAGGGMQRF APLNSWPDNA SLDKARRLLW PVKKKYGKKL S WADLIVFA ...文字列: MPEQHPPITE TTTGAASNGC PVVGHMKYPV EGGGNQDWWP NRLNLKVLHQ NPAVADPMGA AFDYAAEVAT IDVDALTRDI EEVMTTSQP WWPADYGHYG PLFIRMARHA AGTYRIHDGR GGAGGGMQRF APLNSWPDNA SLDKARRLLW PVKKKYGKKL S WADLIVFA GNCALESMGF KTFGFGFGRV DQWEPDEVYW GKEATWLGDE RYSGKRDLEN PLAAVQMGLI YVNPEGPNGN PD PMAAAVD IRETFRRMAM NDVETAALIV GGHTFGKTHG AGPADLVGPE PEAAPLEQMG LGWKSSYGTG TGKDAITSGI EVV WTNTPT KWDNSFLEIL YGYEWELTKS PAGAWQYTAK DGAGAGTIPD PFGGPGRSPT MLATDLSLRV DPIYERITRR WLEH PEELA DEFAKAWYKL IHRDMGPVAR YLGPLVPKQT LLWQDPVPAV SHDLVGEAEI ASLKSQIRAS GLTVSQLVST AWAAA SSFR GSDKRGGANG GRIRLQPQVG WEVNDPDGDL RKVIRTLEEI QESFNSAAPG NIKVSFADLV VLGGCAAIEK AAKAAG HNI TVPFTPGRTD ASQEQTDVES FAVLEPKADG FRNYLGKGNP LPAEYMLLDK ANLLTLSAPE MTVLVGGLRV LGANYKR LP LGVFTEASES LTNDFFVNLL DMGITWEPSP ADDGTYQGKD GSGKVKWTGS RVDLVFGSNS ELRALVEVYG ADDAQPKF V QDFVAAWDKV MNLDRFDVR |
-分子 #2: PROTOPORPHYRIN IX CONTAINING FE
| 分子 | 名称: PROTOPORPHYRIN IX CONTAINING FE / タイプ: ligand / ID: 2 / コピー数: 1 / 式: HEM |
|---|---|
| 分子量 | 理論値: 616.487 Da |
| Chemical component information |  ChemComp-HEM: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 3 mg/mL |
|---|---|
| 緩衝液 | pH: 7.2 |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 277.15 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 検出モード: COUNTING / 撮影したグリッド数: 1 / 実像数: 244867 / 平均電子線量: 39.3 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー

























 Z
Z Y
Y X
X