+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-11127 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Fo domain of Ovine ATP synthase | |||||||||
 マップデータ マップデータ | Map of Fo domain, combined states | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  ATP synthase (ATP合成酵素) / ATP synthase (ATP合成酵素) /  mitochondrial (ミトコンドリア) / mitochondrial (ミトコンドリア) /  respiratory chain (電子伝達系) / respiratory chain (電子伝達系) /  mammalian (哺乳類) / mammalian (哺乳類) /  MEMBRANE PROTEIN (膜タンパク質) MEMBRANE PROTEIN (膜タンパク質) | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報mitochondrial proton-transporting ATP synthase, stator stalk / mitochondrial proton-transporting ATP synthase complex, coupling factor F(o) / proton-transporting ATP synthase complex, coupling factor F(o) / mitochondrial proton-transporting ATP synthase complex / proton motive force-driven ATP synthesis / proton transmembrane transporter activity / proton motive force-driven mitochondrial ATP synthesis / proton-transporting ATP synthase activity, rotational mechanism /  lipid binding lipid binding類似検索 - 分子機能 | |||||||||
| 生物種 |   Ovis aries (ヒツジ) Ovis aries (ヒツジ) | |||||||||
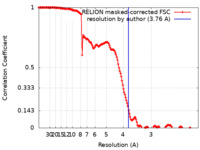
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.76 Å クライオ電子顕微鏡法 / 解像度: 3.76 Å | |||||||||
 データ登録者 データ登録者 | Pinke G / Zhou L | |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2020 ジャーナル: Nat Struct Mol Biol / 年: 2020タイトル: Cryo-EM structure of the entire mammalian F-type ATP synthase. 著者: Gergely Pinke / Long Zhou / Leonid A Sazanov /  要旨: The majority of adenosine triphosphate (ATP) powering cellular processes in eukaryotes is produced by the mitochondrial F1Fo ATP synthase. Here, we present the atomic models of the membrane Fo domain ...The majority of adenosine triphosphate (ATP) powering cellular processes in eukaryotes is produced by the mitochondrial F1Fo ATP synthase. Here, we present the atomic models of the membrane Fo domain and the entire mammalian (ovine) F1Fo, determined by cryo-electron microscopy. Subunits in the membrane domain are arranged in the 'proton translocation cluster' attached to the c-ring and a more distant 'hook apparatus' holding subunit e. Unexpectedly, this subunit is anchored to a lipid 'plug' capping the c-ring. We present a detailed proton translocation pathway in mammalian Fo and key inter-monomer contacts in F1Fo multimers. Cryo-EM maps of F1Fo exposed to calcium reveal a retracted subunit e and a disassembled c-ring, suggesting permeability transition pore opening. We propose a model for the permeability transition pore opening, whereby subunit e pulls the lipid plug out of the c-ring. Our structure will allow the design of drugs for many emerging applications in medicine. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_11127.map.gz emd_11127.map.gz | 5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-11127-v30.xml emd-11127-v30.xml emd-11127.xml emd-11127.xml | 22.3 KB 22.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_11127_fsc.xml emd_11127_fsc.xml | 15.3 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_11127.png emd_11127.png | 69.3 KB | ||
| Filedesc metadata |  emd-11127.cif.gz emd-11127.cif.gz | 6.9 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-11127 http://ftp.pdbj.org/pub/emdb/structures/EMD-11127 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11127 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11127 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_11127.map.gz / 形式: CCP4 / 大きさ: 5.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_11127.map.gz / 形式: CCP4 / 大きさ: 5.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Map of Fo domain, combined states | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.061 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
+全体 : Fo domain of purified ATP synthase from sheep heart mitochondria
+超分子 #1: Fo domain of purified ATP synthase from sheep heart mitochondria
+分子 #1: ATP synthase F(0) complex subunit C1, mitochondrial
+分子 #2: ATP synthase subunit b
+分子 #3: ATP synthase subunit d, mitochondrial
+分子 #4: ATP synthase subunit a
+分子 #5: ATP synthase membrane subunit DAPIT
+分子 #6: ATP synthase membrane subunit 6.8PL
+分子 #7: ATP synthase protein 8
+分子 #8: ATP synthase membrane subunit f
+分子 #9: ATP synthase subunit
+分子 #10: ATP synthase subunit e, mitochondrial
+分子 #11: 1,2-DIPALMITOYL-PHOSPHATIDYL-GLYCEROLE
+分子 #12: CARDIOLIPIN
+分子 #13: O-[(S)-hydroxy{[(2S)-2-hydroxy-3-(octadec-9-enoyloxy)propyl]oxy}p...
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2.8 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 詳細: 20 mM HEPES pH7.4, 35 mM NaCl, 2 mM EDTA, 1% sucrose, 1 mM DTT, 0.1% LMNG, 0.2% CHAPS |
| グリッド | モデル: Quantifoil R0.6/1 / 材質: COPPER / メッシュ: 400 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 120 sec. / 前処理 - 雰囲気: AIR |
| 凍結 | 凍結剤: ETHANE-PROPANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 最大 デフォーカス(補正後): 2.4 µm / 最小 デフォーカス(補正後): 1.0 µm / 倍率(補正後): 131951 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm Bright-field microscopy / Cs: 2.7 mm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: INTEGRATING / デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / 撮影したグリッド数: 1 / 実像数: 3053 / 平均露光時間: 1.0 sec. / 平均電子線量: 106.0 e/Å2 / 詳細: 40 frames per movie |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー