+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | 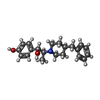 データベース: PDB化学物質要素 / ID: QEL データベース: PDB化学物質要素 / ID: QEL | ||||
|---|---|---|---|---|---|
| 名称 | 名称: 別称: Ifenprodil 日化辞名称: (1R,2S)-2-(4-ベンジルピペリジノ)-1-(4-ヒドロキシフェニル)プロパン-1-オ-ル ウィキペディア |  ウィキペディア - イフェンプロジル: イフェンプロジル(Ifenprodil)は、NMDA受容体、特にGluN1(グリシン結合NMDA受容体サブユニット1)およびGluN2B(グルタミン酸結合NMDA受容体サブユニット2)の阻害剤である。 またイフェンプロジルは、GIRKチャネルを阻害し、アドレナリンα1受容体、セロトニン受容体、Σ受容体と相互作用する... ウィキペディア - イフェンプロジル: イフェンプロジル(Ifenprodil)は、NMDA受容体、特にGluN1(グリシン結合NMDA受容体サブユニット1)およびGluN2B(グルタミン酸結合NMDA受容体サブユニット2)の阻害剤である。 またイフェンプロジルは、GIRKチャネルを阻害し、アドレナリンα1受容体、セロトニン受容体、Σ受容体と相互作用する...コメント | 阻害剤*YM | |
-Chemical information
| 組成 |  | ||||||||
|---|---|---|---|---|---|---|---|---|---|
| その他 | タイプ: NON-POLYMER / PDB分類: HETAIN / 3文字コード: QEL / 理論座標の詳細: Corina | ||||||||
| 履歴 |
| ||||||||
 外部リンク 外部リンク |  UniChem / UniChem /  BindingDB / BindingDB /  ChEMBL / ChEMBL /  日化辞 / 日化辞 /  PubChem / PubChem /  SureChEMBL / SureChEMBL /  ZINC / ZINC /  ChemSpider / ChemSpider /  Wikipedia search / Wikipedia search /  Google search Google search |
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
-詳細
-SMILES
| ACDLabs 12.01 | | CACTVS 3.370 | OpenEye OEToolkits 1.7.0 | |
|---|
-SMILES CANONICAL
| CACTVS 3.370 | | OpenEye OEToolkits 1.7.0 | |
|---|
-InChI
| InChI 1.03 |
|---|
-InChIKey
| InChI 1.03 |
|---|
-SYSTEMATIC NAME
| ACDLabs 12.01 | | OpenEye OEToolkits 1.7.0 | |
|---|
-PDBエントリ
全4件を表示しています

PDB-3qel: 
Crystal structure of amino terminal domains of the NMDA receptor subunit GluN1 and GluN2B in complex with ifenprodil

PDB-4pe5: 
Crystal Structure of GluN1a/GluN2B NMDA Receptor Ion Channel

PDB-5ewj: 
CRYSTAL STRUCTURE OF AMINO TERMINAL DOMAINS OF THE NMDA RECEPTOR SUBUNIT GLUN1 AND GLUN2B IN COMPLEX WITH IFENPRODIL

PDB-7aqi: 
Structure of SARS-CoV-2 Main Protease bound to Ifenprodil
 ムービー
ムービー コントローラー
コントローラー



