+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 7lw1 | ||||||
|---|---|---|---|---|---|---|---|
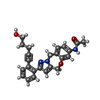
| タイトル | Human phosphofructokinase-1 liver type bound to activator NA-11 | ||||||
 要素 要素 | ATP-dependent 6-phosphofructokinase, liver type | ||||||
 キーワード キーワード |  TRANSFERASE (転移酵素) / TRANSFERASE (転移酵素) /  glycolysis (解糖系) / glycolysis (解糖系) /  phosphofructokinase (ホスホフルクトキナーゼ) / liver type / phosphofructokinase (ホスホフルクトキナーゼ) / liver type /  activated activated | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 fructose binding / fructose binding /  6-phosphofructokinase complex / 6-phosphofructokinase complex /  6-phosphofructokinase / 6-phosphofructokinase /  fructose-6-phosphate binding / fructose-6-phosphate binding /  6-phosphofructokinase activity / fructose 1,6-bisphosphate metabolic process / fructose 6-phosphate metabolic process / 6-phosphofructokinase activity / fructose 1,6-bisphosphate metabolic process / fructose 6-phosphate metabolic process /  monosaccharide binding / canonical glycolysis / monosaccharide binding / canonical glycolysis /  解糖系 ... 解糖系 ... fructose binding / fructose binding /  6-phosphofructokinase complex / 6-phosphofructokinase complex /  6-phosphofructokinase / 6-phosphofructokinase /  fructose-6-phosphate binding / fructose-6-phosphate binding /  6-phosphofructokinase activity / fructose 1,6-bisphosphate metabolic process / fructose 6-phosphate metabolic process / 6-phosphofructokinase activity / fructose 1,6-bisphosphate metabolic process / fructose 6-phosphate metabolic process /  monosaccharide binding / canonical glycolysis / monosaccharide binding / canonical glycolysis /  解糖系 / AMP binding / negative regulation of insulin secretion / response to glucose / glycolytic process / 解糖系 / AMP binding / negative regulation of insulin secretion / response to glucose / glycolytic process /  kinase binding / secretory granule lumen / ficolin-1-rich granule lumen / Neutrophil degranulation / extracellular exosome / extracellular region / kinase binding / secretory granule lumen / ficolin-1-rich granule lumen / Neutrophil degranulation / extracellular exosome / extracellular region /  ATP binding / ATP binding /  生体膜 / identical protein binding / 生体膜 / identical protein binding /  metal ion binding / metal ion binding /  細胞質基質 細胞質基質類似検索 - 分子機能 | ||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||
| 手法 |  電子顕微鏡法 / 電子顕微鏡法 /  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 2.9 Å クライオ電子顕微鏡法 / 解像度: 2.9 Å | ||||||
 データ登録者 データ登録者 | Lynch, E.M. / Kollman, J.M. / Webb, B. | ||||||
| 資金援助 |  米国, 1件 米国, 1件
| ||||||
 引用 引用 |  ジャーナル: Cell / 年: 2021 ジャーナル: Cell / 年: 2021タイトル: Selective activation of PFKL suppresses the phagocytic oxidative burst. 著者: Neri Amara / Madison P Cooper / Maria A Voronkova / Bradley A Webb / Eric M Lynch / Justin M Kollman / Taylur Ma / Kebing Yu / Zijuan Lai / Dewakar Sangaraju / Nobuhiko Kayagaki / Kim Newton ...著者: Neri Amara / Madison P Cooper / Maria A Voronkova / Bradley A Webb / Eric M Lynch / Justin M Kollman / Taylur Ma / Kebing Yu / Zijuan Lai / Dewakar Sangaraju / Nobuhiko Kayagaki / Kim Newton / Matthew Bogyo / Steven T Staben / Vishva M Dixit /  要旨: In neutrophils, nicotinamide adenine dinucleotide phosphate (NADPH) generated via the pentose phosphate pathway fuels NADPH oxidase NOX2 to produce reactive oxygen species for killing invading ...In neutrophils, nicotinamide adenine dinucleotide phosphate (NADPH) generated via the pentose phosphate pathway fuels NADPH oxidase NOX2 to produce reactive oxygen species for killing invading pathogens. However, excessive NOX2 activity can exacerbate inflammation, as in acute respiratory distress syndrome (ARDS). Here, we use two unbiased chemical proteomic strategies to show that small-molecule LDC7559, or a more potent designed analog NA-11, inhibits the NOX2-dependent oxidative burst in neutrophils by activating the glycolytic enzyme phosphofructokinase-1 liver type (PFKL) and dampening flux through the pentose phosphate pathway. Accordingly, neutrophils treated with NA-11 had reduced NOX2-dependent outputs, including neutrophil cell death (NETosis) and tissue damage. A high-resolution structure of PFKL confirmed binding of NA-11 to the AMP/ADP allosteric activation site and explained why NA-11 failed to agonize phosphofructokinase-1 platelet type (PFKP) or muscle type (PFKM). Thus, NA-11 represents a tool for selective activation of PFKL, the main phosphofructokinase-1 isoform expressed in immune cells. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  7lw1.cif.gz 7lw1.cif.gz | 923.4 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb7lw1.ent.gz pdb7lw1.ent.gz | 808.1 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  7lw1.json.gz 7lw1.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/lw/7lw1 https://data.pdbj.org/pub/pdb/validation_reports/lw/7lw1 ftp://data.pdbj.org/pub/pdb/validation_reports/lw/7lw1 ftp://data.pdbj.org/pub/pdb/validation_reports/lw/7lw1 | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
| #1: タンパク質 | 分子量: 85120.375 Da / 分子数: 4 / 由来タイプ: 組換発現 / 由来: (組換発現)   Homo sapiens (ヒト) / 遺伝子: PFKL / 発現宿主: Homo sapiens (ヒト) / 遺伝子: PFKL / 発現宿主:  unidentified baculovirus (ウイルス) / 参照: UniProt: P17858, unidentified baculovirus (ウイルス) / 参照: UniProt: P17858,  6-phosphofructokinase 6-phosphofructokinase#2: 化合物 | ChemComp-YG1 / #3: 糖 | ChemComp-FBP /  フルクトース-1,6-ビスリン酸 フルクトース-1,6-ビスリン酸#4: 糖 | ChemComp-F6P /  フルクトース-6-リン酸 フルクトース-6-リン酸#5: 化合物 | ChemComp-ADP /  アデノシン二リン酸 アデノシン二リン酸研究の焦点であるリガンドがあるか | Y | |
|---|
-実験情報
-実験
| 実験 | 手法:  電子顕微鏡法 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法:  単粒子再構成法 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: Human phosphofructokinase-1 liver type bound to activator NA-11 タイプ: COMPLEX / Entity ID: #1 / 由来: RECOMBINANT |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 由来(組換発現) | 生物種:  unidentified baculovirus (ウイルス) unidentified baculovirus (ウイルス) |
| 緩衝液 | pH: 7.5 |
| 試料 | 包埋: NO / シャドウイング: NO / 染色 : NO / 凍結 : NO / 凍結 : YES : YES |
| 試料支持 | グリッドの材料: COPPER / グリッドのサイズ: 400 divisions/in. / グリッドのタイプ: C-flat-2/2 |
急速凍結 | 装置: FEI VITROBOT MARK IV / 凍結剤: ETHANE / 湿度: 100 % |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS |
| 電子銃 | 電子線源 : :  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD Bright-field microscopy / 倍率(公称値): 130000 X / 最大 デフォーカス(公称値): 2800 nm / 最小 デフォーカス(公称値): 700 nm / Cs Bright-field microscopy / 倍率(公称値): 130000 X / 最大 デフォーカス(公称値): 2800 nm / 最小 デフォーカス(公称値): 700 nm / Cs : 2.7 mm : 2.7 mm |
| 試料ホルダ | 凍結剤: NITROGEN 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 撮影 | 平均露光時間: 10 sec. / 電子線照射量: 90 e/Å2 / 検出モード: SUPER-RESOLUTION フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 撮影したグリッド数: 1 / 実像数: 2529 |
| 画像スキャン | 横: 3710 / 縦: 3838 / 動画フレーム数/画像: 50 / 利用したフレーム数/画像: 1-50 |
- 解析
解析
| EMソフトウェア |
| ||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
CTF補正 | タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION | ||||||||||||||||||||||||||||||||
| 粒子像の選択 | 選択した粒子像数: 3000000 | ||||||||||||||||||||||||||||||||
| 対称性 | 点対称性 : D2 (2回x2回 2面回転対称 : D2 (2回x2回 2面回転対称 ) ) | ||||||||||||||||||||||||||||||||
3次元再構成 | 解像度: 2.9 Å / 解像度の算出法: FSC 0.5 CUT-OFF / 粒子像の数: 63296 / 対称性のタイプ: POINT |
 ムービー
ムービー コントローラー
コントローラー













 PDBj
PDBj