+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 7bkd | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Formate dehydrogenase - heterodisulfide reductase - formylmethanofuran dehydrogenase complex from Methanospirillum hungatei (heterodislfide reductase core and mobile arm in conformational state 1, composite structure) | |||||||||||||||
 要素 要素 |
| |||||||||||||||
 キーワード キーワード |  OXIDOREDUCTASE (酸化還元酵素) / OXIDOREDUCTASE (酸化還元酵素) /  methanogenesis (メタン生成経路) / flavin-based electron bifurcation / CO2-fixation / methanogenesis (メタン生成経路) / flavin-based electron bifurcation / CO2-fixation /  formate dehydrogenase (ギ酸デヒドロゲナーゼ) formate dehydrogenase (ギ酸デヒドロゲナーゼ) | |||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 Oxidoreductases; Acting on the aldehyde or oxo group of donors; With unknown physiological acceptors / oxidoreductase activity, acting on CH or CH2 groups, with an iron-sulfur protein as acceptor / Oxidoreductases; Acting on the aldehyde or oxo group of donors; With unknown physiological acceptors / oxidoreductase activity, acting on CH or CH2 groups, with an iron-sulfur protein as acceptor /  CoB--CoM heterodisulfide reductase activity / formate metabolic process / CoB--CoM heterodisulfide reductase activity / formate metabolic process /  酸化還元酵素; 含硫化合物に対し酸化酵素として働く / 酸化還元酵素; 含硫化合物に対し酸化酵素として働く /  メタン生成経路 / formate dehydrogenase (NAD+) activity / molybdopterin cofactor binding / メタン生成経路 / formate dehydrogenase (NAD+) activity / molybdopterin cofactor binding /  iron-sulfur cluster binding / 4 iron, 4 sulfur cluster binding ... iron-sulfur cluster binding / 4 iron, 4 sulfur cluster binding ... Oxidoreductases; Acting on the aldehyde or oxo group of donors; With unknown physiological acceptors / oxidoreductase activity, acting on CH or CH2 groups, with an iron-sulfur protein as acceptor / Oxidoreductases; Acting on the aldehyde or oxo group of donors; With unknown physiological acceptors / oxidoreductase activity, acting on CH or CH2 groups, with an iron-sulfur protein as acceptor /  CoB--CoM heterodisulfide reductase activity / formate metabolic process / CoB--CoM heterodisulfide reductase activity / formate metabolic process /  酸化還元酵素; 含硫化合物に対し酸化酵素として働く / 酸化還元酵素; 含硫化合物に対し酸化酵素として働く /  メタン生成経路 / formate dehydrogenase (NAD+) activity / molybdopterin cofactor binding / メタン生成経路 / formate dehydrogenase (NAD+) activity / molybdopterin cofactor binding /  iron-sulfur cluster binding / 4 iron, 4 sulfur cluster binding / iron-sulfur cluster binding / 4 iron, 4 sulfur cluster binding /  oxidoreductase activity / oxidoreductase activity /  metal ion binding metal ion binding類似検索 - 分子機能 | |||||||||||||||
| 生物種 |   Methanospirillum hungatei JF-1 (古細菌) Methanospirillum hungatei JF-1 (古細菌) | |||||||||||||||
| 手法 |  電子顕微鏡法 / 電子顕微鏡法 /  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3 Å クライオ電子顕微鏡法 / 解像度: 3 Å | |||||||||||||||
 データ登録者 データ登録者 | Pfeil-Gardiner, O. / Watanabe, T. / Shima, S. / Murphy, B.J. | |||||||||||||||
| 資金援助 |  ドイツ, ドイツ,  日本, 4件 日本, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Science / 年: 2021 ジャーナル: Science / 年: 2021タイトル: Three-megadalton complex of methanogenic electron-bifurcating and CO-fixing enzymes. 著者: Tomohiro Watanabe / Olivia Pfeil-Gardiner / Jörg Kahnt / Jürgen Koch / Seigo Shima / Bonnie J Murphy /  要旨: The first reaction of the methanogenic pathway from carbon dioxide (CO) is the reduction and condensation of CO to formyl-methanofuran, catalyzed by formyl-methanofuran dehydrogenase (Fmd). Strongly ...The first reaction of the methanogenic pathway from carbon dioxide (CO) is the reduction and condensation of CO to formyl-methanofuran, catalyzed by formyl-methanofuran dehydrogenase (Fmd). Strongly reducing electrons for this reaction are generated by heterodisulfide reductase (Hdr) in complex with hydrogenase or formate dehydrogenase (Fdh) using a flavin-based electron-bifurcation mechanism. Here, we report enzymological and structural characterizations of Fdh-Hdr-Fmd complexes from . The complexes catalyze this reaction using electrons from formate and the reduced form of the electron carrier F. Conformational changes in HdrA mediate electron bifurcation, and polyferredoxin FmdF directly transfers electrons to the CO reduction site, as evidenced by methanofuran-dependent flavin-based electron bifurcation even without free ferredoxin, a diffusible electron carrier between Hdr and Fmd. Conservation of Hdr and Fmd structures suggests that this complex is common among hydrogenotrophic methanogens. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  7bkd.cif.gz 7bkd.cif.gz | 555.8 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb7bkd.ent.gz pdb7bkd.ent.gz | 462.5 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  7bkd.json.gz 7bkd.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/bk/7bkd https://data.pdbj.org/pub/pdb/validation_reports/bk/7bkd ftp://data.pdbj.org/pub/pdb/validation_reports/bk/7bkd ftp://data.pdbj.org/pub/pdb/validation_reports/bk/7bkd | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
-タンパク質 , 4種, 5分子 AaFED
| #1: タンパク質 | 分子量: 72885.062 Da / 分子数: 2 / 由来タイプ: 天然 / 由来: (天然)   Methanospirillum hungatei JF-1 (古細菌) Methanospirillum hungatei JF-1 (古細菌)参照: UniProt: Q2FKZ1,  酸化還元酵素; 含硫化合物に対し酸化酵素として働く 酸化還元酵素; 含硫化合物に対し酸化酵素として働く#4: タンパク質 | | 分子量: 15692.425 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)   Methanospirillum hungatei JF-1 (古細菌) / 参照: UniProt: Q2FKZ0 Methanospirillum hungatei JF-1 (古細菌) / 参照: UniProt: Q2FKZ0#5: タンパク質 | |  ギ酸デヒドロゲナーゼ ギ酸デヒドロゲナーゼ分子量: 45639.895 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)   Methanospirillum hungatei JF-1 (古細菌) Methanospirillum hungatei JF-1 (古細菌)参照: UniProt: Q2FME3,  Oxidoreductases; Acting on the aldehyde or oxo group of donors; With unknown physiological acceptors Oxidoreductases; Acting on the aldehyde or oxo group of donors; With unknown physiological acceptors#6: タンパク質 | |  ギ酸デヒドロゲナーゼ ギ酸デヒドロゲナーゼ分子量: 75911.445 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)   Methanospirillum hungatei JF-1 (古細菌) / 参照: UniProt: Q2FRK1, Methanospirillum hungatei JF-1 (古細菌) / 参照: UniProt: Q2FRK1,  ギ酸デヒドロゲナーゼ ギ酸デヒドロゲナーゼ |
|---|
-CoB--CoM heterodisulfide reductase subunit ... , 2種, 4分子 CcBb
| #2: タンパク質 |  CoB-CoMヘテロジスルフィドレダクターゼ CoB-CoMヘテロジスルフィドレダクターゼ分子量: 21706.963 Da / 分子数: 2 / 由来タイプ: 天然 / 由来: (天然)   Methanospirillum hungatei JF-1 (古細菌) / 参照: UniProt: Q2FKZ3 Methanospirillum hungatei JF-1 (古細菌) / 参照: UniProt: Q2FKZ3#3: タンパク質 |  CoB-CoMヘテロジスルフィドレダクターゼ CoB-CoMヘテロジスルフィドレダクターゼ分子量: 32930.938 Da / 分子数: 2 / 由来タイプ: 天然 / 由来: (天然)   Methanospirillum hungatei JF-1 (古細菌) / 参照: UniProt: Q2FKZ2 Methanospirillum hungatei JF-1 (古細菌) / 参照: UniProt: Q2FKZ2 |
|---|
-非ポリマー , 4種, 26分子 

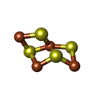




| #7: 化合物 | ChemComp-SF4 /  鉄・硫黄クラスター 鉄・硫黄クラスター#8: 化合物 |  フラビンアデニンジヌクレオチド フラビンアデニンジヌクレオチド#9: 化合物 | ChemComp-9S8 / #10: 化合物 | ChemComp-FES / |  鉄・硫黄クラスター 鉄・硫黄クラスター |
|---|
-詳細
| 研究の焦点であるリガンドがあるか | Y |
|---|
-実験情報
-実験
| 実験 | 手法:  電子顕微鏡法 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法:  単粒子再構成法 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: Dimeric formate dehydrogenase - heterodisulfide reductase - formylmethanofuran dehydrogenase complex タイプ: COMPLEX / Entity ID: #1-#6 / 由来: NATURAL |
|---|---|
| 分子量 | 値: 0.948 MDa / 実験値: NO |
| 由来(天然) | 生物種:   Methanospirillum hungatei JF-1 (古細菌) / 細胞内の位置: cytoplasm Methanospirillum hungatei JF-1 (古細菌) / 細胞内の位置: cytoplasm |
| 緩衝液 | pH: 7.6 |
| 緩衝液成分 | 濃度: 25 mM 名称: Tris-HCl  トリスヒドロキシメチルアミノメタン トリスヒドロキシメチルアミノメタン式: Tris-HCl  トリスヒドロキシメチルアミノメタン トリスヒドロキシメチルアミノメタン |
| 試料 | 濃度: 0.5 mg/ml / 包埋: NO / シャドウイング: NO / 染色 : NO / 凍結 : NO / 凍結 : YES : YES詳細: Preparation in an anaerobic tent (O2 < 20 ppm at all times, nearly always < 2ppm) |
急速凍結 | 装置: HOMEMADE PLUNGER / 凍結剤: ETHANE / 湿度: 70 % / 凍結前の試料温度: 293 K |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS |
| 電子銃 | 電子線源 : :  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD Bright-field microscopy / Cs Bright-field microscopy / Cs : 2.7 mm / C2レンズ絞り径: 70 µm / アライメント法: COMA FREE : 2.7 mm / C2レンズ絞り径: 70 µm / アライメント法: COMA FREE |
| 試料ホルダ | 凍結剤: NITROGEN 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 撮影 | 電子線照射量: 50 e/Å2 フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 実像数: 8745 |
| 電子光学装置 | エネルギーフィルター名称 : GIF Bioquantum / エネルギーフィルタースリット幅: 20 eV : GIF Bioquantum / エネルギーフィルタースリット幅: 20 eV |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| EMソフトウェア |
| ||||||||||||||||||||||||||||||||||||
| 画像処理 | 詳細: Recorded in counted mode | ||||||||||||||||||||||||||||||||||||
CTF補正 | タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION | ||||||||||||||||||||||||||||||||||||
| 対称性 | 点対称性 : C1 (非対称) : C1 (非対称) | ||||||||||||||||||||||||||||||||||||
3次元再構成 | 解像度: 3 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 277450 詳細: This applies to the map of the mobile arm in state1. The uploaded map is a composite map. クラス平均像の数: 1 / 対称性のタイプ: POINT | ||||||||||||||||||||||||||||||||||||
| 原子モデル構築 | プロトコル: OTHER | ||||||||||||||||||||||||||||||||||||
| 精密化 | 交差検証法: THROUGHOUT | ||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso max: 62.27 Å2 / Biso mean: 31.7842 Å2 / Biso min: 5.51 Å2 | ||||||||||||||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー































 PDBj
PDBj

















