+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 6l4t | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Structure of the peripheral FCPI from diatom | ||||||
 要素 要素 | (Fucoxanthin chlorophyll a/c-binding protein ...) x 10 | ||||||
 キーワード キーワード |  ELECTRON TRANSPORT (電子伝達系) / ELECTRON TRANSPORT (電子伝達系) /  Photosystem I (光化学系I) Photosystem I (光化学系I) | ||||||
| 機能・相同性 | Chem-A86 /  クロロフィルa / Chem-DD6 / クロロフィルa / Chem-DD6 /  Chlorophyll c1 / 1,2-DIPALMITOYL-PHOSPHATIDYL-GLYCEROLE / 1,2-DISTEAROYL-MONOGALACTOSYL-DIGLYCERIDE Chlorophyll c1 / 1,2-DIPALMITOYL-PHOSPHATIDYL-GLYCEROLE / 1,2-DISTEAROYL-MONOGALACTOSYL-DIGLYCERIDE 機能・相同性情報 機能・相同性情報 | ||||||
| 生物種 |  Chaetoceros gracilis (珪藻) Chaetoceros gracilis (珪藻) | ||||||
| 手法 |  電子顕微鏡法 / 電子顕微鏡法 /  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 2.6 Å クライオ電子顕微鏡法 / 解像度: 2.6 Å | ||||||
 データ登録者 データ登録者 | Nagao, R. / Kato, K. / Miyazaki, N. / Akita, F. / Shen, J.R. | ||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2020 ジャーナル: Nat Commun / 年: 2020タイトル: Structural basis for assembly and function of a diatom photosystem I-light-harvesting supercomplex. 著者: Ryo Nagao / Koji Kato / Kentaro Ifuku / Takehiro Suzuki / Minoru Kumazawa / Ikuo Uchiyama / Yasuhiro Kashino / Naoshi Dohmae / Seiji Akimoto / Jian-Ren Shen / Naoyuki Miyazaki / Fusamichi Akita /  要旨: Photosynthetic light-harvesting complexes (LHCs) play a pivotal role in collecting solar energy for photochemical reactions in photosynthesis. One of the major LHCs are fucoxanthin chlorophyll a/c- ...Photosynthetic light-harvesting complexes (LHCs) play a pivotal role in collecting solar energy for photochemical reactions in photosynthesis. One of the major LHCs are fucoxanthin chlorophyll a/c-binding proteins (FCPs) present in diatoms, a group of organisms having important contribution to the global carbon cycle. Here, we report a 2.40-Å resolution structure of the diatom photosystem I (PSI)-FCPI supercomplex by cryo-electron microscopy. The supercomplex is composed of 16 different FCPI subunits surrounding a monomeric PSI core. Each FCPI subunit showed different protein structures with different pigment contents and binding sites, and they form a complicated pigment-protein network together with the PSI core to harvest and transfer the light energy efficiently. In addition, two unique, previously unidentified subunits were found in the PSI core. The structure provides numerous insights into not only the light-harvesting strategy in diatom PSI-FCPI but also evolutionary dynamics of light harvesters among oxyphototrophs. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  6l4t.cif.gz 6l4t.cif.gz | 555.1 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb6l4t.ent.gz pdb6l4t.ent.gz | 表示 |  PDB形式 PDB形式 | |
| PDBx/mmJSON形式 |  6l4t.json.gz 6l4t.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/l4/6l4t https://data.pdbj.org/pub/pdb/validation_reports/l4/6l4t ftp://data.pdbj.org/pub/pdb/validation_reports/l4/6l4t ftp://data.pdbj.org/pub/pdb/validation_reports/l4/6l4t | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
-Fucoxanthin chlorophyll a/c-binding protein ... , 10種, 10分子 67810111213141516
| #1: タンパク質 | 分子量: 22362.646 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  Chaetoceros gracilis (珪藻) Chaetoceros gracilis (珪藻) |
|---|---|
| #2: タンパク質 | 分子量: 32238.152 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  Chaetoceros gracilis (珪藻) Chaetoceros gracilis (珪藻) |
| #3: タンパク質 | 分子量: 29704.150 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  Chaetoceros gracilis (珪藻) Chaetoceros gracilis (珪藻) |
| #4: タンパク質 | 分子量: 22216.549 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  Chaetoceros gracilis (珪藻) Chaetoceros gracilis (珪藻) |
| #5: タンパク質 | 分子量: 24842.496 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  Chaetoceros gracilis (珪藻) Chaetoceros gracilis (珪藻) |
| #6: タンパク質 | 分子量: 21318.404 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  Chaetoceros gracilis (珪藻) Chaetoceros gracilis (珪藻) |
| #7: タンパク質 | 分子量: 25994.562 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  Chaetoceros gracilis (珪藻) Chaetoceros gracilis (珪藻) |
| #8: タンパク質 | 分子量: 26875.781 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  Chaetoceros gracilis (珪藻) Chaetoceros gracilis (珪藻) |
| #9: タンパク質 | 分子量: 30545.674 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  Chaetoceros gracilis (珪藻) Chaetoceros gracilis (珪藻) |
| #10: タンパク質 | 分子量: 23533.191 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  Chaetoceros gracilis (珪藻) Chaetoceros gracilis (珪藻) |
-糖 , 1種, 13分子 
| #17: 糖 | ChemComp-LMT / |
|---|
-非ポリマー , 7種, 191分子 
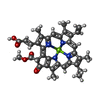

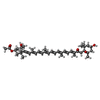









| #11: 化合物 | ChemComp-CLA /  クロロフィルa クロロフィルa#12: 化合物 | ChemComp-KC1 /  Chlorophyll c Chlorophyll c#13: 化合物 | ChemComp-DD6 / (  ジアジノキサンチン ジアジノキサンチン#14: 化合物 | ChemComp-A86 / (  フコキサンチン フコキサンチン#15: 化合物 | ChemComp-LHG / |  Phosphatidylglycerol Phosphatidylglycerol#16: 化合物 | ChemComp-LMG / #18: 水 | ChemComp-HOH / |  水 水 |
|---|
-詳細
| 研究の焦点であるリガンドがあるか | N |
|---|
-実験情報
-実験
| 実験 | 手法:  電子顕微鏡法 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法:  単粒子再構成法 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: peripheral FCPI / タイプ: COMPLEX / Entity ID: #1-#10 / 由来: NATURAL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 分子量 | 値: 0.17 MDa / 実験値: NO | |||||||||||||||
| 由来(天然) | 生物種:  Chaetoceros gracilis (珪藻) Chaetoceros gracilis (珪藻) | |||||||||||||||
| 緩衝液 | pH: 6.5 | |||||||||||||||
| 緩衝液成分 |
| |||||||||||||||
| 試料 | 濃度: 2 mg/ml / 包埋: NO / シャドウイング: NO / 染色 : NO / 凍結 : NO / 凍結 : YES : YES | |||||||||||||||
| 試料支持 | グリッドの材料: MOLYBDENUM / グリッドのサイズ: 300 divisions/in. / グリッドのタイプ: C-flat-1.2/1.3 | |||||||||||||||
急速凍結 | 装置: FEI VITROBOT MARK IV / 凍結剤: ETHANE / 湿度: 100 % / 凍結前の試料温度: 277 K |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS |
| 電子銃 | 電子線源 : :  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy |
| 撮影 | 電子線照射量: 50 e/Å2 / 検出モード: COUNTING フィルム・検出器のモデル: FEI FALCON III (4k x 4k) |
- 解析
解析
| EMソフトウェア |
| ||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
CTF補正 | タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION | ||||||||||||||||||||||||||||||||
| 粒子像の選択 | 選択した粒子像数: 2689965 | ||||||||||||||||||||||||||||||||
| 対称性 | 点対称性 : C1 (非対称) : C1 (非対称) | ||||||||||||||||||||||||||||||||
3次元再構成 | 解像度: 2.6 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 470801 / 対称性のタイプ: POINT | ||||||||||||||||||||||||||||||||
| 原子モデル構築 | プロトコル: FLEXIBLE FIT / 空間: REAL |
 ムービー
ムービー コントローラー
コントローラー













 PDBj
PDBj











