+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-8739 | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
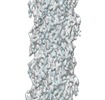
| タイトル | Cryo-EM reconstruction of Neisseria gonorrhoeae Type IV pilus | |||||||||||||||
 マップデータ マップデータ | Neisseria gonorrhoeae Type IV pilus | |||||||||||||||
 試料 試料 |
| |||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 | |||||||||||||||
| 生物種 |   Neisseria gonorrhoeae (淋菌) Neisseria gonorrhoeae (淋菌) | |||||||||||||||
| 手法 | らせん対称体再構成法 /  クライオ電子顕微鏡法 / 解像度: 5.1 Å クライオ電子顕微鏡法 / 解像度: 5.1 Å | |||||||||||||||
 データ登録者 データ登録者 | Wang F / Orlova A / Altindal T / Craig L / Egelman EH | |||||||||||||||
| 資金援助 |  米国, 米国,  フランス, フランス,  カナダ, 4件 カナダ, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Structure / 年: 2017 ジャーナル: Structure / 年: 2017タイトル: Cryoelectron Microscopy Reconstructions of the Pseudomonas aeruginosa and Neisseria gonorrhoeae Type IV Pili at Sub-nanometer Resolution. 著者: Fengbin Wang / Mathieu Coureuil / Tomasz Osinski / Albina Orlova / Tuba Altindal / Gaël Gesbert / Xavier Nassif / Edward H Egelman / Lisa Craig /    要旨: We report here cryoelectron microscopy reconstructions of type IV pili (T4P) from two important human pathogens, Pseudomonas aeruginosa and Neisseria gonorrhoeae, at ∼ 8 and 5 Å resolution, ...We report here cryoelectron microscopy reconstructions of type IV pili (T4P) from two important human pathogens, Pseudomonas aeruginosa and Neisseria gonorrhoeae, at ∼ 8 and 5 Å resolution, respectively. The two structures reveal distinct arrangements of the pilin globular domains on the pilus surfaces, which impart different helical parameters, but similar packing of the conserved N-terminal α helices, α1, in the filament core. In contrast to the continuous α helix seen in the X-ray crystal structures of the P. aeruginosa and N. gonorrhoeae pilin subunits, α1 in the pilus filaments has a melted segment located between conserved helix-breaking residues Gly14 and Pro22, as seen for the Neisseria meningitidis T4P. Using mutagenesis we show that Pro22 is critical for pilus assembly, as are Thr2 and Glu5, which are positioned to interact in the hydrophobic filament core. These structures provide a framework for understanding T4P assembly, function, and biophysical properties. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_8739.map.gz emd_8739.map.gz | 3.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-8739-v30.xml emd-8739-v30.xml emd-8739.xml emd-8739.xml | 12.8 KB 12.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_8739.png emd_8739.png | 162.1 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-8739 http://ftp.pdbj.org/pub/emdb/structures/EMD-8739 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8739 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8739 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_8739.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_8739.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Neisseria gonorrhoeae Type IV pilus | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.05 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Neisseria gonorrhoeae Type IV pilin filament
| 全体 | 名称: Neisseria gonorrhoeae Type IV pilin filament |
|---|---|
| 要素 |
|
-超分子 #1: Neisseria gonorrhoeae Type IV pilin filament
| 超分子 | 名称: Neisseria gonorrhoeae Type IV pilin filament / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:   Neisseria gonorrhoeae (淋菌) / 株: C30 Neisseria gonorrhoeae (淋菌) / 株: C30 |
-分子 #1: Fimbrial protein
| 分子 | 名称: Fimbrial protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 21 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Neisseria gonorrhoeae (淋菌) / 株: C30 Neisseria gonorrhoeae (淋菌) / 株: C30 |
| 分子量 | 理論値: 17.196467 KDa |
| 配列 | 文字列: FTLIELMIVI AIVGILAAVA LPAYQDYTAR AQVSEAILLA EGQKSAVTEY YLNHGKWPEN NTSAGVASSP TDIKGKYVKE VEVKNGVVT ATMLSSGVNN EIKGKKLSLW ARRENGSVKW FCGQPVTRTD DDTVADAKDG KEIDTKHLPS TCRDNFDAK |
-分子 #3: PHOSPHORIC ACID MONO-(2-AMINO-ETHYL) ESTER
| 分子 | 名称: PHOSPHORIC ACID MONO-(2-AMINO-ETHYL) ESTER / タイプ: ligand / ID: 3 / コピー数: 21 / 式: OPE |
|---|---|
| 分子量 | 理論値: 141.063 Da |
| Chemical component information | 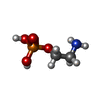 ChemComp-OPE: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 濃度 | 0.1 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 / 詳細: PBS buffer |
| グリッド | 前処理 - タイプ: PLASMA CLEANING / 詳細: unspecified |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 90 % / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy |
| 撮影 | フィルム・検出器のモデル: FEI FALCON II (4k x 4k) 検出モード: INTEGRATING / 平均露光時間: 2.0 sec. / 平均電子線量: 20.0 e/Å2 詳細: Images were stored containing seven parts, where each part represented a set of frames corresponding to a dose of ~20 electrons per Angstrom^2. The full dose image stack was used for the ...詳細: Images were stored containing seven parts, where each part represented a set of frames corresponding to a dose of ~20 electrons per Angstrom^2. The full dose image stack was used for the estimation of the CTF as well as for boxing filaments. Only the first two parts were used for the reconstruction (~5 electrons per Angstrom^2). |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| CTF補正 | ソフトウェア - 名称: CTFFIND3 |
|---|---|
| 初期モデル | モデルのタイプ: NONE / 詳細: featureless cylinder |
| 最終 角度割当 | タイプ: NOT APPLICABLE / ソフトウェア - 名称: SPIDER |
| 最終 再構成 | 想定した対称性 - らせんパラメータ - Δz: 10.1 Å 想定した対称性 - らせんパラメータ - ΔΦ: 100.8 ° 想定した対称性 - らせんパラメータ - 軸対称性: C1 (非対称) 解像度のタイプ: BY AUTHOR / 解像度: 5.1 Å / 解像度の算出法: OTHER / ソフトウェア - 名称: SPIDER / 詳細: model-map FSC 0.38 cut-off / 使用した粒子像数: 9855 |
-原子モデル構築 1
| 精密化 | 空間: REAL |
|---|---|
| 得られたモデル |  PDB-5vxx: |
 ムービー
ムービー コントローラー
コントローラー