+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-6455 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | TRPV2 ion channel from rabbit | |||||||||
 マップデータ マップデータ | TRPV2, sharpened with a B factor of -76 using RELION postprocess | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  TRP channel (TRPチャネル) / TRP channel (TRPチャネル) /  ion transport ion transport | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報growth cone membrane / response to temperature stimulus / positive regulation of calcium ion import / positive regulation of axon extension / axonal growth cone /  calcium channel activity / positive regulation of cold-induced thermogenesis / calcium channel activity / positive regulation of cold-induced thermogenesis /  cell body / cell body /  細胞膜 / identical protein binding 細胞膜 / identical protein binding類似検索 - 分子機能 | |||||||||
| 生物種 |   Oryctolagus cuniculus (ウサギ) Oryctolagus cuniculus (ウサギ) | |||||||||
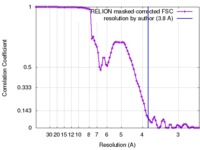
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.8 Å クライオ電子顕微鏡法 / 解像度: 3.8 Å | |||||||||
 データ登録者 データ登録者 | Herzik MA / Zubcevic L / Chung BC / Lander GC / Lee SY | |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2016 ジャーナル: Nat Struct Mol Biol / 年: 2016タイトル: Cryo-electron microscopy structure of the TRPV2 ion channel. 著者: Lejla Zubcevic / Mark A Herzik / Ben C Chung / Zhirui Liu / Gabriel C Lander / Seok-Yong Lee /  要旨: Transient receptor potential vanilloid (TRPV) cation channels are polymodal sensors involved in a variety of physiological processes. TRPV2, a member of the TRPV family, is regulated by temperature, ...Transient receptor potential vanilloid (TRPV) cation channels are polymodal sensors involved in a variety of physiological processes. TRPV2, a member of the TRPV family, is regulated by temperature, by ligands, such as probenecid and cannabinoids, and by lipids. TRPV2 has been implicated in many biological functions, including somatosensation, osmosensation and innate immunity. Here we present the atomic model of rabbit TRPV2 in its putative desensitized state, as determined by cryo-EM at a nominal resolution of ∼4 Å. In the TRPV2 structure, the transmembrane segment 6 (S6), which is involved in gate opening, adopts a conformation different from the one observed in TRPV1. Structural comparisons of TRPV1 and TRPV2 indicate that a rotation of the ankyrin-repeat domain is coupled to pore opening via the TRP domain, and this pore opening can be modulated by rearrangements in the secondary structure of S6. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_6455.map.gz emd_6455.map.gz | 14.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-6455-v30.xml emd-6455-v30.xml emd-6455.xml emd-6455.xml | 10.1 KB 10.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_6455_fsc.xml emd_6455_fsc.xml | 9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  400_6455.gif 400_6455.gif 80_6455.gif 80_6455.gif | 59.5 KB 4.2 KB | ||
| その他 |  recls4_run1_half1_class001_unfil.map.gz recls4_run1_half1_class001_unfil.map.gz recls4_run1_half2_class001_unfil.map.gz recls4_run1_half2_class001_unfil.map.gz unsharpened.map.gz unsharpened.map.gz | 45.9 MB 45.9 MB 45.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-6455 http://ftp.pdbj.org/pub/emdb/structures/EMD-6455 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6455 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6455 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_6455.map.gz / 形式: CCP4 / 大きさ: 15.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_6455.map.gz / 形式: CCP4 / 大きさ: 15.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | TRPV2, sharpened with a B factor of -76 using RELION postprocess | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.31 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-添付マップデータ: recls4 run1 half1 class001 unfil.map
| ファイル | recls4_run1_half1_class001_unfil.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
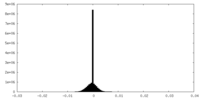
| 密度ヒストグラム |
-添付マップデータ: recls4 run1 half2 class001 unfil.map
| ファイル | recls4_run1_half2_class001_unfil.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
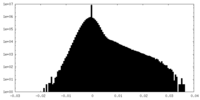
| 密度ヒストグラム |
-添付マップデータ: unsharpened.map
| ファイル | unsharpened.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
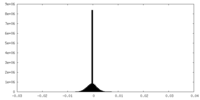
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Rabbit TRPV2
| 全体 | 名称: Rabbit TRPV2 |
|---|---|
| 要素 |
|
-超分子 #1000: Rabbit TRPV2
| 超分子 | 名称: Rabbit TRPV2 / タイプ: sample / ID: 1000 / 集合状態: homotetramer / Number unique components: 1 |
|---|
-分子 #1: TRPV2
| 分子 | 名称: TRPV2 / タイプ: protein_or_peptide / ID: 1 / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:   Oryctolagus cuniculus (ウサギ) / 別称: Rabbit Oryctolagus cuniculus (ウサギ) / 別称: Rabbit |
| 組換発現 | 生物種:   Spodoptera frugiperda (ツマジロクサヨトウ) Spodoptera frugiperda (ツマジロクサヨトウ)組換細胞: SF9 / 組換プラスミド: pFastBac |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.0 mg/mL |
|---|---|
| 緩衝液 | pH: 7.6 / 詳細: PBS |
| グリッド | 詳細: 400 mesh C-flat copper grid with carbon support, 1.2 micron holes spaced 1.3 microns |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 120 K / 装置: GATAN CRYOPLUNGE 3 / 手法: Blot for 2.5 seconds before plunging. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 38168 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.2 µm / 倍率(公称値): 22500 Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.2 µm / 倍率(公称値): 22500 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 温度 | 最低: 77 K / 最高: 79 K / 平均: 78 K |
| アライメント法 | Legacy - 非点収差: Objective astigmatism was corrected at 22500 times magnification using Thon rings visualized with a K2 camera. |
| 詳細 | Data was acquired using Leginon and collected in K2 super-resolution mode. |
| 日付 | 2015年5月12日 |
| 撮影 | カテゴリ: CCD / フィルム・検出器のモデル: GATAN K2 (4k x 4k) / 実像数: 747 / 平均電子線量: 57 e/Å2 / 詳細: 50 frames, 200 ms per frame |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー














 Z
Z Y
Y X
X