+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-4551 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Near Atomic Structure of an Atadenovirus Shows a possible gene duplication event and Intergenera Variations in Cementing Proteins | |||||||||
 マップデータ マップデータ | Cryo-EM structure of a lizard atadenovirus, LAdV-2, at 3.4 A resolution | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | adenovirus atadenovirus virus evolution /  VIRUS (ウイルス) VIRUS (ウイルス) | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報hexon binding /  viral capsid, decoration / T=25 icosahedral viral capsid / microtubule-dependent intracellular transport of viral material towards nucleus / endocytosis involved in viral entry into host cell / viral capsid, decoration / T=25 icosahedral viral capsid / microtubule-dependent intracellular transport of viral material towards nucleus / endocytosis involved in viral entry into host cell /  カプシド / symbiont entry into host cell / host cell nucleus / virion attachment to host cell / structural molecule activity / カプシド / symbiont entry into host cell / host cell nucleus / virion attachment to host cell / structural molecule activity /  細胞核 細胞核類似検索 - 分子機能 | |||||||||
| 生物種 |  Lizard adenovirus 2 (ウイルス) Lizard adenovirus 2 (ウイルス) | |||||||||
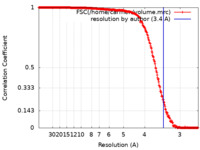
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.4 Å クライオ電子顕微鏡法 / 解像度: 3.4 Å | |||||||||
 データ登録者 データ登録者 | Condezo GN / Marabini R | |||||||||
 引用 引用 |  ジャーナル: Sci Adv / 年: 2021 ジャーナル: Sci Adv / 年: 2021タイトル: Near-atomic structure of an atadenovirus reveals a conserved capsid-binding motif and intergenera variations in cementing proteins. 著者: Roberto Marabini / Gabriela N Condezo / Mart Krupovic / Rosa Menéndez-Conejero / Josué Gómez-Blanco / Carmen San Martín /   要旨: Of five known adenovirus genera, high-resolution structures are available only for mammalian-infecting mastadenoviruses. We present the first high-resolution structure of an adenovirus with ...Of five known adenovirus genera, high-resolution structures are available only for mammalian-infecting mastadenoviruses. We present the first high-resolution structure of an adenovirus with nonmammalian host: lizard atadenovirus LAdV-2. We find a large conformational difference in the internal vertex protein IIIa between mast- and atadenoviruses, induced by the presence of an extended polypeptide. This polypeptide, and α-helical clusters beneath the facet, likely correspond to genus-specific proteins LH2 and p32k. Another genus-specific protein, LH3, with a fold typical of bacteriophage tailspikes, contacts the capsid surface via a triskelion structure identical to that used by mastadenovirus protein IX, revealing a conserved capsid-binding motif and an ancient gene duplication event. Our data also suggest that mastadenovirus E1B-55 K was exapted from the atadenovirus-like LH3 protein. This work provides new information on the evolution of adenoviruses, emphasizing the importance of minor coat proteins for determining specific physicochemical properties of virions and most likely their tropism. #1:  ジャーナル: Biorxiv / 年: 2020 ジャーナル: Biorxiv / 年: 2020タイトル: Near Atomic Structure of an Atadenovirus Reveals a Conserved Capsid-Binding Motif and Intergenera Variations in Cementing Proteins 著者: Marabini R / Condezo GN / Gomez-Blanco J / San Martin C | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_4551.map.gz emd_4551.map.gz | 1.6 GB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-4551-v30.xml emd-4551-v30.xml emd-4551.xml emd-4551.xml | 17.1 KB 17.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_4551_fsc.xml emd_4551_fsc.xml | 29.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_4551.png emd_4551.png | 314.4 KB | ||
| Filedesc metadata |  emd-4551.cif.gz emd-4551.cif.gz | 7.3 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-4551 http://ftp.pdbj.org/pub/emdb/structures/EMD-4551 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4551 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4551 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_4551.map.gz / 形式: CCP4 / 大きさ: 1.8 GB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_4551.map.gz / 形式: CCP4 / 大きさ: 1.8 GB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM structure of a lizard atadenovirus, LAdV-2, at 3.4 A resolution | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.35 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Lizard adenovirus 2
| 全体 | 名称:  Lizard adenovirus 2 (ウイルス) Lizard adenovirus 2 (ウイルス) |
|---|---|
| 要素 |
|
-超分子 #1: Lizard adenovirus 2
| 超分子 | 名称: Lizard adenovirus 2 / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: all / NCBI-ID: 874272 / 生物種: Lizard adenovirus 2 / ウイルスタイプ: VIRION / ウイルス・単離状態: SPECIES / ウイルス・エンベロープ: No / ウイルス・中空状態: No |
|---|---|
| 宿主 | 生物種:   Heloderma horridum (メキシコドクトカゲ) Heloderma horridum (メキシコドクトカゲ) |
| 分子量 | 理論値: 150 MDa |
| ウイルス殻 | Shell ID: 1 / 直径: 940.0 Å / T番号(三角分割数): 25 |
-分子 #1: Hexon protein
| 分子 | 名称: Hexon protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 12 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Lizard adenovirus 2 (ウイルス) Lizard adenovirus 2 (ウイルス) |
| 分子量 | 理論値: 101.86193 KDa |
| 配列 | 文字列: MEPQREFFHI AGRSAKEYLS ENLVQFIQAT QNYFNIGEKF RDPYVAPSAG VTTDRSQKLQ LRVVPIQTED NVNYYKARFT LNVGDNRLV DLGSSYFDIK GTLDRGPSFK PYGGTAYNPL APKSAPINSA FTVGNDTHFV AQLPQTYAAG GTGVTEAIQQ Q VSGVDPNP ...文字列: MEPQREFFHI AGRSAKEYLS ENLVQFIQAT QNYFNIGEKF RDPYVAPSAG VTTDRSQKLQ LRVVPIQTED NVNYYKARFT LNVGDNRLV DLGSSYFDIK GTLDRGPSFK PYGGTAYNPL APKSAPINSA FTVGNDTHFV AQLPQTYAAG GTGVTEAIQQ Q VSGVDPNP QVGQPNYAGP VVVNTTNNAG LGRIVSADSE GQQFPCYGAY APPQSAGGDV STAAVTKTYI NTTNNNGRVS GT MATDTIT WENPDAHFAD FVDDRRATAA GNRPNYIGFR DNFIGMMYYN SGSNTGSFSS QTQQLNIVLD LNDRNSELSY QYL LADLTS RWHYFALWNQ AVDDYDHHVR ILENDGYEEG PPNLAFPPHV ISNPFAPAAV GTGMTVNEQQ QTAAVTANTV ALIG YGNIP AVEMNLPANL KRTFLYSNVA MYLPDTYKFT PANVDLPENH LSYGYINGRL PLPNIVDTWT DIGARWSLDV MDTVN PFNH HRNTGLKYRS QLLGNGRYCD FHIQVPQKFF AIKNLLLLPG TYNYEWYFRK DPNMVLQSTL GNDLRADGAS ITYTQI NLY VSFFPMNYDT QSELELMLRN ATNDQNFSDY LGAVNNLYQI PAGSSTVVVN IPDRSWGAFR GWSFTRLKVS ETPRIGA TQ DPNFQYSGSI PYLDGTFYLS HTFQRCSIQW DSSVPWPGND RMLTPNWFEI KRPINQDAEG NDTMQSNLTK DFFMVQMA A SYNQGYQGFN WPNCTKHYGF INNFEPMSRQ VPEYGANYPN LMAAYLANPQ TMPIWNNCGF QQKTATNVLL ERCGHPYVA NWPYPLSGRN AVPNQVTERK FLVDRYLWQI PFSSNFLNMG TLTDLGQNVM YANSSHSLNM QFTVDPMTEP TYLMLLFGVF DQVVINQPT RSGISVAYLR LPFASGSAAT UniProtKB:  Hexon protein Hexon protein |
-分子 #2: Protein LH3
| 分子 | 名称: Protein LH3 / タイプ: protein_or_peptide / ID: 2 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Lizard adenovirus 2 (ウイルス) Lizard adenovirus 2 (ウイルス) |
| 分子量 | 理論値: 41.158344 KDa |
| 配列 | 文字列: MTSVEELYVI NPINQWPAPG SFSSQKPPGT LLPGEDPEAV FKQYHVVYLV PGAQYHWKNI LIEKPVWIYG NGATVRTSGT GPILRIVGN RTEKRDVRIQ DISFFGEDCT PNRMEPMSEK LVYQMAIWVT DMKRVTIKGC NFTNFAGAAV FFEETAYNGF F WSMQHLIT ...文字列: MTSVEELYVI NPINQWPAPG SFSSQKPPGT LLPGEDPEAV FKQYHVVYLV PGAQYHWKNI LIEKPVWIYG NGATVRTSGT GPILRIVGN RTEKRDVRIQ DISFFGEDCT PNRMEPMSEK LVYQMAIWVT DMKRVTIKGC NFTNFAGAAV FFEETAYNGF F WSMQHLIT ECRFTGCRIG IANGGRSEYS TASFNNFFDC QICFNVVGGN WNRCGNIAAN CRCVYLHTTN MWYEGAGGNF NA AHGSFTG NTMNHCDYGG NLWPTAFQLP DREIQLAGFY FDNARARCPT WTGNTQYYGD MKILNFNQAN DAAIFVIDGC ALY GQPGDT GSIETTAALT DKVFIQGCQG NKVTLFNIKA ANVVPAIGTI KQKP UniProtKB: Protein LH3 |
-分子 #3: Pre-hexon-linking protein VIII
| 分子 | 名称: Pre-hexon-linking protein VIII / タイプ: protein_or_peptide / ID: 3 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Lizard adenovirus 2 (ウイルス) Lizard adenovirus 2 (ウイルス) |
| 分子量 | 理論値: 30.744453 KDa |
| 配列 | 文字列: MDAPVTPYIW QYQPQTGKAA GARQNYGAVI NWLSADNNMF HRVQTVNRAR NLIDEIREET VRPDLAASFN DWTYDQLTQP PGTAYLPAP DPLTGPTTIR DKVLSAEGEQ LAGSRPSVLH GGASLPPSAY SLGDGREYMK LTRDALPFPQ NWMVKENGVW T PIVEQRAA ...文字列: MDAPVTPYIW QYQPQTGKAA GARQNYGAVI NWLSADNNMF HRVQTVNRAR NLIDEIREET VRPDLAASFN DWTYDQLTQP PGTAYLPAP DPLTGPTTIR DKVLSAEGEQ LAGSRPSVLH GGASLPPSAY SLGDGREYMK LTRDALPFPQ NWMVKENGVW T PIVEQRAA LRGGSANALS SYPTLLFNQP PILRYRRPGQ QLQGSGVIAP SSKVLSLLSE APRIPRTEGM TPYQFANSFP PV VYEDPFS QNLAVFPKEF SPLFEPENQV LASSLATLQY N UniProtKB: Pre-hexon-linking protein VIII |
-分子 #4: PIIIa
| 分子 | 名称: PIIIa / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Lizard adenovirus 2 (ウイルス) Lizard adenovirus 2 (ウイルス) |
| 分子量 | 理論値: 67.049266 KDa |
| 配列 | 文字列: MDPGLKPSSL WTHKIIDSII ANRSLSAVQN FRKQPLANKL TALEDAIVQP RKDTTPETVA AILQELVAMG ALQPNEVGPM FSDLMIRVH KYNSTNVQNN LSVLLGDIRA AQSEAIRSTN VGELSNQVVL NDFLSREPAV VPQGQHNYEA FKQTLRLMVN E APNVTLFK ...文字列: MDPGLKPSSL WTHKIIDSII ANRSLSAVQN FRKQPLANKL TALEDAIVQP RKDTTPETVA AILQELVAMG ALQPNEVGPM FSDLMIRVH KYNSTNVQNN LSVLLGDIRA AQSEAIRSTN VGELSNQVVL NDFLSREPAV VPQGQHNYEA FKQTLRLMVN E APNVTLFK SGPDTLMQVN IRGVNTVNLN SAFKNLKNFW GVQLDTEIVP GSISSKLSSN TRVLLLFLAP FTNDSTFTPD TF ISQIMRL YRETVAASIE QPQETELEVA ETIRELGGDV EDIGRTMAFL LKNKEEIVSN PRTLSPRQLN VLRYVQESLQ DRI DRNGEE PEDALRNLVF SFAPSYFEAN GPFIRRLISY LEVALINSPN YFREIYSNKY WTPPASFWTQ NYGDFHLERE AEAE RRAAS EAGYGDFEEG DFALPDNLGE GSDLAWDDFN TAMSPSVPPT PSVRSAPASL SYGRTSPSSV SSLTASDRNI GATLA RAVI PPAAAAIGSA AGEALYPSLG QYLAPAASLA ATRLLNLTRA RRQRLKRDSL ARHRRITEVR GVYPKAAPTR SSSSSS VSS TPTVFEPLPG AFSVINPLMR PEGDRDVSGT GIVNPFSHLK PRNGLQ UniProtKB: PIIIa |
-分子 #5: Penton protein
| 分子 | 名称: Penton protein / タイプ: protein_or_peptide / ID: 5 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Lizard adenovirus 2 (ウイルス) Lizard adenovirus 2 (ウイルス) |
| 分子量 | 理論値: 50.619848 KDa |
| 配列 | 文字列: MEVYVPPPRV MAPTEGRNSI SYNPIAPLQD TTHIYIIDNK TSDIENLNIH KDHSNFYTNI VQNVDVAPSD AATQTIKLDE RSRWGGELH TILKTNAPNV TEFFNSNSFK ALLMSDKTDP ANPVYTWFEL SIPEGDYTVG SLIDMLNNAV VENYLEVGRQ K GVQISDIG ...文字列: MEVYVPPPRV MAPTEGRNSI SYNPIAPLQD TTHIYIIDNK TSDIENLNIH KDHSNFYTNI VQNVDVAPSD AATQTIKLDE RSRWGGELH TILKTNAPNV TEFFNSNSFK ALLMSDKTDP ANPVYTWFEL SIPEGDYTVG SLIDMLNNAV VENYLEVGRQ K GVQISDIG VKFDTRNFSL GRDPLTSLVT PGNYTFKAFH PDIVLLPGCG VDFTHSRINN MLGMRKRFPY EPGYVITYED LV GGNIPAL LDLAKYPGET SPVLQDPDGN SYHVEEVSPK KWQTKYRSWC LAYNSSQGTL KSEQILTVPD ITGGLGQLYW SLP DAFKPP VTFTNNTTDI STQPVTGMHL FPLSQRIVYN TSAVYAQLVE QMTNNTKVFN RFPKNAILMQ PPYDTTQWIS ENVP YVADH GIQPLKNSLT GVQRVTLTDD RRRSCPYIYK TLATVTPKVL SSATLQ UniProtKB:  Penton protein Penton protein |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 / 詳細: PBS |
|---|---|
| グリッド | モデル: Quantifoil R2/4 / 材質: COPPER/RHODIUM |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy |
| 撮影 | フィルム・検出器のモデル: FEI FALCON II (4k x 4k) 検出モード: INTEGRATING / 平均露光時間: 2.0 sec. / 平均電子線量: 54.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー