+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of ADP-bound and phosphorylated Pediculus humanus (Ph) PINK1 dimer | |||||||||
 マップデータ マップデータ | Map (sharpened) of the ADP-bound phosphorylated PhPINK1 dimer | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  PINK1 / PINK1 /  Kinase (キナーゼ) / Kinase (キナーゼ) /  Mitophagy (マイトファジー) / Mitophagy (マイトファジー) /  Parkinson's Disease (パーキンソン病) / Parkinson's Disease (パーキンソン病) /  Ubiquitin (ユビキチン) / Ubiquitin (ユビキチン) /  Phosphorylation (リン酸化) / Phospho-ubiquitin / Phosphorylation (リン酸化) / Phospho-ubiquitin /  TRANSFERASE (転移酵素) TRANSFERASE (転移酵素) | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報positive regulation of mitochondrial fission /  オートファジー / regulation of apoptotic process / mitochondrial outer membrane / オートファジー / regulation of apoptotic process / mitochondrial outer membrane /  ミトコンドリア内膜 / ミトコンドリア内膜 /  non-specific serine/threonine protein kinase / non-specific serine/threonine protein kinase /  protein kinase activity / protein kinase activity /  リン酸化 / protein serine/threonine kinase activity / リン酸化 / protein serine/threonine kinase activity /  ATP binding ...positive regulation of mitochondrial fission / ATP binding ...positive regulation of mitochondrial fission /  オートファジー / regulation of apoptotic process / mitochondrial outer membrane / オートファジー / regulation of apoptotic process / mitochondrial outer membrane /  ミトコンドリア内膜 / ミトコンドリア内膜 /  non-specific serine/threonine protein kinase / non-specific serine/threonine protein kinase /  protein kinase activity / protein kinase activity /  リン酸化 / protein serine/threonine kinase activity / リン酸化 / protein serine/threonine kinase activity /  ATP binding / ATP binding /  metal ion binding / metal ion binding /  細胞質基質 細胞質基質類似検索 - 分子機能 | |||||||||
| 生物種 |   Pediculus humanus corporis (キモノジラミ) Pediculus humanus corporis (キモノジラミ) | |||||||||
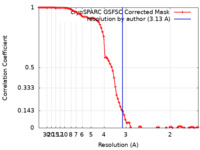
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.13 Å クライオ電子顕微鏡法 / 解像度: 3.13 Å | |||||||||
 データ登録者 データ登録者 | Gan ZY / Kirk NS / Leis A / Komander D | |||||||||
| 資金援助 |  オーストラリア, オーストラリア,  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Sci Adv / 年: 2024 ジャーナル: Sci Adv / 年: 2024タイトル: Interaction of PINK1 with nucleotides and kinetin. 著者: Zhong Yan Gan / Sylvie Callegari / Thanh N Nguyen / Nicholas S Kirk / Andrew Leis / Michael Lazarou / Grant Dewson / David Komander /  要旨: The ubiquitin kinase PINK1 accumulates on damaged mitochondria to trigger mitophagy, and PINK1 loss-of-function mutations cause early onset Parkinson's disease. Nucleotide analogs such as kinetin ...The ubiquitin kinase PINK1 accumulates on damaged mitochondria to trigger mitophagy, and PINK1 loss-of-function mutations cause early onset Parkinson's disease. Nucleotide analogs such as kinetin triphosphate (KTP) were reported to enhance PINK1 activity and may represent a therapeutic strategy for the treatment of Parkinson's disease. Here, we investigate the interaction of PINK1 with nucleotides, including KTP. We establish a cryo-EM platform exploiting the dodecamer assembly of () PINK1 and determine PINK1 structures bound to AMP-PNP and ADP, revealing conformational changes in the kinase N-lobe that help establish PINK1's ubiquitin binding site. Notably, we find that KTP is unable to bind PINK1 or human () PINK1 due to a steric clash with the kinase "gatekeeper" methionine residue, and mutation to Ala or Gly is required for PINK1 to bind and use KTP as a phosphate donor in ubiquitin phosphorylation and mitophagy. PINK1 M318G can be used to conditionally uncouple PINK1 stabilization and activity on mitochondria. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_42807.map.gz emd_42807.map.gz | 59.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-42807-v30.xml emd-42807-v30.xml emd-42807.xml emd-42807.xml | 18.8 KB 18.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_42807_fsc.xml emd_42807_fsc.xml | 8.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_42807.png emd_42807.png | 74.1 KB | ||
| Filedesc metadata |  emd-42807.cif.gz emd-42807.cif.gz | 6.3 KB | ||
| その他 |  emd_42807_half_map_1.map.gz emd_42807_half_map_1.map.gz emd_42807_half_map_2.map.gz emd_42807_half_map_2.map.gz | 59.5 MB 59.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-42807 http://ftp.pdbj.org/pub/emdb/structures/EMD-42807 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-42807 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-42807 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_42807.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_42807.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Map (sharpened) of the ADP-bound phosphorylated PhPINK1 dimer | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.808 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: Half map of the ADP-bound phosphorylated PhPINK1 dimer
| ファイル | emd_42807_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map of the ADP-bound phosphorylated PhPINK1 dimer | ||||||||||||
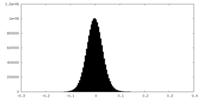
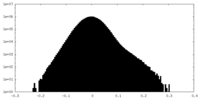
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map of the ADP-bound phosphorylated PhPINK1 dimer
| ファイル | emd_42807_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map of the ADP-bound phosphorylated PhPINK1 dimer | ||||||||||||
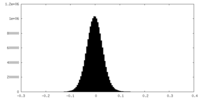
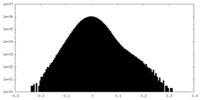
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : ADP-bound and phosphorylated Pediculus humanus (Ph) PINK1 dodecamer
| 全体 | 名称: ADP-bound and phosphorylated Pediculus humanus (Ph) PINK1 dodecamer |
|---|---|
| 要素 |
|
-超分子 #1: ADP-bound and phosphorylated Pediculus humanus (Ph) PINK1 dodecamer
| 超分子 | 名称: ADP-bound and phosphorylated Pediculus humanus (Ph) PINK1 dodecamer タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:   Pediculus humanus corporis (キモノジラミ) Pediculus humanus corporis (キモノジラミ) |
-分子 #1: Serine/threonine-protein kinase Pink1, mitochondrial
| 分子 | 名称: Serine/threonine-protein kinase Pink1, mitochondrial タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Pediculus humanus corporis (キモノジラミ) Pediculus humanus corporis (キモノジラミ) |
| 分子量 | 理論値: 53.133582 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: GPSGLLTKDD ELEGICWEIR EAVSKGKWND SESENVEQLQ AANLDELDLG EPIAKGCNAV VYSAKLKNVQ SNKLAHQLAV KMMFNYDVE (SEP)NSTAILKAM YRETVPAMSY FFNQNLFNIE NISDFKIRLP PHPNIVRMYS VFADRIPDLQ CNKQLYP EA ...文字列: GPSGLLTKDD ELEGICWEIR EAVSKGKWND SESENVEQLQ AANLDELDLG EPIAKGCNAV VYSAKLKNVQ SNKLAHQLAV KMMFNYDVE (SEP)NSTAILKAM YRETVPAMSY FFNQNLFNIE NISDFKIRLP PHPNIVRMYS VFADRIPDLQ CNKQLYP EA LPPRINPEGS GRNMSLFLVM KRYDCTLKEY LRDK(TPO)PNMRS SILLLSQLLE AVAHMNIHNI SHRDLKSDNI LVDL SEGDA YPTIVITDFG CCLCDKQNGL VIPYRSEDQD KGGNRALMAP EIANAKPGTF SWLNYKKSDL WAVGAIAYEI FNIDN PFYD KTMKLLSKSY KEEDLPELPD TIPFIIRNLV SNMLSRSTNK RLDCDVAATV AQLYLWAPSS WLKENYTLPN SNEIIQ WLL CLSSKVLCER DITARNKTNT MSESVSKAQY KGRRSLPEYE LIASFLRRVR LHLVRKGLKW IQELHIYN UniProtKB: Serine/threonine-protein kinase Pink1, mitochondrial |
-分子 #2: ADENOSINE-5'-DIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-DIPHOSPHATE / タイプ: ligand / ID: 2 / コピー数: 2 / 式: ADP |
|---|---|
| 分子量 | 理論値: 427.201 Da |
| Chemical component information |  ChemComp-ADP: |
-分子 #3: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 3 / コピー数: 4 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.9 mg/mL | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8.5 構成要素:
| ||||||||||||||||||
| グリッド | モデル: UltrAuFoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: GOLD / 支持フィルム - トポロジー: HOLEY ARRAY / 前処理 - 時間: 120 sec. / 前処理 - 雰囲気: AIR | ||||||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: OTHER / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 1.5 µm / 最小 デフォーカス(公称値): 0.5 µm Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 1.5 µm / 最小 デフォーカス(公称値): 0.5 µm |
| 特殊光学系 | エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) 平均電子線量: 50.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー









 Z
Z Y
Y X
X