[日本語] English
 万見
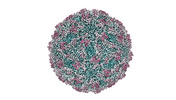
万見- EMDB-41125: Zophobas morio black wasting virus strain NJ2-molitor virion structure -
+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Zophobas morio black wasting virus strain NJ2-molitor virion structure | |||||||||
 マップデータ マップデータ | Main map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  Capsid (カプシド) / Capsid (カプシド) /  Virion (ウイルス) / Virion (ウイルス) /  Parvovirus (パルボウイルス) / Parvovirus (パルボウイルス) /  Densovirus / Densovirus /  Invertebrate (無脊椎動物) / Invertebrate (無脊椎動物) /  Insect (昆虫) / Insect (昆虫) /  Pathogen (病原体) / Pathogen (病原体) /  ssDNA (デオキシリボ核酸) / ssDNA (デオキシリボ核酸) /  VIRUS (ウイルス) / VIRUS-DNA complex VIRUS (ウイルス) / VIRUS-DNA complex | |||||||||
| 生物種 |  Zophobas morio black wasting virus (ウイルス) / Zophobas morio black wasting virus (ウイルス) /  Zophobas morio densovirus (ウイルス) Zophobas morio densovirus (ウイルス) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 2.0 Å クライオ電子顕微鏡法 / 解像度: 2.0 Å | |||||||||
 データ登録者 データ登録者 | Penzes JJ / Kaelber JT | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: Sequencing-free discovery by cryo-EM of a pathogenic parvovirus causing mass mortality of farmed beetles 著者: Penzes JJ / Holm M / Firlar E / Kaelber JT | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_41125.map.gz emd_41125.map.gz | 2.6 GB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-41125-v30.xml emd-41125-v30.xml emd-41125.xml emd-41125.xml | 19.3 KB 19.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_41125.png emd_41125.png | 82 KB | ||
| Filedesc metadata |  emd-41125.cif.gz emd-41125.cif.gz | 6.2 KB | ||
| その他 |  emd_41125_half_map_1.map.gz emd_41125_half_map_1.map.gz emd_41125_half_map_2.map.gz emd_41125_half_map_2.map.gz | 2.5 GB 2.5 GB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-41125 http://ftp.pdbj.org/pub/emdb/structures/EMD-41125 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41125 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41125 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_41125.map.gz / 形式: CCP4 / 大きさ: 2.7 GB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_41125.map.gz / 形式: CCP4 / 大きさ: 2.7 GB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Main map | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.63 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: Half map of D 1000275531 em-volume P1.map.V3
| ファイル | emd_41125_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map of D_1000275531_em-volume_P1.map.V3 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
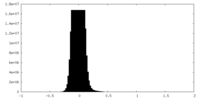
| 密度ヒストグラム |
-ハーフマップ: Half map of D 1000275531 em-volume P1.map.V3
| ファイル | emd_41125_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map of D_1000275531_em-volume_P1.map.V3 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
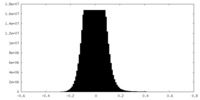
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Zophobas morio densovirus
| 全体 | 名称:  Zophobas morio densovirus (ウイルス) Zophobas morio densovirus (ウイルス) |
|---|---|
| 要素 |
|
-超分子 #1: Zophobas morio densovirus
| 超分子 | 名称: Zophobas morio densovirus / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 詳細: Purified from T. molitor larvae, which were asymptomatic NCBI-ID: 2750924 / 生物種: Zophobas morio densovirus / Sci species strain: NJ2-molitor / ウイルスタイプ: VIRION / ウイルス・単離状態: SPECIES / ウイルス・エンベロープ: No / ウイルス・中空状態: No |
|---|---|
| 宿主 | 生物種:   Tenebrio molitor (チャイロコメノゴミムシダマシ) Tenebrio molitor (チャイロコメノゴミムシダマシ) |
| ウイルス殻 | Shell ID: 1 / 直径: 28.0 Å / T番号(三角分割数): 1 |
-分子 #1: Major capsid protein
| 分子 | 名称: Major capsid protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Zophobas morio black wasting virus (ウイルス) Zophobas morio black wasting virus (ウイルス) |
| 分子量 | 理論値: 48.085629 KDa |
| 配列 | 文字列: ATAILRPIGL HVEKFQQTYR KKWRFLTSAN ANVILAEAAS GERPARWALT TGMASIPWEY LFFYMSPAEY NRMKNYPGTF AKSASVRIR TWNTRVAFQT GDTQTANATL NQNKFLQVAK GIRSIPFICS TNRKYTYSDT EPMQPTGFAT LTSYEYRDGL K IAMYGYDN ...文字列: ATAILRPIGL HVEKFQQTYR KKWRFLTSAN ANVILAEAAS GERPARWALT TGMASIPWEY LFFYMSPAEY NRMKNYPGTF AKSASVRIR TWNTRVAFQT GDTQTANATL NQNKFLQVAK GIRSIPFICS TNRKYTYSDT EPMQPTGFAT LTSYEYRDGL K IAMYGYDN DSADFAKKPP ADATGAEIYL QDYLTIYTND ARATTGTKIL AGFPPYKNFI EEFDASACIN TDVVAMDYDF SY APLVPQF APVPNNLITQ NYNASYPAGT KNEVTAVKTT DSSQATPPTQ VRNAPRKYIQ GPNADTTFFD EEQNYLRVPI EQG GIFEEV NVETVHDTQM PSINVGIRAV PKLTTIDETT QANSWLDAQG YFEVDCVLTT ESVDPYTYIK GGCYSANTKS QLQY FASDG RPIAKVYDNP NVYGRMQMIK TVKP |
-分子 #2: DNA (5'-D(P*CP*GP*A)-3')
| 分子 | 名称: DNA (5'-D(P*CP*GP*A)-3') / タイプ: dna / ID: 2 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  Zophobas morio black wasting virus (ウイルス) Zophobas morio black wasting virus (ウイルス) |
| 分子量 | 理論値: 886.637 Da |
| 配列 | 文字列: (DC)(DG)(DA) |
-分子 #3: DNA (5'-D(P*CP*AP*GP*GP*CP*CP*AP*AP*A)-3')
| 分子 | 名称: DNA (5'-D(P*CP*AP*GP*GP*CP*CP*AP*AP*A)-3') / タイプ: dna / ID: 3 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  Zophobas morio black wasting virus (ウイルス) Zophobas morio black wasting virus (ウイルス) |
| 分子量 | 理論値: 2.733827 KDa |
| 配列 | 文字列: (DC)(DA)(DG)(DG)(DC)(DC)(DA)(DA)(DA) |
-分子 #4: DNA (5'-D(P*TP*CP*GP*AP*A)-3')
| 分子 | 名称: DNA (5'-D(P*TP*CP*GP*AP*A)-3') / タイプ: dna / ID: 4 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  Zophobas morio black wasting virus (ウイルス) Zophobas morio black wasting virus (ウイルス) |
| 分子量 | 理論値: 1.504037 KDa |
| 配列 | 文字列: (DT)(DC)(DG)(DA)(DA) |
-分子 #5: water
| 分子 | 名称: water / タイプ: ligand / ID: 5 / コピー数: 80 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 / 構成要素 - 濃度: 1.0 x / 構成要素 - 式: PBS / 構成要素 - 名称: Phosphate-buffered saline |
| グリッド | モデル: UltrAuFoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: GOLD / 支持フィルム - トポロジー: HOLEY / 支持フィルム - Film thickness: 20 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 300 sec. / 前処理 - 雰囲気: AIR |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 293 K / 装置: FEI VITROBOT MARK IV |
| 詳細 | Purified virus from homogenized T. molitor larval tissue |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 1.2 µm / 最小 デフォーカス(公称値): 0.5 µm Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 1.2 µm / 最小 デフォーカス(公称値): 0.5 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 撮影したグリッド数: 1 / 平均露光時間: 3.0 sec. / 平均電子線量: 32.0 e/Å2 |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
- 画像解析
画像解析
| 粒子像選択 | 選択した数: 156329 |
|---|---|
| 初期モデル | モデルのタイプ: INSILICO MODEL / In silico モデル: Ab-initio 3D model |
| 初期 角度割当 | タイプ: ANGULAR RECONSTITUTION / ソフトウェア - 名称: cryoSPARC |
| 最終 3次元分類 | クラス数: 50 / ソフトウェア - 名称: cryoSPARC |
| 最終 角度割当 | タイプ: ANGULAR RECONSTITUTION / ソフトウェア - 名称: cryoSPARC |
| 最終 再構成 | 想定した対称性 - 点群: I (正20面体型対称 ) / 解像度のタイプ: BY AUTHOR / 解像度: 2.0 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: cryoSPARC / 使用した粒子像数: 15964 ) / 解像度のタイプ: BY AUTHOR / 解像度: 2.0 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: cryoSPARC / 使用した粒子像数: 15964 |
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL |
|---|---|
| 得られたモデル |  PDB-8t9x: |
 ムービー
ムービー コントローラー
コントローラー










 Z
Z Y
Y X
X