+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
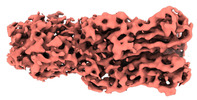
| タイトル | Cyanophage Pam3 portal-adaptor | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 | Cyanophage Pam3 != uncultured cyanophage Cyanophage Pam3
| |||||||||
| 生物種 |  uncultured cyanophage (ファージ) uncultured cyanophage (ファージ) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.57 Å クライオ電子顕微鏡法 / 解像度: 3.57 Å | |||||||||
 データ登録者 データ登録者 | Yang F / Jiang YL / Zhou CZ | |||||||||
| 資金援助 |  中国, 1件 中国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2023 ジャーナル: Proc Natl Acad Sci U S A / 年: 2023タイトル: Fine structure and assembly pattern of a minimal myophage Pam3. 著者: Feng Yang / Yong-Liang Jiang / Jun-Tao Zhang / Jie Zhu / Kang Du / Rong-Cheng Yu / Zi-Lu Wei / Wen-Wen Kong / Ning Cui / Wei-Fang Li / Yuxing Chen / Qiong Li / Cong-Zhao Zhou /  要旨: The myophage possesses a contractile tail that penetrates its host cell envelope. Except for investigations on the bacteriophage T4 with a rather complicated structure, the assembly pattern and tail ...The myophage possesses a contractile tail that penetrates its host cell envelope. Except for investigations on the bacteriophage T4 with a rather complicated structure, the assembly pattern and tail contraction mechanism of myophage remain largely unknown. Here, we present the fine structure of a freshwater cyanophage Pam3, which has an icosahedral capsid of ~680 Å in diameter, connected via a three-section neck to an 840-Å-long contractile tail, ending with a three-module baseplate composed of only six protein components. This simplified baseplate consists of a central hub-spike surrounded by six wedge heterotriplexes, to which twelve tail fibers are covalently attached via disulfide bonds in alternating upward and downward configurations. In vitro reduction assays revealed a putative redox-dependent mechanism of baseplate assembly and tail sheath contraction. These findings establish a minimal myophage that might become a user-friendly chassis phage in synthetic biology. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_34679.map.gz emd_34679.map.gz | 69.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-34679-v30.xml emd-34679-v30.xml emd-34679.xml emd-34679.xml | 15.1 KB 15.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_34679.png emd_34679.png | 132.7 KB | ||
| その他 |  emd_34679_half_map_1.map.gz emd_34679_half_map_1.map.gz emd_34679_half_map_2.map.gz emd_34679_half_map_2.map.gz | 75.9 MB 75.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-34679 http://ftp.pdbj.org/pub/emdb/structures/EMD-34679 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-34679 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-34679 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_34679.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_34679.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.013 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_34679_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
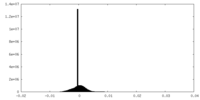
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_34679_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
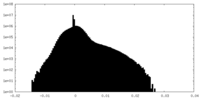
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Cyanophage Pam3
| 全体 | 名称: Cyanophage Pam3 |
|---|---|
| 要素 |
|
-超分子 #1: uncultured cyanophage
| 超分子 | 名称: uncultured cyanophage / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: all / NCBI-ID: 215796 / 生物種: uncultured cyanophage / ウイルスタイプ: VIRION / ウイルス・単離状態: OTHER / ウイルス・エンベロープ: No / ウイルス・中空状態: No |
|---|---|
| 宿主 | 生物種:  Pseudanabaena mucicola (バクテリア) Pseudanabaena mucicola (バクテリア) |
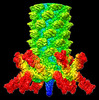
-分子 #1: Pam3 portal protein
| 分子 | 名称: Pam3 portal protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 12 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  uncultured cyanophage (ファージ) uncultured cyanophage (ファージ) |
| 分子量 | 理論値: 69.800445 KDa |
| 配列 | 文字列: MSDKLPTADS LRSMISGLGD PLRDKNASTF HWDRQLDDRQ LLYAYRNSWV ARKAVTIPAL DAVRKWRDWQ ADQKDISRIE ATEKRLGLQ QKLLQCKTLA RLWGGAAIVI GVKDQDMATP FEPETVNKDD LVYLTVMSRR ELSPEELEQD PLDEFYARPK R YQVSNGRN ...文字列: MSDKLPTADS LRSMISGLGD PLRDKNASTF HWDRQLDDRQ LLYAYRNSWV ARKAVTIPAL DAVRKWRDWQ ADQKDISRIE ATEKRLGLQ QKLLQCKTLA RLWGGAAIVI GVKDQDMATP FEPETVNKDD LVYLTVMSRR ELSPEELEQD PLDEFYARPK R YQVSNGRN LSFVHPSRIV HQVGETHPDP MLATGVNVGW GDSTLQALYD AMMNSDNTQA NIASLVFEAN VDVIGLPDFM EN MSSEVYR QKLLDRFTLA AAGKGINKTL MLDAEEVFTA HSRSFANLDK IMEQFILFVA GAADIPLTRF LGQSPAGMSS TGQ HDMKNY HDRIQSIQTL DLQPSMYRLD EAIIRSSLGA RPEELFYIWS PLEQMSEKER AEIGKLHAET VNVIAGTGLF MQEE LREVF GNQLVETGLY PGLGDLLAQN GNELPEWDLE QRSAEASTKT AEAAALAAET ASRQPARAVT DATPRPLYMR RDVVN GDEI LRHYREQGVE GLYAAEELHV TIAYSKKPLD WMKLGEPWSA KLEVAPGGPR VHELFGDDSS VLVLCFAASE LNWRAQ QVI DAGGSSDHGE YQSHISLSLQ GRPEQIETLR PYQGRIVLGP ELFEQIEEGD WREKVKTD |
-分子 #2: Pam3 adaptor protein
| 分子 | 名称: Pam3 adaptor protein / タイプ: protein_or_peptide / ID: 2 / コピー数: 12 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  uncultured cyanophage (ファージ) uncultured cyanophage (ファージ) |
| 分子量 | 理論値: 13.871434 KDa |
| 配列 | 文字列: MNPIPAASDL KTRYPEFTGV SDAVVNAIIA EVNGMVDDGW EVSDQKPAVL ALAAHMLSRE GYPGRATNPN SFDPTNRPIL SRKVGDVST TFGRTDGGAA EGGANSYNYS STVYGQTFLR LLRLNAPAVG LV |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 300 K |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.0 µm Bright-field microscopy / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 50.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期 角度割当 | タイプ: RANDOM ASSIGNMENT |
|---|---|
| 最終 角度割当 | タイプ: RANDOM ASSIGNMENT |
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.57 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: RELION (ver. 3.1) / 使用した粒子像数: 126613 |
 ムービー
ムービー コントローラー
コントローラー













 Z
Z Y
Y X
X