+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-3329 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo electron microscopy of a complex of Tor and Lst8 | |||||||||
 マップデータ マップデータ | volume for dimer of Tor/Lst8 heterodimers | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  cryo-EM (低温電子顕微鏡法) / Tor / Lst8 / cryo-EM (低温電子顕微鏡法) / Tor / Lst8 /  mTOR (MTOR) / mTOR (MTOR) /  kinase (キナーゼ) / PIKK / S/T protein kinase / kinase (キナーゼ) / PIKK / S/T protein kinase /  TORC1 / TORC1 /  mTORC1 (MTORC1) mTORC1 (MTORC1) | |||||||||
| 機能・相同性 |  TORC1 complex / TORC1 complex /  : / : /  : :  機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |   Kluyveromyces marxianus (酵母) Kluyveromyces marxianus (酵母) | |||||||||
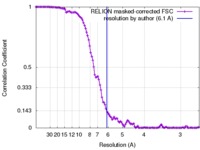
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 6.1 Å クライオ電子顕微鏡法 / 解像度: 6.1 Å | |||||||||
 データ登録者 データ登録者 | Baretic D / Berndt A / Ohashi Y / Johnson CM / Williams RL | |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2016 ジャーナル: Nat Commun / 年: 2016タイトル: Tor forms a dimer through an N-terminal helical solenoid with a complex topology. 著者: Domagoj Baretić / Alex Berndt / Yohei Ohashi / Christopher M Johnson / Roger L Williams /  要旨: The target of rapamycin (Tor) is a Ser/Thr protein kinase that regulates a range of anabolic and catabolic processes. Tor is present in two complexes, TORC1 and TORC2, in which the Tor-Lst8 ...The target of rapamycin (Tor) is a Ser/Thr protein kinase that regulates a range of anabolic and catabolic processes. Tor is present in two complexes, TORC1 and TORC2, in which the Tor-Lst8 heterodimer forms a common sub-complex. We have determined the cryo-electron microscopy (EM) structure of Tor bound to Lst8. Two Tor-Lst8 heterodimers assemble further into a dyad-symmetry dimer mediated by Tor-Tor interactions. The first 1,300 residues of Tor form a HEAT repeat-containing α-solenoid with four distinct segments: a highly curved 800-residue N-terminal 'spiral', followed by a 400-residue low-curvature 'bridge' and an extended 'railing' running along the bridge leading to the 'cap' that links to FAT region. This complex topology was verified by domain insertions and offers a new interpretation of the mTORC1 structure. The spiral of one TOR interacts with the bridge of another, which together form a joint platform for the Regulatory Associated Protein of TOR (RAPTOR) regulatory subunit. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_3329.map.gz emd_3329.map.gz | 116.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-3329-v30.xml emd-3329-v30.xml emd-3329.xml emd-3329.xml | 11.2 KB 11.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_3329_fsc.xml emd_3329_fsc.xml | 11 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  EMD-3329_image.png EMD-3329_image.png | 69 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-3329 http://ftp.pdbj.org/pub/emdb/structures/EMD-3329 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3329 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3329 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_3329.map.gz / 形式: CCP4 / 大きさ: 122.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_3329.map.gz / 形式: CCP4 / 大きさ: 122.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | volume for dimer of Tor/Lst8 heterodimers | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.33 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Complex of Tor1 with Lst8
| 全体 | 名称: Complex of Tor1 with Lst8 |
|---|---|
| 要素 |
|
-超分子 #1000: Complex of Tor1 with Lst8
| 超分子 | 名称: Complex of Tor1 with Lst8 / タイプ: sample / ID: 1000 / 集合状態: Dimer of Tor1/Lst8 heterodimers / Number unique components: 2 |
|---|---|
| 分子量 | 理論値: 622 KDa |
-分子 #1: Target of rapamycin (Tor)
| 分子 | 名称: Target of rapamycin (Tor) / タイプ: protein_or_peptide / ID: 1 / Name.synonym: Tor1 / コピー数: 2 / 集合状態: dimer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:   Kluyveromyces marxianus (酵母) Kluyveromyces marxianus (酵母) |
| 分子量 | 理論値: 277 KDa |
| 組換発現 | 生物種:   Kluyveromyces marxianus (酵母) Kluyveromyces marxianus (酵母) |
| 配列 | GO:  TORC1 complex TORC1 complex |
-分子 #2: Lst8
| 分子 | 名称: Lst8 / タイプ: protein_or_peptide / ID: 2 / Name.synonym: Lethal with SEC13 protein 8 / コピー数: 2 / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:   Kluyveromyces marxianus (酵母) Kluyveromyces marxianus (酵母) |
| 分子量 | 理論値: 34 KDa |
| 組換発現 | 生物種:   Kluyveromyces marxianus (酵母) Kluyveromyces marxianus (酵母) |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2.5 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 詳細: 50 mM Hepes pH 7.4 (23 deg C), 75 mM KCl, 250 mM NaCl, 0.3 % (v/v) CHAPS, 1 mM TCEP |
| グリッド | 詳細: Quantifoil Au R 0.6/1.0 or Au R 1.2/1.3, 300 mesh grids, blotted for 11-13 s at 4 deg C |
| 凍結 | 凍結剤: ETHANE / 装置: OTHER / 手法: 11-13 s at 4 deg C |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 105263 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.5 µm / 最小 デフォーカス(公称値): 2.5 µm / 倍率(公称値): 47000 Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.5 µm / 最小 デフォーカス(公称値): 2.5 µm / 倍率(公称値): 47000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 日付 | 2015年5月16日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: FEI FALCON II (4k x 4k) 実像数: 2332 / 平均電子線量: 40 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - #0 - Chain ID: A / Chain - #1 - Chain ID: B |
|---|---|
| ソフトウェア | 名称:  Chimera Chimera |
| 詳細 | An initial model for the FATKIN was derived from 4JSV. A poly-ALA model was built into the remaining density (about half of the total). The full model was refined by Phenix real space, global minimization, 2-fold NCS, one B-factor per residue, secondary structure rstraints, ramachandran restraints |
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT / 当てはまり具合の基準: correlation |
| 得られたモデル |  PDB-5fvm: |
-原子モデル構築 2
| 初期モデル | PDB ID: Chain - #0 - Chain ID: C / Chain - #1 - Chain ID: D |
|---|---|
| ソフトウェア | 名称:  Chimera Chimera |
| 詳細 | An initial model for the Lst8 was derived from 4JSV. A poly-ALA model was adjusted to the density. The full model was refined by Phenix real space, global minimization, 2-fold NCS, one B-factor per residue, secondary structure rstraints, ramachandran restraints |
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT / 当てはまり具合の基準: correlation |
| 得られたモデル |  PDB-5fvm: |
 ムービー
ムービー コントローラー
コントローラー