+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
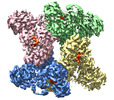
| タイトル | Trichodesmium erythraeum cyanophycin synthetase 1 (TeCphA1) with ATPgammaS, 4x(beta-Asp-Arg), and aspartate | |||||||||
 マップデータ マップデータ | Map without b-factor sharpening | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 cyanophycin synthase (L-aspartate-adding) / cyanophycin synthase (L-aspartate-adding) /  cyanophycin synthase (L-arginine-adding) / cyanophycin synthetase activity (L-aspartate-adding) / cyanophycin synthetase activity (L-arginine-adding) / cyanophycin synthase (L-arginine-adding) / cyanophycin synthetase activity (L-aspartate-adding) / cyanophycin synthetase activity (L-arginine-adding) /  tetrahydrofolylpolyglutamate synthase activity / macromolecule biosynthetic process / tetrahydrofolylpolyglutamate synthase activity / macromolecule biosynthetic process /  ATP binding / ATP binding /  metal ion binding metal ion binding類似検索 - 分子機能 | |||||||||
| 生物種 |   Trichodesmium erythraeum IMS101 (バクテリア) / Synthetic construct (人工物) Trichodesmium erythraeum IMS101 (バクテリア) / Synthetic construct (人工物) | |||||||||
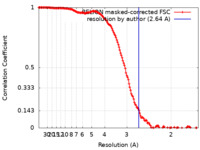
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 2.64 Å クライオ電子顕微鏡法 / 解像度: 2.64 Å | |||||||||
 データ登録者 データ登録者 | Miyakawa T / Yang J / Kawasaki M / Adachi N / Fujii A / Miyauchi Y / Muramatsu T / Moriya T / Senda T / Tanokura M | |||||||||
| 資金援助 |  日本, 2件 日本, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2022 ジャーナル: Nat Commun / 年: 2022タイトル: Structural bases for aspartate recognition and polymerization efficiency of cyanobacterial cyanophycin synthetase. 著者: Takuya Miyakawa / Jian Yang / Masato Kawasaki / Naruhiko Adachi / Ayumu Fujii / Yumiko Miyauchi / Tomonari Muramatsu / Toshio Moriya / Toshiya Senda / Masaru Tanokura /   要旨: Cyanophycin is a natural biopolymer consisting of equimolar amounts of aspartate and arginine as the backbone and branched sidechain, respectively. It is produced by a single enzyme, cyanophycin ...Cyanophycin is a natural biopolymer consisting of equimolar amounts of aspartate and arginine as the backbone and branched sidechain, respectively. It is produced by a single enzyme, cyanophycin synthetase (CphA1), and accumulates as a nitrogen reservoir during N fixation by most cyanobacteria. A recent structural study showed that three constituent domains of CphA1 function as two distinct catalytic sites and an oligomerization interface in cyanophycin synthesis. However, it remains unclear how the ATP-dependent addition of aspartate to cyanophycin is initiated at the catalytic site of the glutathione synthetase-like domain. Here, we report the cryogenic electron microscopy structures of CphA1, including a complex with aspartate, cyanophycin primer peptide, and ATP analog. These structures reveal the aspartate binding mode and phosphate-binding loop movement to the active site required for the reaction. Furthermore, structural and mutational data show a potential role of protein dynamics in the catalytic efficiency of the arginine condensation reaction. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_32383.map.gz emd_32383.map.gz | 256.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-32383-v30.xml emd-32383-v30.xml emd-32383.xml emd-32383.xml | 25.3 KB 25.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_32383_fsc.xml emd_32383_fsc.xml | 14.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_32383.png emd_32383.png | 194.4 KB | ||
| マスクデータ |  emd_32383_msk_1.map emd_32383_msk_1.map | 274.6 MB |  マスクマップ マスクマップ | |
| その他 |  emd_32383_additional_1.map.gz emd_32383_additional_1.map.gz emd_32383_half_map_1.map.gz emd_32383_half_map_1.map.gz emd_32383_half_map_2.map.gz emd_32383_half_map_2.map.gz | 257.5 MB 219 MB 218.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-32383 http://ftp.pdbj.org/pub/emdb/structures/EMD-32383 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-32383 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-32383 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_32383.map.gz / 形式: CCP4 / 大きさ: 274.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_32383.map.gz / 形式: CCP4 / 大きさ: 274.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Map without b-factor sharpening | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.83 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_32383_msk_1.map emd_32383_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Map with b-factor sharpening
| ファイル | emd_32383_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Map with b-factor sharpening | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_32383_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_32383_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : TeCphA1 with ATPgammaS, 4x(beta-Asp-Arg) and aspartate
| 全体 | 名称: TeCphA1 with ATPgammaS, 4x(beta-Asp-Arg) and aspartate |
|---|---|
| 要素 |
|
-超分子 #1: TeCphA1 with ATPgammaS, 4x(beta-Asp-Arg) and aspartate
| 超分子 | 名称: TeCphA1 with ATPgammaS, 4x(beta-Asp-Arg) and aspartate タイプ: complex / キメラ: Yes / ID: 1 / 親要素: 0 / 含まれる分子: #1 / 詳細: Homotetramer |
|---|---|
| 由来(天然) | 生物種:   Trichodesmium erythraeum IMS101 (バクテリア) Trichodesmium erythraeum IMS101 (バクテリア) |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) / 組換細胞: BL21(DE3) / 組換プラスミド: pET-22b(+) Escherichia coli (大腸菌) / 組換細胞: BL21(DE3) / 組換プラスミド: pET-22b(+) |
| 分子量 | 理論値: 400 KDa |
-分子 #1: Cyanophycin synthase
| 分子 | 名称: Cyanophycin synthase / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO / EC番号:  cyanophycin synthase (L-aspartate-adding) cyanophycin synthase (L-aspartate-adding) |
|---|---|
| 由来(天然) | 生物種:   Trichodesmium erythraeum IMS101 (バクテリア) Trichodesmium erythraeum IMS101 (バクテリア)株: IMS101 |
| 分子量 | 理論値: 99.079914 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: MKILKLQTLR GPNYWSIHRH KLVVMRLDLE DLYEKYTSDI PGFYKGLTEV LPSLVEHLCS PGVKGGFLTR VEKGTLIGHV IEHVAIELQ ELAGMPVGFG RTRETSTTGV FQVVIEYENE QAGRYAARAA VRLCQSIVDT GTYPATELQQ DLEDLKELKN Q ASLGPSTE ...文字列: MKILKLQTLR GPNYWSIHRH KLVVMRLDLE DLYEKYTSDI PGFYKGLTEV LPSLVEHLCS PGVKGGFLTR VEKGTLIGHV IEHVAIELQ ELAGMPVGFG RTRETSTTGV FQVVIEYENE QAGRYAARAA VRLCQSIVDT GTYPATELQQ DLEDLKELKN Q ASLGPSTE AIVKEAEARG IPWTQLGARF MIQFGYGVNQ KKIQATLSNQ TGILGVELAC DKEGTKRILK DAGVPVPRGT VA RYFDELQ DAIEYVGGYP IVIKPLDGNH GRGITIDVKN WQEAEEAYDL ARKASKTKTV IVERYYTGKD HRVLVVNGKV VAV AERVPA HVVGNGKSTI AELIEETNRD PQRGDGHDNI LTRITVDKSA LDILGKQGYS IDSIPLKGKK CFLRATANLS TGGI AVDRT DEIHPENVWL LSRVAKIIGL DIAGIDVVTE DISQPLREVE GVIVEVNAAP GFRMHVAPSR GLARNVAGAV MDMLF PGSK NGRIPILSVT GTNGKTTTTR LLAHIIKQTG KVVGYTTTDG TYIGEYLAET GDNTGPQSAH LILSDPTVEV AVLETA RGG ILRSGLGFSS CEVGIVLNVT ADHLGIGDID TIEQLAKLKS VVAESVMPKG YAVLNAEDPL VAAMADRVKG QVAYFSM DP NNELLLRHTE AGGLAAIYEN GYISILKGDW TLRIEKAVNV PITMAGKAPF MIANALAACL AVFTQGVKIE HIRKGLST F VASVDQTPGR MNMFNMGSYH ALVDYAHNPA SYEALGGFVR NWPGKRIGVV GGPGDRRDED FVSLGELAAD IFDEIIIKE DDDTRGRPRG NAAELICQGV KQFLNGIKNS ESKATYESIL DETAAINTAL DRAPIDGLVV ILPESVNRAI SLIEGRHVIQ DIELLQDSQ REPKDQEVLK SSILEHHHHH H |
-分子 #2: 4x(beta-Asp-Arg)
| 分子 | 名称: 4x(beta-Asp-Arg) / タイプ: protein_or_peptide / ID: 2 詳細: Backbone peptide bonds are formed between Asp residues. An Arg residue forms a peptide bond with the beta-carboxy group of Asp. コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: Synthetic construct (人工物) |
| 分子量 | 理論値: 478.366 Da |
| 配列 | 文字列: DDDD |
-分子 #3: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 3 / コピー数: 11 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-分子 #4: PHOSPHOTHIOPHOSPHORIC ACID-ADENYLATE ESTER
| 分子 | 名称: PHOSPHOTHIOPHOSPHORIC ACID-ADENYLATE ESTER / タイプ: ligand / ID: 4 / コピー数: 8 / 式: AGS |
|---|---|
| 分子量 | 理論値: 523.247 Da |
| Chemical component information |  ChemComp-AGS: |
-分子 #5: ASPARTIC ACID
| 分子 | 名称: ASPARTIC ACID / タイプ: ligand / ID: 5 / コピー数: 1 / 式: ASP |
|---|---|
| 分子量 | 理論値: 133.103 Da |
| Chemical component information |  ChemComp-ASP: |
-分子 #6: ARGININE
| 分子 | 名称: ARGININE / タイプ: ligand / ID: 6 / コピー数: 8 / 式: ARG |
|---|---|
| 分子量 | 理論値: 175.209 Da |
| Chemical component information |  ChemComp-ARG: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.40 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8.2 構成要素:
詳細: The buffer contains 10 mM ATPgammaS, 100 mM aspartate and 20 mM 4-mer cyanophycin peptide as ligands. | ||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR / 詳細: The grid was washed by acetone prior to use. | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 291 K / 装置: FEI VITROBOT MARK IV / 詳細: Blotting time was 10 seconds (blot force 10).. | ||||||||||||
| 詳細 | This sample was mono-disperse. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 1.8 µm / 最小 デフォーカス(公称値): 0.84 µm / 倍率(公称値): 105000 Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 1.8 µm / 最小 デフォーカス(公称値): 0.84 µm / 倍率(公称値): 105000 |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 検出モード: COUNTING / 撮影したグリッド数: 1 / 実像数: 3843 / 平均露光時間: 4.63 sec. / 平均電子線量: 49.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー














 Z
Z Y
Y X
X