+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
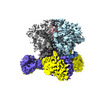
| タイトル | SIV E660.CR54 SOS-2P Env Trimer with ITS92.02 | |||||||||
 マップデータ マップデータ | sharpened map | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報membrane fusion involved in viral entry into host cell / host cell endosome membrane / membrane => GO:0016020 / symbiont entry into host cell /  エンベロープ (ウイルス) / virion attachment to host cell / host cell plasma membrane / virion membrane / structural molecule activity / エンベロープ (ウイルス) / virion attachment to host cell / host cell plasma membrane / virion membrane / structural molecule activity /  細胞膜 細胞膜類似検索 - 分子機能 | |||||||||
| 生物種 |    Influenza A virus (A型インフルエンザウイルス) / Influenza A virus (A型インフルエンザウイルス) /   Simian immunodeficiency virus (サル免疫不全ウイルス) / Simian immunodeficiency virus (サル免疫不全ウイルス) /   Macaca mulatta (アカゲザル) Macaca mulatta (アカゲザル) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 4.32 Å クライオ電子顕微鏡法 / 解像度: 4.32 Å | |||||||||
 データ登録者 データ登録者 | Gorman J / Kwong PD | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2022 ジャーナル: Nat Struct Mol Biol / 年: 2022タイトル: Cryo-EM structures of prefusion SIV envelope trimer. 著者: Jason Gorman / Chunyan Wang / Rosemarie D Mason / Alexandra F Nazzari / Hugh C Welles / Tongqing Zhou / Julian W Bess / Tatsiana Bylund / Myungjin Lee / Yaroslav Tsybovsky / Raffaello Verardi ...著者: Jason Gorman / Chunyan Wang / Rosemarie D Mason / Alexandra F Nazzari / Hugh C Welles / Tongqing Zhou / Julian W Bess / Tatsiana Bylund / Myungjin Lee / Yaroslav Tsybovsky / Raffaello Verardi / Shuishu Wang / Yongping Yang / Baoshan Zhang / Reda Rawi / Brandon F Keele / Jeffrey D Lifson / Jun Liu / Mario Roederer / Peter D Kwong /  要旨: Simian immunodeficiency viruses (SIVs) are lentiviruses that naturally infect non-human primates of African origin and seeded cross-species transmissions of HIV-1 and HIV-2. Here we report prefusion ...Simian immunodeficiency viruses (SIVs) are lentiviruses that naturally infect non-human primates of African origin and seeded cross-species transmissions of HIV-1 and HIV-2. Here we report prefusion stabilization and cryo-EM structures of soluble envelope (Env) trimers from rhesus macaque SIV (SIV) in complex with neutralizing antibodies. These structures provide residue-level definition for SIV-specific disulfide-bonded variable loops (V1 and V2), which we used to delineate variable-loop coverage of the Env trimer. The defined variable loops enabled us to investigate assembled Env-glycan shields throughout SIV, which we found to comprise both N- and O-linked glycans, the latter emanating from V1 inserts, which bound the O-link-specific lectin jacalin. We also investigated in situ SIV-Env trimers on virions, determining cryo-electron tomography structures at subnanometer resolutions for an antibody-bound complex and a ligand-free state. Collectively, these structures define the prefusion-closed structure of the SIV-Env trimer and delineate variable-loop and glycan-shielding mechanisms of immune evasion conserved throughout SIV evolution. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
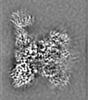
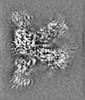
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_27718.map.gz emd_27718.map.gz | 117.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-27718-v30.xml emd-27718-v30.xml emd-27718.xml emd-27718.xml | 23.3 KB 23.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_27718.png emd_27718.png | 88.6 KB | ||
| マスクデータ |  emd_27718_msk_1.map emd_27718_msk_1.map | 125 MB |  マスクマップ マスクマップ | |
| その他 |  emd_27718_additional_1.map.gz emd_27718_additional_1.map.gz emd_27718_additional_2.map.gz emd_27718_additional_2.map.gz emd_27718_half_map_1.map.gz emd_27718_half_map_1.map.gz emd_27718_half_map_2.map.gz emd_27718_half_map_2.map.gz | 17.8 MB 24.3 MB 116.2 MB 116.2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-27718 http://ftp.pdbj.org/pub/emdb/structures/EMD-27718 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27718 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27718 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8duaMC  8dvdC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_27718.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_27718.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | sharpened map | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.083 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_27718_msk_1.map emd_27718_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
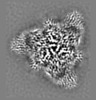
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: density modified map
| ファイル | emd_27718_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | density modified map | ||||||||||||
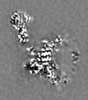
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: unsharpened map
| ファイル | emd_27718_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | unsharpened map | ||||||||||||
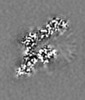
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_27718_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_27718_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : SIV E660.CR54 SOS-2P with ITS92.02
| 全体 | 名称: SIV E660.CR54 SOS-2P with ITS92.02 |
|---|---|
| 要素 |
|
-超分子 #1: SIV E660.CR54 SOS-2P with ITS92.02
| 超分子 | 名称: SIV E660.CR54 SOS-2P with ITS92.02 / タイプ: complex / キメラ: Yes / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 |
|---|---|
| 由来(天然) | 生物種:    Influenza A virus (A型インフルエンザウイルス) Influenza A virus (A型インフルエンザウイルス) |
-分子 #1: Envelope glycoprotein gp41
| 分子 | 名称: Envelope glycoprotein gp41 / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Simian immunodeficiency virus (サル免疫不全ウイルス) Simian immunodeficiency virus (サル免疫不全ウイルス) |
| 分子量 | 理論値: 16.852207 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: GVFVLGFLGF LATAGSAMGA ASLTLSAQSR TLLAGIVQQQ QQLLDVPKRQ QELLRLPVWG TKNLQTRVTA IEKYLKDQAQ LNSWGCAFR QVCCTTVPWP NETLVPNWSN MTWQEWERQV DFLEANITQL LEEAQIQQEK NMYELQKLN |
-分子 #2: Envelope glycoprotein gp120
| 分子 | 名称: Envelope glycoprotein gp120 / タイプ: protein_or_peptide / ID: 2 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Simian immunodeficiency virus (サル免疫不全ウイルス) Simian immunodeficiency virus (サル免疫不全ウイルス) |
| 分子量 | 理論値: 59.053578 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: IQYVTVFYGV PAWKNATIPL FCATKNRDTW GTTQCLPDND DYSELAINIT EAFDAWNNTV TEQAIEDVWN LFETSIKPCV KLTPLCIAM RCNKTETDRW GLTRNAGTTT TTTTTTTAAT PSVAENVINE SNPCIKNNSC AGLEQEPMIG CKFNMTGLKR D KRIEYNET ...文字列: IQYVTVFYGV PAWKNATIPL FCATKNRDTW GTTQCLPDND DYSELAINIT EAFDAWNNTV TEQAIEDVWN LFETSIKPCV KLTPLCIAM RCNKTETDRW GLTRNAGTTT TTTTTTTAAT PSVAENVINE SNPCIKNNSC AGLEQEPMIG CKFNMTGLKR D KRIEYNET WYSRDLICEQ SANESESKCY MHHCNTSVIQ ESCDKHYWDA IRFRYCAPPG YALLRCNDSN YSGFAPNCSK VV VSSCTRM METQTSTWFG FNGTRAENRT YIYWHGKSNR TIISLNKYYN LTMRCRRPGN KTVLPVTIMS GLVFHSQPIN ERP KQAWCW FGGSWKEAIQ EVKETLVKHP RYTGTNDTRK INLTAPAGGD PEVTFMWTNC RGEFLYCKMN WFLNWVEDRD QKSS RWRQQ NTRERQKKNY VPCHIRQIIN TWHKVGKNVY LPPREGDLTC NSTVTSLIAE IDWTNNNETN ITMSAEVAEL YRLEL GDYK LVEITPIGLA PTSCRRYTTT GASRRRRR |
-分子 #3: ITS92.02 Heavy Chain
| 分子 | 名称: ITS92.02 Heavy Chain / タイプ: protein_or_peptide / ID: 3 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Macaca mulatta (アカゲザル) Macaca mulatta (アカゲザル) |
| 分子量 | 理論値: 24.275268 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: QVQLQESGPG LVKPSETLSL TCAVSGGSVS GNWWSWIRQP PGKGLEYIGR ISGSDGTTYY RSSLKVTISR DTSKNQFSLR LNSVTAADT AVYYCARKPT WDWFDVWGPG LLVTVSSAST KGPSVFPLAP SSKSTSGGTA ALGCLVKDYF PEPVTVSWNS G ALTSGVHT ...文字列: QVQLQESGPG LVKPSETLSL TCAVSGGSVS GNWWSWIRQP PGKGLEYIGR ISGSDGTTYY RSSLKVTISR DTSKNQFSLR LNSVTAADT AVYYCARKPT WDWFDVWGPG LLVTVSSAST KGPSVFPLAP SSKSTSGGTA ALGCLVKDYF PEPVTVSWNS G ALTSGVHT FPAVLQSSGL YSLSSVVTVP SSSLGTQTYI CNVNHKPSNT KVDKKVEPKS CDKGLEVLFQ |
-分子 #4: ITS92.02 Light Chain
| 分子 | 名称: ITS92.02 Light Chain / タイプ: protein_or_peptide / ID: 4 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Macaca mulatta (アカゲザル) Macaca mulatta (アカゲザル) |
| 分子量 | 理論値: 23.397963 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: DIQMTQSPSS LSASVGDRVT ITCRASQGID NYLNWYQQKP GKAPKRLIFA ASSLHNGVPS RFSGSGSGTK FTLTISSLQP EDLGTYYCL QYYSDPYTFG QGTKVEIKRT VAAPSVFIFP PSEDQVKSGT VSVVCLLNNF YPREASVKWK VDGALKTGNS Q ESVTEQDS ...文字列: DIQMTQSPSS LSASVGDRVT ITCRASQGID NYLNWYQQKP GKAPKRLIFA ASSLHNGVPS RFSGSGSGTK FTLTISSLQP EDLGTYYCL QYYSDPYTFG QGTKVEIKRT VAAPSVFIFP PSEDQVKSGT VSVVCLLNNF YPREASVKWK VDGALKTGNS Q ESVTEQDS KDNTYSLSST LTLSSTEYQS HKVYACEVTH QGLSSPVTKS FNRGEC |
-分子 #8: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 8 / コピー数: 27 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 / 詳細: PBS |
| 凍結 | 凍結剤: ETHANE |
| 詳細 | SIV E660.CR54 SOS-2P with ITS92.02 |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.2 µm Bright-field microscopy / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.2 µm |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均露光時間: 2.0 sec. / 平均電子線量: 51.16 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
|---|---|
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 4.32 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: cryoSPARC / 使用した粒子像数: 32347 |
 ムービー
ムービー コントローラー
コントローラー












 Z
Z Y
Y X
X