+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-2763 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CryoEM structure of a partial yeast 48S preinitiation complex | |||||||||
 マップデータ マップデータ | Reconstruction of a partial 48S preinitiation complex from yeast. In order to see all the components it is recommended to apply a gaussian filter of 1.5 width and then visualize it at a contour level of 0.02. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Eukaryotic translation initiation / 48S / small ribosome subunit. | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報formation of translation initiation ternary complex / Cellular response to mitochondrial stress / Recycling of eIF2:GDP / selenocysteine metabolic process / ABC-family proteins mediated transport / methionyl-initiator methionine tRNA binding / translation reinitiation / eukaryotic translation initiation factor 2 complex /  selenocysteine insertion sequence binding / multi-eIF complex ...formation of translation initiation ternary complex / Cellular response to mitochondrial stress / Recycling of eIF2:GDP / selenocysteine metabolic process / ABC-family proteins mediated transport / methionyl-initiator methionine tRNA binding / translation reinitiation / eukaryotic translation initiation factor 2 complex / selenocysteine insertion sequence binding / multi-eIF complex ...formation of translation initiation ternary complex / Cellular response to mitochondrial stress / Recycling of eIF2:GDP / selenocysteine metabolic process / ABC-family proteins mediated transport / methionyl-initiator methionine tRNA binding / translation reinitiation / eukaryotic translation initiation factor 2 complex /  selenocysteine insertion sequence binding / multi-eIF complex / eukaryotic 43S preinitiation complex / selenocysteine insertion sequence binding / multi-eIF complex / eukaryotic 43S preinitiation complex /  protein-synthesizing GTPase / formation of translation preinitiation complex / formation of cytoplasmic translation initiation complex / eukaryotic 48S preinitiation complex / positive regulation of translational fidelity / Formation of the ternary complex, and subsequently, the 43S complex / Translation initiation complex formation / Ribosomal scanning and start codon recognition / Formation of a pool of free 40S subunits / endonucleolytic cleavage to generate mature 3'-end of SSU-rRNA from (SSU-rRNA, 5.8S rRNA, LSU-rRNA) / protein-synthesizing GTPase / formation of translation preinitiation complex / formation of cytoplasmic translation initiation complex / eukaryotic 48S preinitiation complex / positive regulation of translational fidelity / Formation of the ternary complex, and subsequently, the 43S complex / Translation initiation complex formation / Ribosomal scanning and start codon recognition / Formation of a pool of free 40S subunits / endonucleolytic cleavage to generate mature 3'-end of SSU-rRNA from (SSU-rRNA, 5.8S rRNA, LSU-rRNA) /  ribosomal small subunit binding / L13a-mediated translational silencing of Ceruloplasmin expression / regulation of translational fidelity / endonucleolytic cleavage in ITS1 to separate SSU-rRNA from 5.8S rRNA and LSU-rRNA from tricistronic rRNA transcript (SSU-rRNA, 5.8S rRNA, LSU-rRNA) / translation regulator activity / 90S preribosome / translational initiation / ribosomal small subunit binding / L13a-mediated translational silencing of Ceruloplasmin expression / regulation of translational fidelity / endonucleolytic cleavage in ITS1 to separate SSU-rRNA from 5.8S rRNA and LSU-rRNA from tricistronic rRNA transcript (SSU-rRNA, 5.8S rRNA, LSU-rRNA) / translation regulator activity / 90S preribosome / translational initiation /  translation initiation factor binding / translation initiation factor binding /  translation initiation factor activity / rescue of stalled ribosome / maturation of SSU-rRNA from tricistronic rRNA transcript (SSU-rRNA, 5.8S rRNA, LSU-rRNA) / maturation of SSU-rRNA / positive regulation of apoptotic signaling pathway / small-subunit processome / translation initiation factor activity / rescue of stalled ribosome / maturation of SSU-rRNA from tricistronic rRNA transcript (SSU-rRNA, 5.8S rRNA, LSU-rRNA) / maturation of SSU-rRNA / positive regulation of apoptotic signaling pathway / small-subunit processome /  protein kinase C binding / maintenance of translational fidelity / modification-dependent protein catabolic process / cytoplasmic stress granule / rRNA processing / protein kinase C binding / maintenance of translational fidelity / modification-dependent protein catabolic process / cytoplasmic stress granule / rRNA processing /  protein tag activity / protein tag activity /  ribosomal small subunit biogenesis / ribosomal small subunit biogenesis /  ribosomal small subunit assembly / small ribosomal subunit rRNA binding / ribosomal small subunit assembly / small ribosomal subunit rRNA binding /  double-stranded RNA binding / double-stranded RNA binding /  ribosome binding / small ribosomal subunit / cytosolic small ribosomal subunit / cytosolic large ribosomal subunit / cytoplasmic translation / ribosome binding / small ribosomal subunit / cytosolic small ribosomal subunit / cytosolic large ribosomal subunit / cytoplasmic translation /  rRNA binding / rRNA binding /  リボソーム / protein ubiquitination / structural constituent of ribosome / リボソーム / protein ubiquitination / structural constituent of ribosome /  ribonucleoprotein complex / ribonucleoprotein complex /  翻訳 (生物学) / positive regulation of protein phosphorylation / 翻訳 (生物学) / positive regulation of protein phosphorylation /  GTPase activity / GTPase activity /  mRNA binding / mRNA binding /  ubiquitin protein ligase binding / ubiquitin protein ligase binding /  核小体 / GTP binding / 核小体 / GTP binding /  protein kinase binding / protein kinase binding /  RNA binding / zinc ion binding / RNA binding / zinc ion binding /  metal ion binding / metal ion binding /  細胞核 / 細胞核 /  細胞質基質 / 細胞質基質 /  細胞質 細胞質類似検索 - 分子機能 | |||||||||
| 生物種 |   Kluyveromyces lactis (酵母) / Kluyveromyces lactis (酵母) /   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母) | |||||||||
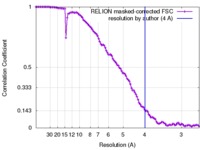
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 4.0 Å クライオ電子顕微鏡法 / 解像度: 4.0 Å | |||||||||
 データ登録者 データ登録者 | Hussain T / Llacer JL / Fernandez IS / Savva CG / Ramakrishnan V | |||||||||
 引用 引用 |  ジャーナル: Cell / 年: 2014 ジャーナル: Cell / 年: 2014タイトル: Structural changes enable start codon recognition by the eukaryotic translation initiation complex. 著者: Tanweer Hussain / Jose L Llácer / Israel S Fernández / Antonio Munoz / Pilar Martin-Marcos / Christos G Savva / Jon R Lorsch / Alan G Hinnebusch / V Ramakrishnan /   要旨: During eukaryotic translation initiation, initiator tRNA does not insert fully into the P decoding site on the 40S ribosomal subunit. This conformation (POUT) is compatible with scanning mRNA for the ...During eukaryotic translation initiation, initiator tRNA does not insert fully into the P decoding site on the 40S ribosomal subunit. This conformation (POUT) is compatible with scanning mRNA for the AUG start codon. Base pairing with AUG is thought to promote isomerization to a more stable conformation (PIN) that arrests scanning and promotes dissociation of eIF1 from the 40S subunit. Here, we present a cryoEM reconstruction of a yeast preinitiation complex at 4.0 Å resolution with initiator tRNA in the PIN state, prior to eIF1 release. The structure reveals stabilization of the codon-anticodon duplex by the N-terminal tail of eIF1A, changes in the structure of eIF1 likely instrumental in its subsequent release, and changes in the conformation of eIF2. The mRNA traverses the entire mRNA cleft and makes connections to the regulatory domain of eIF2?, eIF1A, and ribosomal elements that allow recognition of context nucleotides surrounding the AUG codon. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_2763.map.gz emd_2763.map.gz | 96.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-2763-v30.xml emd-2763-v30.xml emd-2763.xml emd-2763.xml | 15.9 KB 15.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_2763_fsc.xml emd_2763_fsc.xml | 10.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  EMD-2763.png EMD-2763.png emd_2763.png emd_2763.png | 151.3 KB 151.3 KB | ||
| その他 |  py48S_1.34_1.map py48S_1.34_1.map | 103 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-2763 http://ftp.pdbj.org/pub/emdb/structures/EMD-2763 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2763 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2763 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_2763.map.gz / 形式: CCP4 / 大きさ: 100.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_2763.map.gz / 形式: CCP4 / 大きさ: 100.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Reconstruction of a partial 48S preinitiation complex from yeast. In order to see all the components it is recommended to apply a gaussian filter of 1.5 width and then visualize it at a contour level of 0.02. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.34 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-添付マップデータ: py48S 1.34 1.map
| ファイル | py48S_1.34_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Partial yeast 48S preinitiation complex
| 全体 | 名称: Partial yeast 48S preinitiation complex |
|---|---|
| 要素 |
|
-超分子 #1000: Partial yeast 48S preinitiation complex
| 超分子 | 名称: Partial yeast 48S preinitiation complex / タイプ: sample / ID: 1000 / 集合状態: 1 / Number unique components: 6 |
|---|---|
| 分子量 | 理論値: 1.4 MDa |
-超分子 #1: Ribosome small subunit
| 超分子 | 名称: Ribosome small subunit / タイプ: complex / ID: 1 / Name.synonym: 40S / 組換発現: No / Ribosome-details: ribosome-eukaryote: SSU 40S, SSU RNA 18S |
|---|---|
| 由来(天然) | 生物種:   Kluyveromyces lactis (酵母) Kluyveromyces lactis (酵母) |
| 分子量 | 理論値: 1.2 MDa |
-分子 #1: Eukaryotic initiation factor 1
| 分子 | 名称: Eukaryotic initiation factor 1 / タイプ: protein_or_peptide / ID: 1 / Name.synonym: eIF1 / コピー数: 1 / 集合状態: Monomer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae (パン酵母) / 別称: Baker's yeast Saccharomyces cerevisiae (パン酵母) / 別称: Baker's yeast |
| 分子量 | 理論値: 12.3 KDa |
| 組換発現 | 生物種:   Escherichia coli BL21(DE3) (大腸菌) / 組換株: Rosetta / 組換プラスミド: pTYB2 Escherichia coli BL21(DE3) (大腸菌) / 組換株: Rosetta / 組換プラスミド: pTYB2 |
| 配列 | UniProtKB: Eukaryotic translation initiation factor eIF-1 |
-分子 #2: Eukaryotic initiation factor 1A
| 分子 | 名称: Eukaryotic initiation factor 1A / タイプ: protein_or_peptide / ID: 2 / Name.synonym: eIF1A / コピー数: 1 / 集合状態: Monomer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae (パン酵母) / 別称: baker's yeast Saccharomyces cerevisiae (パン酵母) / 別称: baker's yeast |
| 分子量 | 理論値: 17.4 KDa |
| 組換発現 | 生物種:   Escherichia coli BL21(DE3) (大腸菌) / 組換株: Rosetta / 組換プラスミド: pTYB2 Escherichia coli BL21(DE3) (大腸菌) / 組換株: Rosetta / 組換プラスミド: pTYB2 |
| 配列 | UniProtKB: Eukaryotic translation initiation factor 1A |
-分子 #3: Eukaryotic initiation factor 2
| 分子 | 名称: Eukaryotic initiation factor 2 / タイプ: protein_or_peptide / ID: 3 / Name.synonym: eIF2 詳細: Uniprot codes are: alpha-P20459 beta-P09064 gamma-P32481 コピー数: 1 / 集合状態: Three subunits, alpha, beta, gamma / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae (パン酵母) / 別称: Baker's yeast Saccharomyces cerevisiae (パン酵母) / 別称: Baker's yeast |
| 分子量 | 理論値: 124 KDa |
| 組換発現 | 生物種:   Saccharomyces cerevisiae (パン酵母) / 組換株: GP3511 Saccharomyces cerevisiae (パン酵母) / 組換株: GP3511 |
-分子 #4: Initiator transfer RNA
| 分子 | 名称: Initiator transfer RNA / タイプ: rna / ID: 4 / Name.synonym: Met-tRNAi 詳細: Doble mutation (G31U:C39A) when compared with yeast WT initiator tRNA. 分類: TRANSFER / Structure: SINGLE STRANDED / Synthetic?: Yes |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae (パン酵母) / 別称: Baker's yeast Saccharomyces cerevisiae (パン酵母) / 別称: Baker's yeast |
| 分子量 | 理論値: 23 KDa |
| 配列 | 文字列: AGCGCCGUGG CGCAGUGGAA GCGCGCAGGU CUCAUAAACC UGAUGUCCUC GGAUCGAAAC CGAGCGGCGC UACCA |
-分子 #5: Messenger RNA
| 分子 | 名称: Messenger RNA / タイプ: rna / ID: 5 / Name.synonym: mRNA / 分類: OTHER / Structure: SINGLE STRANDED / Synthetic?: Yes |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母) |
| 分子量 | 理論値: 7.4 KDa |
| 配列 | 文字列: GGAAUCUCUC UCUAUGCUCU CUCUC |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.17 mg/mL |
|---|---|
| 緩衝液 | pH: 6.5 詳細: 20mM MES-KOH, 40mM K-acetate, 10mM NH4-acetate, 8mM Mg-acetate, 2mM DTT |
| グリッド | 詳細: Quantifoil R2/2 400 mesh copper grids with 4-5 nm thin carbon on top |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 120 K / 装置: FEI VITROBOT MARK I / Timed resolved state: 30 second incubation time / 手法: Blot for 2.5 seconds before plunging |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI POLARA 300 |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 104478 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2 mm / 最大 デフォーカス(公称値): 0.004 µm / 最小 デフォーカス(公称値): 0.0016 µm / 倍率(公称値): 78000 Bright-field microscopy / Cs: 2 mm / 最大 デフォーカス(公称値): 0.004 µm / 最小 デフォーカス(公称値): 0.0016 µm / 倍率(公称値): 78000 |
| 試料ステージ | 試料ホルダーモデル: GATAN LIQUID NITROGEN |
| アライメント法 | Legacy - 非点収差: Objective lens astigmatism was corrected at 59,000 times magnification |
| 詳細 | Complete dataset was collected in 5 non-consecutive sessions |
| 日付 | 2013年10月28日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: FEI FALCON II (4k x 4k) 実像数: 1791 / 平均電子線量: 28 e/Å2 詳細: Complete dataset was collected in 5 non-consecutive sessions |
| 実験機器 |  モデル: Tecnai Polara / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | PDB ID:  3u5b Chain - Chain ID: 2 |
|---|---|
| ソフトウェア | 名称: Chimera, refmac |
| 精密化 | 空間: RECIPROCAL / プロトコル: FLEXIBLE FIT / 当てはまり具合の基準: R-factor, FSC |
| 得られたモデル |  PDB-3j81: |
-原子モデル構築 2
| 初期モデル | PDB ID:  3u5c |
|---|---|
| ソフトウェア | 名称: Chimera, refmac |
| 精密化 | 空間: RECIPROCAL / プロトコル: FLEXIBLE FIT / 当てはまり具合の基準: R-factor, FSC |
| 得られたモデル |  PDB-3j81: |
 ムービー
ムービー コントローラー
コントローラー






























 Z
Z Y
Y X
X