+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Beta-Arf1 homodimeric interface within AP-1 lattice on narrow membrane tubes | ||||||||||||
 マップデータ マップデータ | |||||||||||||
 試料 試料 |
| ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Glycosphingolipid transport /  basolateral protein secretion / perturbation by virus of host immune response / negative regulation of CD4 production / mitotic cleavage furrow ingression / symbiont-mediated suppression of host T-cell mediated immune response / AP-1 adaptor complex / endosome to melanosome transport / trans-Golgi Network Vesicle Budding / Lysosome Vesicle Biogenesis ...Glycosphingolipid transport / basolateral protein secretion / perturbation by virus of host immune response / negative regulation of CD4 production / mitotic cleavage furrow ingression / symbiont-mediated suppression of host T-cell mediated immune response / AP-1 adaptor complex / endosome to melanosome transport / trans-Golgi Network Vesicle Budding / Lysosome Vesicle Biogenesis ...Glycosphingolipid transport /  basolateral protein secretion / perturbation by virus of host immune response / negative regulation of CD4 production / mitotic cleavage furrow ingression / symbiont-mediated suppression of host T-cell mediated immune response / AP-1 adaptor complex / endosome to melanosome transport / trans-Golgi Network Vesicle Budding / Lysosome Vesicle Biogenesis / platelet dense granule organization / basolateral protein secretion / perturbation by virus of host immune response / negative regulation of CD4 production / mitotic cleavage furrow ingression / symbiont-mediated suppression of host T-cell mediated immune response / AP-1 adaptor complex / endosome to melanosome transport / trans-Golgi Network Vesicle Budding / Lysosome Vesicle Biogenesis / platelet dense granule organization /  regulation of receptor internalization / protein trimerization / regulation of receptor internalization / protein trimerization /  melanosome assembly / regulation of Arp2/3 complex-mediated actin nucleation / symbiont-mediated suppression of host antigen processing and presentation of peptide antigen via MHC class I / Intra-Golgi traffic / Golgi to vacuole transport / symbiont-mediated suppression of host antigen processing and presentation of peptide antigen via MHC class II / Synthesis of PIPs at the Golgi membrane / Golgi Associated Vesicle Biogenesis / suppression by virus of host autophagy / clathrin adaptor activity / MHC class II antigen presentation / melanosome assembly / regulation of Arp2/3 complex-mediated actin nucleation / symbiont-mediated suppression of host antigen processing and presentation of peptide antigen via MHC class I / Intra-Golgi traffic / Golgi to vacuole transport / symbiont-mediated suppression of host antigen processing and presentation of peptide antigen via MHC class II / Synthesis of PIPs at the Golgi membrane / Golgi Associated Vesicle Biogenesis / suppression by virus of host autophagy / clathrin adaptor activity / MHC class II antigen presentation /  thioesterase binding / CD4 receptor binding / Nef Mediated CD4 Down-regulation / dendritic spine organization / determination of left/right symmetry / long-term synaptic depression / COPI-dependent Golgi-to-ER retrograde traffic / thioesterase binding / CD4 receptor binding / Nef Mediated CD4 Down-regulation / dendritic spine organization / determination of left/right symmetry / long-term synaptic depression / COPI-dependent Golgi-to-ER retrograde traffic /  クラスリン / Lysosome Vesicle Biogenesis / クラスリン / Lysosome Vesicle Biogenesis /  clathrin binding / Golgi Associated Vesicle Biogenesis / T cell mediated cytotoxicity directed against tumor cell target / positive regulation of memory T cell activation / TAP complex binding / host cell Golgi membrane / Golgi medial cisterna / cell leading edge / antigen processing and presentation of exogenous peptide antigen via MHC class I / positive regulation of CD8-positive, alpha-beta T cell activation / CD8-positive, alpha-beta T cell activation / positive regulation of CD8-positive, alpha-beta T cell proliferation / CD8 receptor binding / Synthesis of PIPs at the plasma membrane / antigen processing and presentation of endogenous peptide antigen via MHC class I via ER pathway, TAP-dependent / MHC class I protein binding / intracellular copper ion homeostasis / endoplasmic reticulum exit site / clathrin binding / Golgi Associated Vesicle Biogenesis / T cell mediated cytotoxicity directed against tumor cell target / positive regulation of memory T cell activation / TAP complex binding / host cell Golgi membrane / Golgi medial cisterna / cell leading edge / antigen processing and presentation of exogenous peptide antigen via MHC class I / positive regulation of CD8-positive, alpha-beta T cell activation / CD8-positive, alpha-beta T cell activation / positive regulation of CD8-positive, alpha-beta T cell proliferation / CD8 receptor binding / Synthesis of PIPs at the plasma membrane / antigen processing and presentation of endogenous peptide antigen via MHC class I via ER pathway, TAP-dependent / MHC class I protein binding / intracellular copper ion homeostasis / endoplasmic reticulum exit site /  protein targeting / TAP binding / protein targeting / TAP binding /  protection from natural killer cell mediated cytotoxicity / protection from natural killer cell mediated cytotoxicity /  beta-2-microglobulin binding / COPI-mediated anterograde transport / beta-2-microglobulin binding / COPI-mediated anterograde transport /  クラスリン / vesicle-mediated transport / regulation of calcium-mediated signaling / detection of bacterium / クラスリン / vesicle-mediated transport / regulation of calcium-mediated signaling / detection of bacterium /  ウイルスのライフサイクル / ウイルスのライフサイクル /  T cell receptor binding / MHC class II antigen presentation / Gene and protein expression by JAK-STAT signaling after Interleukin-12 stimulation / Neutrophil degranulation / antigen processing and presentation of endogenous peptide antigen via MHC class I via ER pathway, TAP-independent / antigen processing and presentation of endogenous peptide antigen via MHC class Ib / T cell receptor binding / MHC class II antigen presentation / Gene and protein expression by JAK-STAT signaling after Interleukin-12 stimulation / Neutrophil degranulation / antigen processing and presentation of endogenous peptide antigen via MHC class I via ER pathway, TAP-independent / antigen processing and presentation of endogenous peptide antigen via MHC class Ib /  sarcomere / sarcomere /  低分子量GTPアーゼ / trans-Golgi network membrane / 低分子量GTPアーゼ / trans-Golgi network membrane /  kidney development / Nef mediated downregulation of MHC class I complex cell surface expression / kidney development / Nef mediated downregulation of MHC class I complex cell surface expression /  virion component / lumenal side of endoplasmic reticulum membrane / Endosomal/Vacuolar pathway / Antigen Presentation: Folding, assembly and peptide loading of class I MHC / virion component / lumenal side of endoplasmic reticulum membrane / Endosomal/Vacuolar pathway / Antigen Presentation: Folding, assembly and peptide loading of class I MHC /  intracellular protein transport / intracellular protein transport /  ゴルジ体 / cytoplasmic vesicle membrane / ER to Golgi transport vesicle membrane / peptide antigen assembly with MHC class I protein complex / MHC class I peptide loading complex / T cell mediated cytotoxicity / antigen processing and presentation of endogenous peptide antigen via MHC class I / positive regulation of T cell cytokine production / cellular response to virus / MHC class I protein complex / ゴルジ体 / cytoplasmic vesicle membrane / ER to Golgi transport vesicle membrane / peptide antigen assembly with MHC class I protein complex / MHC class I peptide loading complex / T cell mediated cytotoxicity / antigen processing and presentation of endogenous peptide antigen via MHC class I / positive regulation of T cell cytokine production / cellular response to virus / MHC class I protein complex /  SH3 domain binding / positive regulation of T cell mediated cytotoxicity / recycling endosome membrane / phagocytic vesicle membrane / peptide antigen binding / positive regulation of type II interferon production / Immunoregulatory interactions between a Lymphoid and a non-Lymphoid cell / Interferon gamma signaling / Interferon alpha/beta signaling / E3 ubiquitin ligases ubiquitinate target proteins / SH3 domain binding / positive regulation of T cell mediated cytotoxicity / recycling endosome membrane / phagocytic vesicle membrane / peptide antigen binding / positive regulation of type II interferon production / Immunoregulatory interactions between a Lymphoid and a non-Lymphoid cell / Interferon gamma signaling / Interferon alpha/beta signaling / E3 ubiquitin ligases ubiquitinate target proteins /  heart development / ER-Phagosome pathway / T cell receptor signaling pathway heart development / ER-Phagosome pathway / T cell receptor signaling pathway類似検索 - 分子機能 | ||||||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||||||||
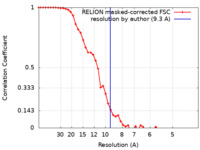
| 手法 | サブトモグラム平均法 /  クライオ電子顕微鏡法 / 解像度: 9.3 Å クライオ電子顕微鏡法 / 解像度: 9.3 Å | ||||||||||||
 データ登録者 データ登録者 | Hooy RM / Hurley JH | ||||||||||||
| 資金援助 |  米国, 3件 米国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Sci Adv / 年: 2022 ジャーナル: Sci Adv / 年: 2022タイトル: Self-assembly and structure of a clathrin-independent AP-1:Arf1 tubular membrane coat. 著者: Richard M Hooy / Yuichiro Iwamoto / Dan A Tudorica / Xuefeng Ren / James H Hurley /  要旨: The adaptor protein (AP) complexes not only form the inner layer of clathrin coats but also have clathrin-independent roles in membrane traffic whose mechanisms are unknown. HIV-1 Nef hijacks AP-1 to ...The adaptor protein (AP) complexes not only form the inner layer of clathrin coats but also have clathrin-independent roles in membrane traffic whose mechanisms are unknown. HIV-1 Nef hijacks AP-1 to sequester major histocompatibility complex class I (MHC-I), evading immune detection. We found that AP-1:Arf1:Nef:MHC-I forms a coat on tubulated membranes without clathrin and determined its structure. The coat assembles via Arf1 dimer interfaces. AP-1-positive tubules are enriched in cells upon clathrin knockdown. Nef localizes preferentially to AP-1 tubules in cells, explaining how Nef sequesters MHC-I. Coat contact residues are conserved across Arf isoforms and the Arf-dependent AP complexes AP-1, AP-3, and AP-4. Thus, AP complexes can self-assemble with Arf1 into tubular coats without clathrin or other scaffolding factors. The AP-1:Arf1 coat defines the structural basis of a broader class of tubulovesicular membrane coats as an intermediate in clathrin vesicle formation from internal membranes and as an MHC-I sequestration mechanism in HIV-1 infection. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_27186.map.gz emd_27186.map.gz | 5.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-27186-v30.xml emd-27186-v30.xml emd-27186.xml emd-27186.xml | 21.2 KB 21.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_27186_fsc.xml emd_27186_fsc.xml | 4.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_27186.png emd_27186.png | 35.5 KB | ||
| マスクデータ |  emd_27186_msk_1.map emd_27186_msk_1.map | 8 MB |  マスクマップ マスクマップ | |
| その他 |  emd_27186_half_map_1.map.gz emd_27186_half_map_1.map.gz emd_27186_half_map_2.map.gz emd_27186_half_map_2.map.gz | 7.3 MB 7.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-27186 http://ftp.pdbj.org/pub/emdb/structures/EMD-27186 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27186 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27186 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8d9wMC  7ux3C  8d4cC  8d4dC  8d4eC  8d4fC  8d4gC  8d9rC  8d9sC  8d9tC  8d9uC  8d9vC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_27186.map.gz / 形式: CCP4 / 大きさ: 8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_27186.map.gz / 形式: CCP4 / 大きさ: 8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 2.1 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_27186_msk_1.map emd_27186_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
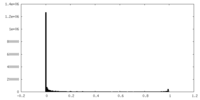
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_27186_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
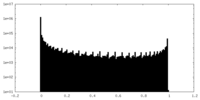
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_27186_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
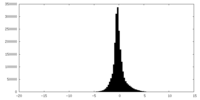
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Assembly of AP-1, Arf1 and Nef complexes on narrow membrane tubes
| 全体 | 名称: Assembly of AP-1, Arf1 and Nef complexes on narrow membrane tubes |
|---|---|
| 要素 |
|
-超分子 #1: Assembly of AP-1, Arf1 and Nef complexes on narrow membrane tubes
| 超分子 | 名称: Assembly of AP-1, Arf1 and Nef complexes on narrow membrane tubes タイプ: complex / キメラ: Yes / ID: 1 / 親要素: 0 / 含まれる分子: #1-#8 詳細: Subtomogram average of beta-Arf1 homodimeric interface within AP-1 lattice on narrow membrane tubes |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
-超分子 #2: AP-1 heterotetramer
| 超分子 | 名称: AP-1 heterotetramer / タイプ: complex / キメラ: Yes / ID: 2 / 親要素: 1 / 含まれる分子: #4-#5, #7-#8 詳細: All four subunits are co-expressed from the same plasmid. Assembly occurs in situ during expression. |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:   Escherichia coli BL21(DE3) (大腸菌) Escherichia coli BL21(DE3) (大腸菌) |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.2 mg/mL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.2 構成要素:
詳細: HEPES/KOAc concentrated stocks are diluted to their final concentrations then pH'd to 7.2 with KOH prior to use in experiments. | |||||||||||||||
| グリッド | モデル: EMS Lacey Carbon / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: LACEY / 支持フィルム - Film thickness: 50.0 nm | |||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 298 K / 装置: FEI VITROBOT MARK IV 詳細: 60 second wait, 3-5 second blot, 597 filter paper, 0.5 second drain. Sample was supplemented with 10nm BSA-gold fiducials. 3.5ul of the mixture was double-side blotted.. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 4.5 µm / 最小 デフォーカス(公称値): 1.5 µm / 倍率(公称値): 42000 Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 4.5 µm / 最小 デフォーカス(公称値): 1.5 µm / 倍率(公称値): 42000 |
| 特殊光学系 | エネルギーフィルター - スリット幅: 25 eV |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) デジタル化 - サイズ - 横: 5760 pixel / デジタル化 - サイズ - 縦: 4092 pixel / 撮影したグリッド数: 1 / 平均露光時間: 3.0 sec. / 平均電子線量: 3.0 e/Å2 詳細: Tilt images were collected in movie-mode. Each movie/tilt consisted of 3-4 frames each |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 精密化 | プロトコル: RIGID BODY FIT |
|---|---|
| 得られたモデル |  PDB-8d9w: |
 ムービー
ムービー コントローラー
コントローラー













































 Z
Z Y
Y X
X