+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of T/F100 SOSIP.664 HIV-1 Env trimer with LMHS mutations in complex with Temsavir, 8ANC195, and 10-1074 | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | T/F100 SOSIP / Clade A/E HIV-1 / 8ANC195 / 10-1074 / BMS-626529 / Temsavir / LMHS mutant. /  VIRAL PROTEIN-INHIBITOR complex (ウイルス性) VIRAL PROTEIN-INHIBITOR complex (ウイルス性) | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報positive regulation of plasma membrane raft polarization / positive regulation of receptor clustering / positive regulation of establishment of T cell polarity / virus-mediated perturbation of host defense response / host cell endosome membrane / clathrin-dependent endocytosis of virus by host cell /  viral protein processing / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane / viral protein processing / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane /  エンベロープ (ウイルス) ...positive regulation of plasma membrane raft polarization / positive regulation of receptor clustering / positive regulation of establishment of T cell polarity / virus-mediated perturbation of host defense response / host cell endosome membrane / clathrin-dependent endocytosis of virus by host cell / エンベロープ (ウイルス) ...positive regulation of plasma membrane raft polarization / positive regulation of receptor clustering / positive regulation of establishment of T cell polarity / virus-mediated perturbation of host defense response / host cell endosome membrane / clathrin-dependent endocytosis of virus by host cell /  viral protein processing / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane / viral protein processing / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane /  エンベロープ (ウイルス) / virion attachment to host cell / host cell plasma membrane / virion membrane / structural molecule activity / エンベロープ (ウイルス) / virion attachment to host cell / host cell plasma membrane / virion membrane / structural molecule activity /  細胞膜 細胞膜類似検索 - 分子機能 | |||||||||
| 生物種 |   Severe acute respiratory syndrome coronavirus 2 (SARSコロナウイルス2) / Severe acute respiratory syndrome coronavirus 2 (SARSコロナウイルス2) /    Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) / Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) /   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
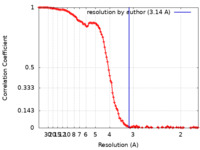
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.14 Å クライオ電子顕微鏡法 / 解像度: 3.14 Å | |||||||||
 データ登録者 データ登録者 | Chen Y / Pozharski E / Tolbert W / Pazgier M | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2023 ジャーナル: Nat Commun / 年: 2023タイトル: Structure-function analyses reveal key molecular determinants of HIV-1 CRF01_AE resistance to the entry inhibitor temsavir. 著者: Jérémie Prévost / Yaozong Chen / Fei Zhou / William D Tolbert / Romain Gasser / Halima Medjahed / Manon Nayrac / Dung N Nguyen / Suneetha Gottumukkala / Ann J Hessell / Venigalla B Rao / ...著者: Jérémie Prévost / Yaozong Chen / Fei Zhou / William D Tolbert / Romain Gasser / Halima Medjahed / Manon Nayrac / Dung N Nguyen / Suneetha Gottumukkala / Ann J Hessell / Venigalla B Rao / Edwin Pozharski / Rick K Huang / Doreen Matthies / Andrés Finzi / Marzena Pazgier /   要旨: The HIV-1 entry inhibitor temsavir prevents the viral receptor CD4 (cluster of differentiation 4) from interacting with the envelope glycoprotein (Env) and blocks its conformational changes. To do ...The HIV-1 entry inhibitor temsavir prevents the viral receptor CD4 (cluster of differentiation 4) from interacting with the envelope glycoprotein (Env) and blocks its conformational changes. To do this, temsavir relies on the presence of a residue with small side chain at position 375 in Env and is unable to neutralize viral strains like CRF01_AE carrying His375. Here we investigate the mechanism of temsavir resistance and show that residue 375 is not the sole determinant of resistance. At least six additional residues within the gp120 inner domain layers, including five distant from the drug-binding pocket, contribute to resistance. A detailed structure-function analysis using engineered viruses and soluble trimer variants reveals that the molecular basis of resistance is mediated by crosstalk between His375 and the inner domain layers. Furthermore, our data confirm that temsavir can adjust its binding mode to accommodate changes in Env conformation, a property that likely contributes to its broad antiviral activity. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_27103.map.gz emd_27103.map.gz | 122.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-27103-v30.xml emd-27103-v30.xml emd-27103.xml emd-27103.xml | 25.2 KB 25.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_27103_fsc.xml emd_27103_fsc.xml | 14.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_27103.png emd_27103.png | 194.4 KB | ||
| Filedesc metadata |  emd-27103.cif.gz emd-27103.cif.gz | 7.7 KB | ||
| その他 |  emd_27103_half_map_1.map.gz emd_27103_half_map_1.map.gz emd_27103_half_map_2.map.gz emd_27103_half_map_2.map.gz | 109.6 MB 109.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-27103 http://ftp.pdbj.org/pub/emdb/structures/EMD-27103 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27103 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27103 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_27103.map.gz / 形式: CCP4 / 大きさ: 129.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_27103.map.gz / 形式: CCP4 / 大きさ: 129.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 0.8893 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_27103_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_27103_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Cryo-EM structure of T/F100 SOSIP.664 HIV-1 Env trimer with LMHS ...
| 全体 | 名称: Cryo-EM structure of T/F100 SOSIP.664 HIV-1 Env trimer with LMHS mutations in complex with Temsavir, 8ANC195, and 10-1074 |
|---|---|
| 要素 |
|
-超分子 #1: Cryo-EM structure of T/F100 SOSIP.664 HIV-1 Env trimer with LMHS ...
| 超分子 | 名称: Cryo-EM structure of T/F100 SOSIP.664 HIV-1 Env trimer with LMHS mutations in complex with Temsavir, 8ANC195, and 10-1074 タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#6 詳細: LM mutations refer to layer mutants (H61Y/Q105H/V108I/N474D/I475M/K476R) and HS mutation refer to H375S. Temsavir (BMS-626529) is a novel small-molecule HIV-1 attachment inhibitor. |
|---|---|
| 由来(天然) | 生物種:   Severe acute respiratory syndrome coronavirus 2 (SARSコロナウイルス2) Severe acute respiratory syndrome coronavirus 2 (SARSコロナウイルス2) |
| 分子量 | 理論値: 6.3 MDa |
-分子 #1: CRF01_AE T/F100 HIV-1 gp120
| 分子 | 名称: CRF01_AE T/F100 HIV-1 gp120 / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:    Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
| 分子量 | 理論値: 54.784176 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: ATNNLWVTVY YGVPVWRDAD TTLFCASDAK AYETEVHNVW ATHACVPTDP NPQEMHLKNV TENFNMWKNN MVEQMHEDII SLWDQSLKP CVKLTPLCVT LNCTSATVTN YTKVNDTSDI IGNITDDVRN CSFNMTTELR DKQQKVYALF YKLDIVPIDD S SNNGSSNF ...文字列: ATNNLWVTVY YGVPVWRDAD TTLFCASDAK AYETEVHNVW ATHACVPTDP NPQEMHLKNV TENFNMWKNN MVEQMHEDII SLWDQSLKP CVKLTPLCVT LNCTSATVTN YTKVNDTSDI IGNITDDVRN CSFNMTTELR DKQQKVYALF YKLDIVPIDD S SNNGSSNF SEYRLINCNT SVIKQACPKV SFDPIPIHYC TPAGYAILRC NDKKFNGTGP CKNVSSVQCT HGIKPVVSTQ LL LNGSLAE EGIIIRSENL TNNAKTIIVH FNESVKINCT RPSNNTRTGI HIGPGQVFYK TGDIIGDIRK AYCNISGAQW HKV LGRVAN KLKEHFNNKT IVFKPSSGGD PEITMHSFNC RGEFFYCNTT KLFNSTWGGN KNETRDNGTI TIPCRIKQII NMWQ GVGQA MYAPPIKGVI KCLSNITGIL LTRDGGNDST ENNETFRPGG GDMRDNWRNE LYKYKVVQIE PLGIAPTKCK RRVVE RRRR RR UniProtKB: Envelope glycoprotein gp160 |
-分子 #2: CRF-1_AE T/F100 HIV-1 gp41
| 分子 | 名称: CRF-1_AE T/F100 HIV-1 gp41 / タイプ: protein_or_peptide / ID: 2 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:    Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
| 分子量 | 理論値: 17.389781 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: AVGLGAMIFG FLGAAGSTMG AASITLTVQA RQLLSGIVQQ QSNLLRAPEA QQHLLQLTVW GIKQLQARVL AVERYLQDQK FLGLWGCSG KIICCTAVPW NSSWSNKTFE EIWNNMTWIE WEREISNYTS QIYDILTISQ TQQEKNEKDL LELDAA UniProtKB: Envelope glycoprotein gp160 |
-分子 #3: Heavy chain of 8ANC195 Fab
| 分子 | 名称: Heavy chain of 8ANC195 Fab / タイプ: protein_or_peptide / ID: 3 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 25.324402 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: QIHLVQSGTE VKKPGSSVTV SCKAYGVNTF GLYAVNWVRQ APGQSLEYIG QIWRWKSSAS HHFRGRVLIS AVDLTGSSPP ISSLEIKNL TSDDTAVYFC TTTSTYDRWS GLHHDGVMAF SSWGQGTLIS VSAASTKGPS VFPLAPSSKS TSGGTAALGC L VKDYFPEP ...文字列: QIHLVQSGTE VKKPGSSVTV SCKAYGVNTF GLYAVNWVRQ APGQSLEYIG QIWRWKSSAS HHFRGRVLIS AVDLTGSSPP ISSLEIKNL TSDDTAVYFC TTTSTYDRWS GLHHDGVMAF SSWGQGTLIS VSAASTKGPS VFPLAPSSKS TSGGTAALGC L VKDYFPEP VTVSWNSGAL TSGVHTFPAV LQSSGLYSLS SVVTVPSSSL GTQTYICNVN HKPSNTKVDK RVEPKSCDKT |
-分子 #4: Light chain of 8ANC195 Fab
| 分子 | 名称: Light chain of 8ANC195 Fab / タイプ: protein_or_peptide / ID: 4 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 23.460047 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: DIQMTQSPST LSASTGDTVR ISCRASQSIT GNWVAWYQQR PGKAPRLLIY RGAALLGGVP SRFRGSAAGT DFTLTIGNLQ AEDFGTFYC QQYDTYPGTF GQGTKVEVKR TVAAPSVFIF PPSDEQLKSG TASVVCLLNN FYPREAKVQW KVDNALQSGN S QESVTEQD ...文字列: DIQMTQSPST LSASTGDTVR ISCRASQSIT GNWVAWYQQR PGKAPRLLIY RGAALLGGVP SRFRGSAAGT DFTLTIGNLQ AEDFGTFYC QQYDTYPGTF GQGTKVEVKR TVAAPSVFIF PPSDEQLKSG TASVVCLLNN FYPREAKVQW KVDNALQSGN S QESVTEQD SKDSTYSLSS TLTLSKADYE KHKVYACEVT HQGLSSPVTK SFNRGEC |
-分子 #5: Heavy chain of 10-1074 Fab
| 分子 | 名称: Heavy chain of 10-1074 Fab / タイプ: protein_or_peptide / ID: 5 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 25.661688 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: QVQLQESGPG LVKPSETLSV TCSVSGDSMN NYYWTWIRQS PGKGLEWIGY ISDRESATYN PSLNSRVVIS RDTSKNQLSL KLNSVTPAD TAVYYCATAR RGQRIYGVVS FGEFFYYYSM DVWGKGTTVT VSSASTKGPS VFPLAPSSKS TSGGTAALGC L VKDYFPEP ...文字列: QVQLQESGPG LVKPSETLSV TCSVSGDSMN NYYWTWIRQS PGKGLEWIGY ISDRESATYN PSLNSRVVIS RDTSKNQLSL KLNSVTPAD TAVYYCATAR RGQRIYGVVS FGEFFYYYSM DVWGKGTTVT VSSASTKGPS VFPLAPSSKS TSGGTAALGC L VKDYFPEP VTVSWNSGAL TSGVHTFPAV LQSSGLYSLS SVVTVPSSSL GTQTYICNVN HKPSNTKVDK RVEPKSCDKT |
-分子 #6: Light chain of 10-1074 Fab
| 分子 | 名称: Light chain of 10-1074 Fab / タイプ: protein_or_peptide / ID: 6 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 23.18076 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: SYVRPLSVAL GETARISCGR QALGSRAVQW YQHRPGQAPI LLIYNNQDRP SGIPERFSGT PDINFGTRAT LTISGVEAGD EADYYCHMW DSRSGFSWSF GGATRLTVLG QPKAAPSVTL FPPSSEELQA NKATLVCLIS DFYPGAVTVA WKADSSPVKA G VETTTPSK ...文字列: SYVRPLSVAL GETARISCGR QALGSRAVQW YQHRPGQAPI LLIYNNQDRP SGIPERFSGT PDINFGTRAT LTISGVEAGD EADYYCHMW DSRSGFSWSF GGATRLTVLG QPKAAPSVTL FPPSSEELQA NKATLVCLIS DFYPGAVTVA WKADSSPVKA G VETTTPSK QSNNKYAASS YLSLTPEQWK SHRSYSCQVT HEGSTVEKTV APTECS |
-分子 #14: 1-[4-(benzenecarbonyl)piperazin-1-yl]-2-[4-methoxy-7-(3-methyl-1H...
| 分子 | 名称: 1-[4-(benzenecarbonyl)piperazin-1-yl]-2-[4-methoxy-7-(3-methyl-1H-1,2,4-triazol-1-yl)-1H-pyrrolo[2,3-c]pyridin-3-yl]ethane-1,2-dione タイプ: ligand / ID: 14 / コピー数: 3 / 式: 83J |
|---|---|
| 分子量 | 理論値: 473.484 Da |
| Chemical component information | 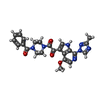 ChemComp-83J: |
-分子 #15: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 15 / コピー数: 34 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.93 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 支持フィルム - Film thickness: 0.2 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS GLACIOS |
|---|---|
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 2.7 µm / 最小 デフォーカス(公称値): 0.5 µm Bright-field microscopy / 最大 デフォーカス(公称値): 2.7 µm / 最小 デフォーカス(公称値): 0.5 µm |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 実像数: 1484 / 平均電子線量: 58.14 e/Å2 |
- 画像解析
画像解析
-原子モデル構築 1
| 初期モデル |
| ||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT | ||||||||||||||
| 得られたモデル |  PDB-8czz: |
 ムービー
ムービー コントローラー
コントローラー













 Z
Z Y
Y X
X