+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of RecT protein from Listeria innoccua phage A118 in complex with 83-mer annealed duplex | |||||||||
 マップデータ マップデータ | Density modified map from Phenix-Resolve, including 10-fold NCS averaging. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | DNA Recombination / DNA Annealing /  DNA BINDING PROTEIN (DNA結合タンパク質) / DNA BINDING PROTEIN-DNA complex DNA BINDING PROTEIN (DNA結合タンパク質) / DNA BINDING PROTEIN-DNA complex | |||||||||
| 機能・相同性 | DNA single-strand annealing protein RecT /  RecT family / RecT family /  RecT family / DNA metabolic process / RecT family / DNA metabolic process /  DNA binding / Recombinase [Bacteriophage A118] DNA binding / Recombinase [Bacteriophage A118] 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |   Listeria innocua Clip11262 (バクテリア) / Listeria innocua Clip11262 (バクテリア) /   Escherichia virus M13 (ウイルス) Escherichia virus M13 (ウイルス) | |||||||||
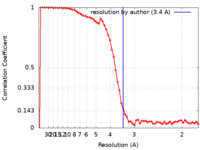
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.4 Å クライオ電子顕微鏡法 / 解像度: 3.4 Å | |||||||||
 データ登録者 データ登録者 | Bell CE / Caldwell BJ | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2022 ジャーナル: Nat Commun / 年: 2022タイトル: Structure of a RecT/Redβ family recombinase in complex with a duplex intermediate of DNA annealing. 著者: Brian J Caldwell / Andrew S Norris / Caroline F Karbowski / Alyssa M Wiegand / Vicki H Wysocki / Charles E Bell /  要旨: Some bacteriophage encode a recombinase that catalyzes single-stranded DNA annealing (SSA). These proteins are apparently related to RAD52, the primary human SSA protein. The best studied protein, ...Some bacteriophage encode a recombinase that catalyzes single-stranded DNA annealing (SSA). These proteins are apparently related to RAD52, the primary human SSA protein. The best studied protein, Redβ from bacteriophage λ, binds weakly to ssDNA, not at all to dsDNA, but tightly to a duplex intermediate of annealing formed when two complementary DNA strands are added to the protein sequentially. We used single particle cryo-electron microscopy (cryo-EM) to determine a 3.4 Å structure of a Redβ homolog from a prophage of Listeria innocua in complex with two complementary 83mer oligonucleotides. The structure reveals a helical protein filament bound to a DNA duplex that is highly extended and unwound. Native mass spectrometry confirms that the complex seen by cryo-EM is the predominant species in solution. The protein shares a common core fold with RAD52 and a similar mode of ssDNA-binding. These data provide insights into the mechanism of protein-catalyzed SSA. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
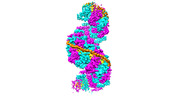
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_26434.map.gz emd_26434.map.gz | 77.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-26434-v30.xml emd-26434-v30.xml emd-26434.xml emd-26434.xml | 19.8 KB 19.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_26434_fsc.xml emd_26434_fsc.xml | 7.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_26434.png emd_26434.png | 46.5 KB | ||
| Filedesc metadata |  emd-26434.cif.gz emd-26434.cif.gz | 6.7 KB | ||
| その他 |  emd_26434_half_map_1.map.gz emd_26434_half_map_1.map.gz emd_26434_half_map_2.map.gz emd_26434_half_map_2.map.gz | 77.6 MB 77.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-26434 http://ftp.pdbj.org/pub/emdb/structures/EMD-26434 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26434 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26434 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7ub2MC  7ubbC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_26434.map.gz / 形式: CCP4 / 大きさ: 83.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_26434.map.gz / 形式: CCP4 / 大きさ: 83.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Density modified map from Phenix-Resolve, including 10-fold NCS averaging. | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.899 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: Un-masked half map
| ファイル | emd_26434_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Un-masked half map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Un-masked half map
| ファイル | emd_26434_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Un-masked half map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : RecT protein from Listeria innocua phage A118, complexed with two...
| 全体 | 名称: RecT protein from Listeria innocua phage A118, complexed with two complementary strands of ssDNA that were added to the protein sequentially 矩形関数 矩形関数 |
|---|---|
| 要素 |
|
-超分子 #1: RecT protein from Listeria innocua phage A118, complexed with two...
| 超分子 | 名称: RecT protein from Listeria innocua phage A118, complexed with two complementary strands of ssDNA that were added to the protein sequentially タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: The protein was purified by Nickel affinity and anion exchange chromatography. The DNA was chemically synthesized and HPLC purified. |
|---|---|
| 由来(天然) | 生物種:   Listeria innocua Clip11262 (バクテリア) Listeria innocua Clip11262 (バクテリア) |
| 分子量 | 理論値: 602 KDa |
-分子 #1: RecT
| 分子 | 名称: RecT / タイプ: protein_or_peptide / ID: 1 / コピー数: 10 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Listeria innocua Clip11262 (バクテリア) / 株: ATCC BAA-680 / CLIP 11262 Listeria innocua Clip11262 (バクテリア) / 株: ATCC BAA-680 / CLIP 11262 |
| 分子量 | 理論値: 30.9391 KDa |
| 組換発現 | 生物種:   Escherichia coli BL21 (大腸菌) Escherichia coli BL21 (大腸菌) |
| 配列 | 文字列: GSHMATNDEL KNQLANKQNG GQVASAQSLD LKGLLEAPTM RKKFEKVLDK KAPQFLTSLL NLYNGDDYLQ KTDPMTVVTS AMVAATLDL PIDKNLGYAW IVPYKGRAQF QLGYKGYIQL ALRTGQYKSI NVIEVREGEL LKWNRLTEEI ELDLDNNTSE K VVGYCGYF ...文字列: GSHMATNDEL KNQLANKQNG GQVASAQSLD LKGLLEAPTM RKKFEKVLDK KAPQFLTSLL NLYNGDDYLQ KTDPMTVVTS AMVAATLDL PIDKNLGYAW IVPYKGRAQF QLGYKGYIQL ALRTGQYKSI NVIEVREGEL LKWNRLTEEI ELDLDNNTSE K VVGYCGYF QLINGFEKTV YWTRKEIEAH KQKFSKSDFG WKKDYDAMAK KTVLRNMLSK WGILSIDMQT AVTEDEAEPR ER KDVTDDE SIPDIIDAPV TPSDTLEAGS VVQGSMI UniProtKB: Recombinase [Bacteriophage A118] |
-分子 #2: DNA (49-mer)
| 分子 | 名称: DNA (49-mer) / タイプ: dna / ID: 2 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:   Escherichia virus M13 (ウイルス) Escherichia virus M13 (ウイルス) |
| 分子量 | 理論値: 15.30217 KDa |
| 配列 | 文字列: (DA)(DA)(DA)(DA)(DA)(DA)(DA)(DA)(DA)(DA) (DA)(DA)(DA)(DA)(DA)(DA)(DA)(DA)(DA)(DA) (DA)(DA)(DA)(DA)(DA)(DA)(DA)(DA)(DA) (DA)(DA)(DA)(DA)(DA)(DA)(DA)(DA)(DA)(DA) (DA) (DA)(DA)(DA)(DA)(DA)(DA)(DA)(DA) (DA) |
-分子 #3: DNA (49-mer)
| 分子 | 名称: DNA (49-mer) / タイプ: dna / ID: 3 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:   Escherichia virus M13 (ウイルス) Escherichia virus M13 (ウイルス) |
| 分子量 | 理論値: 14.86049 KDa |
| 配列 | 文字列: (DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT) (DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT) (DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT) (DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT) (DT) (DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT) (DT) |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.7 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 6 構成要素:
詳細: The LiRecT protein was mixed at 37C with two oligonucleotides added sequentially, and placed on ice for 90 min. Then immediately prior to vitrification, 1 ul of 1.5 mM n-dodecyl-beta- ...詳細: The LiRecT protein was mixed at 37C with two oligonucleotides added sequentially, and placed on ice for 90 min. Then immediately prior to vitrification, 1 ul of 1.5 mM n-dodecyl-beta-maltoside (Anatrace) was added (0.5 CMC). | ||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 60 sec. / 前処理 - 雰囲気: AIR / 詳細: 20 mA with Pelco easiGlow | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV / 詳細: 1.5 second blot time. Ted Pella 595 filter paper.. | ||||||||||||
| 詳細 | This sample was monodisperse |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 3.5 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 81000 Bright-field microscopy / 最大 デフォーカス(公称値): 3.5 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 81000 |
| 特殊光学系 | 球面収差補正装置: Cs corrector was used / エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) デジタル化 - サイズ - 横: 6000 pixel / デジタル化 - サイズ - 縦: 4000 pixel / 撮影したグリッド数: 1 / 実像数: 2038 / 平均露光時間: 2.7 sec. / 平均電子線量: 66.0 e/Å2 / 詳細: 36 fractions, 24.28 e-/A2/s |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 詳細 | Phenix Real Space Refinement included secondary restraints and 10-fold NCS constraints. |
|---|---|
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL |
| 得られたモデル |  PDB-7ub2: |
 ムービー
ムービー コントローラー
コントローラー




 Z
Z Y
Y X
X