+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of human Elp123 in complex with tRNA, desulpho-CoA, 5'-deoxyadenosine and methionine | |||||||||
 マップデータ マップデータ | deepEMhancer sharpened map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Elongator / tRNA modification / acetyl-CoA hydrolysis /  TRANSLATION (翻訳 (生物学)) TRANSLATION (翻訳 (生物学)) | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報phosphorylase kinase regulator activity / tRNA uridine(34) acetyltransferase activity / elongator holoenzyme complex / tRNA wobble base 5-methoxycarbonylmethyl-2-thiouridinylation / tRNA wobble uridine modification / regulation of receptor signaling pathway via JAK-STAT /  acetyltransferase activity / acetyltransferase activity /  転移酵素; アシル基を移すもの; アミノアシル基以外のアシル基を移すもの / transcription elongation factor complex / 転移酵素; アシル基を移すもの; アミノアシル基以外のアシル基を移すもの / transcription elongation factor complex /  central nervous system development ...phosphorylase kinase regulator activity / tRNA uridine(34) acetyltransferase activity / elongator holoenzyme complex / tRNA wobble base 5-methoxycarbonylmethyl-2-thiouridinylation / tRNA wobble uridine modification / regulation of receptor signaling pathway via JAK-STAT / central nervous system development ...phosphorylase kinase regulator activity / tRNA uridine(34) acetyltransferase activity / elongator holoenzyme complex / tRNA wobble base 5-methoxycarbonylmethyl-2-thiouridinylation / tRNA wobble uridine modification / regulation of receptor signaling pathway via JAK-STAT /  acetyltransferase activity / acetyltransferase activity /  転移酵素; アシル基を移すもの; アミノアシル基以外のアシル基を移すもの / transcription elongation factor complex / 転移酵素; アシル基を移すもの; アミノアシル基以外のアシル基を移すもの / transcription elongation factor complex /  central nervous system development / transcription elongation by RNA polymerase II / central nervous system development / transcription elongation by RNA polymerase II /  neuron migration / neuron migration /  : / : /  regulation of translation / 4 iron, 4 sulfur cluster binding / HATs acetylate histones / regulation of translation / 4 iron, 4 sulfur cluster binding / HATs acetylate histones /  tRNA binding / positive regulation of cell migration / tRNA binding / positive regulation of cell migration /  核小体 / regulation of transcription by RNA polymerase II / 核小体 / regulation of transcription by RNA polymerase II /  protein kinase binding / protein kinase binding /  核質 / 核質 /  metal ion binding / metal ion binding /  細胞核 / 細胞核 /  細胞質基質 / 細胞質基質 /  細胞質 細胞質類似検索 - 分子機能 | |||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
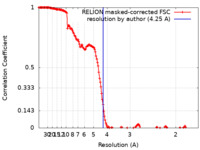
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 4.25 Å クライオ電子顕微鏡法 / 解像度: 4.25 Å | |||||||||
 データ登録者 データ登録者 | Abbassi N / Jaciuk M / Lin T-Y / Glatt S | |||||||||
| 資金援助 | European Union, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2024 ジャーナル: Nat Commun / 年: 2024タイトル: Cryo-EM structures of the human Elongator complex at work. 著者: Nour-El-Hana Abbassi / Marcin Jaciuk / David Scherf / Pauline Böhnert / Alexander Rau / Alexander Hammermeister / Michał Rawski / Paulina Indyka / Grzegorz Wazny / Andrzej Chramiec-Głąbik ...著者: Nour-El-Hana Abbassi / Marcin Jaciuk / David Scherf / Pauline Böhnert / Alexander Rau / Alexander Hammermeister / Michał Rawski / Paulina Indyka / Grzegorz Wazny / Andrzej Chramiec-Głąbik / Dominika Dobosz / Bozena Skupien-Rabian / Urszula Jankowska / Juri Rappsilber / Raffael Schaffrath / Ting-Yu Lin / Sebastian Glatt /    要旨: tRNA modifications affect ribosomal elongation speed and co-translational folding dynamics. The Elongator complex is responsible for introducing 5-carboxymethyl at wobble uridine bases (cmU) in ...tRNA modifications affect ribosomal elongation speed and co-translational folding dynamics. The Elongator complex is responsible for introducing 5-carboxymethyl at wobble uridine bases (cmU) in eukaryotic tRNAs. However, the structure and function of human Elongator remain poorly understood. In this study, we present a series of cryo-EM structures of human ELP123 in complex with tRNA and cofactors at four different stages of the reaction. The structures at resolutions of up to 2.9 Å together with complementary functional analyses reveal the molecular mechanism of the modification reaction. Our results show that tRNA binding exposes a universally conserved uridine at position 33 (U), which triggers acetyl-CoA hydrolysis. We identify a series of conserved residues that are crucial for the radical-based acetylation of U and profile the molecular effects of patient-derived mutations. Together, we provide the high-resolution view of human Elongator and reveal its detailed mechanism of action. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_17927.map.gz emd_17927.map.gz | 321.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-17927-v30.xml emd-17927-v30.xml emd-17927.xml emd-17927.xml | 26.2 KB 26.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_17927_fsc.xml emd_17927_fsc.xml | 16.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_17927.png emd_17927.png | 103.9 KB | ||
| マスクデータ |  emd_17927_msk_1.map emd_17927_msk_1.map | 371.3 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-17927.cif.gz emd-17927.cif.gz | 9 KB | ||
| その他 |  emd_17927_half_map_1.map.gz emd_17927_half_map_1.map.gz emd_17927_half_map_2.map.gz emd_17927_half_map_2.map.gz | 296.7 MB 296.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-17927 http://ftp.pdbj.org/pub/emdb/structures/EMD-17927 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-17927 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-17927 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_17927.map.gz / 形式: CCP4 / 大きさ: 371.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_17927.map.gz / 形式: CCP4 / 大きさ: 371.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | deepEMhancer sharpened map | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.86 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_17927_msk_1.map emd_17927_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_17927_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_17927_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Human Elp123 in complex with glutamine tRNA, desulpho-CoA, 5'-deo...
| 全体 | 名称: Human Elp123 in complex with glutamine tRNA, desulpho-CoA, 5'-deoxyadenosine and methionine |
|---|---|
| 要素 |
|
-超分子 #1: Human Elp123 in complex with glutamine tRNA, desulpho-CoA, 5'-deo...
| 超分子 | 名称: Human Elp123 in complex with glutamine tRNA, desulpho-CoA, 5'-deoxyadenosine and methionine タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 635 KDa |
-分子 #1: Elongator complex protein 1
| 分子 | 名称: Elongator complex protein 1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 150.427484 KDa |
| 組換発現 | 生物種:   Spodoptera frugiperda (ツマジロクサヨトウ) Spodoptera frugiperda (ツマジロクサヨトウ) |
| 配列 | 文字列: MRNLKLFRTL EFRDIQGPGN PQCFSLRTEQ GTVLIGSEHG LIEVDPVSRE VKNEVSLVAE GFLPEDGSGR IVGVQDLLDQ ESVCVATAS GDVILCSLST QQLECVGSVA SGISVMSWSP DQELVLLATG QQTLIMMTKD FEPILEQQIH QDDFGESKFI T VGWGRKET ...文字列: MRNLKLFRTL EFRDIQGPGN PQCFSLRTEQ GTVLIGSEHG LIEVDPVSRE VKNEVSLVAE GFLPEDGSGR IVGVQDLLDQ ESVCVATAS GDVILCSLST QQLECVGSVA SGISVMSWSP DQELVLLATG QQTLIMMTKD FEPILEQQIH QDDFGESKFI T VGWGRKET QFHGSEGRQA AFQMQMHESA LPWDDHRPQV TWRGDGQFFA VSVVCPETGA RKVRVWNREF ALQSTSEPVA GL GPALAWK PSGSLIASTQ DKPNQQDIVF FEKNGLLHGH FTLPFLKDEV KVNDLLWNAD SSVLAVWLED LQREESSIPK TCV QLWTVG NYHWYLKQSL SFSTCGKSKI VSLMWDPVTP YRLHVLCQGW HYLAYDWHWT TDRSVGDNSS DLSNVAVIDG NRVL VTVFR QTVVPPPMCT YQLLFPHPVN QVTFLAHPQK SNDLAVLDAS NQISVYKCGD CPSADPTVKL GAVGGSGFKV CLRTP HLEK RYKIQFENNE DQDVNPLKLG LLTWIEEDVF LAVSHSEFSP RSVIHHLTAA SSEMDEEHGQ LNVSSSAAVD GVIISL CCN SKTKSVVLQL ADGQIFKYLW ESPSLAIKPW KNSGGFPVRF PYPCTQTELA MIGEEECVLG LTDRCRFFIN DIEVASN IT SFAVYDEFLL LTTHSHTCQC FCLRDASFKT LQAGLSSNHV SHGEVLRKVE RGSRIVTVVP QDTKLVLQMP RGNLEVVH H RALVLAQIRK WLDKLMFKEA FECMRKLRIN LNLIYDHNPK VFLGNVETFI KQIDSVNHIN LFFTELKEED VTKTMYPAP VTSSVYLSRD PDGNKIDLVC DAMRAVMESI NPHKYCLSIL TSHVKKTTPE LEIVLQKVHE LQGNAPSDPD AVSAEEALKY LLHLVDVNE LYDHSLGTYD FDLVLMVAEK SQKDPKEYLP FLNTLKKMET NYQRFTIDKY LKRYEKAIGH LSKCGPEYFP E CLNLIKDK NLYNEALKLY SPSSQQYQDI SIAYGEHLMQ EHMYEPAGLM FARCGAHEKA LSAFLTCGNW KQALCVAAQL NF TKDQLVG LGRTLAGKLV EQRKHIDAAM VLEECAQDYE EAVLLLLEGA AWEEALRLVY KYNRLDIIET NVKPSILEAQ KNY MAFLDS QTATFSRHKK RLLVVRELKE QAQQAGLDDE VPHGQESDLF SETSSVVSGS EMSGKYSHSN SRISARSSKN RRKA ERKKH SLKEGSPLED LALLEALSEV VQNTENLKDE VYHILKVLFL FEFDEQGREL QKAFEDTLQL MERSLPEIWT LTYQQ NSAT PVLGPNSTAN SIMASYQQQK TSVPVLDAEL FIPPKINRRT QWKLSLLD UniProtKB: Elongator complex protein 1 |
-分子 #2: Elongator complex protein 2
| 分子 | 名称: Elongator complex protein 2 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 92.597766 KDa |
| 組換発現 | 生物種:   Spodoptera frugiperda (ツマジロクサヨトウ) Spodoptera frugiperda (ツマジロクサヨトウ) |
| 配列 | 文字列: MVAPVLETSH VFCCPNRVRG VLNWSSGPRG LLAFGTSCSV VLYDPLKRVV VTNLNGHTAR VNCIQWICKQ DGSPSTELVS GGSDNQVIH WEIEDNQLLK AVHLQGHEGP VYAVHAVYQR RTSDPALCTL IVSAAADSAV RLWSKKGPEV MCLQTLNFGN G FALALCLS ...文字列: MVAPVLETSH VFCCPNRVRG VLNWSSGPRG LLAFGTSCSV VLYDPLKRVV VTNLNGHTAR VNCIQWICKQ DGSPSTELVS GGSDNQVIH WEIEDNQLLK AVHLQGHEGP VYAVHAVYQR RTSDPALCTL IVSAAADSAV RLWSKKGPEV MCLQTLNFGN G FALALCLS FLPNTDVPIL ACGNDDCRIH IFAQQNDQFQ KVLSLCGHED WIRGVEWAAF GRDLFLASCS QDCLIRIWKL YI KSTSLET QDDDNIRLKE NTFTIENESV KIAFAVTLET VLAGHENWVN AVHWQPVFYK DGVLQQPVRL LSASMDKTMI LWA PDEESG VWLEQVRVGE VGGNTLGFYD CQFNEDGSMI IAHAFHGALH LWKQNTVNPR EWTPEIVISG HFDGVQDLVW DPEG EFIIT VGTDQTTRLF APWKRKDQSQ VTWHEIARPQ IHGYDLKCLA MINRFQFVSG ADEKVLRVFS APRNFVENFC AITGQ SLNH VLCNQDSDLP EGATVPALGL SNKAVFQGDI ASQPSDEEEL LTSTGFEYQQ VAFQPSILTE PPTEDHLLQN TLWPEV QKL YGHGYEIFCV TCNSSKTLLA SACKAAKKEH AAIILWNTTS WKQVQNLVFH SLTVTQMAFS PNEKFLLAVS RDRTWSL WK KQDTISPEFE PVFSLFAFTN KITSVHSRII WSCDWSPDSK YFFTGSRDKK VVVWGECDST DDCIEHNIGP CSSVLDVG G AVTAVSVCPV LHPSQRYVVA VGLECGKICL YTWKKTDQVP EINDWTHCVE TSQSQSHTLA IRKLCWKNCS GKTEQKEAE GAEWLHFASC GEDHTVKIHR VNKCAL UniProtKB: Elongator complex protein 2 |
-分子 #3: Elongator complex protein 3
| 分子 | 名称: Elongator complex protein 3 / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO EC番号:  転移酵素; アシル基を移すもの; アミノアシル基以外のアシル基を移すもの 転移酵素; アシル基を移すもの; アミノアシル基以外のアシル基を移すもの |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 65.740539 KDa |
| 組換発現 | 生物種:   Spodoptera frugiperda (ツマジロクサヨトウ) Spodoptera frugiperda (ツマジロクサヨトウ) |
| 配列 | 文字列: MRQKRKGDLS PAELMMLTIG DVIKQLIEAH EQGKDIDLNK VKTKTAAKYG LSAQPRLVDI IAAVPPQYRK VLMPKLKAKP IRTASGIAV VAVMCKPHRC PHISFTGNIC VYCPGGPDSD FEYSTQSYTG YEPTSMRAIR ARYDPFLQTR HRIEQLKQLG H SVDKVEFI ...文字列: MRQKRKGDLS PAELMMLTIG DVIKQLIEAH EQGKDIDLNK VKTKTAAKYG LSAQPRLVDI IAAVPPQYRK VLMPKLKAKP IRTASGIAV VAVMCKPHRC PHISFTGNIC VYCPGGPDSD FEYSTQSYTG YEPTSMRAIR ARYDPFLQTR HRIEQLKQLG H SVDKVEFI VMGGTFMALP EEYRDYFIRN LHDALSGHTS NNIYEAVKYS ERSLTKCIGI TIETRPDYCM KRHLSDMLTY GC TRLEIGV QSVYEDVARD TNRGHTVKAV CESFHLAKDS GFKVVAHMMP DLPNVGLERD IEQFTEFFEN PAFRPDGLKL YPT LVIRGT GLYELWKSGR YKSYSPSDLV ELVARILALV PPWTRVYRVQ RDIPMPLVSS GVEHGNLREL ALARMKDLGI QCRD VRTRE VGIQEIHHKV RPYQVELVRR DYVANGGWET FLSYEDPDQD ILIGLLRLRK CSEETFRFEL GGGVSIVREL HVYGS VVPV SSRDPTKFQH QGFGMLLMEE AERIAREEHG SGKIAVISGV GTRNYYRKIG YRLQGPYMVK MLKGLEGSAW SHPQFE KGG GSGGGSGGSA WSHPQFEK UniProtKB: Elongator complex protein 3 |
-分子 #4: tRNA Gln
| 分子 | 名称: tRNA Gln / タイプ: rna / ID: 4 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 24.047236 KDa |
| 配列 | 文字列: GGCCCCAUGG UGUAAUGGUU AGCACUCUGG ACUUUGAAUC CAGCGAUCCG AGUUCAAAUC UCGGUGGGAC CUCCA |
-分子 #5: DESULFO-COENZYME A
| 分子 | 名称: DESULFO-COENZYME A / タイプ: ligand / ID: 5 / コピー数: 1 / 式: DCA |
|---|---|
| 分子量 | 理論値: 735.469 Da |
| Chemical component information |  ChemComp-DCA: |
-分子 #6: IRON/SULFUR CLUSTER
| 分子 | 名称: IRON/SULFUR CLUSTER / タイプ: ligand / ID: 6 / コピー数: 1 / 式: SF4 |
|---|---|
| 分子量 | 理論値: 351.64 Da |
| Chemical component information |  ChemComp-FS1: |
-分子 #7: 5'-DEOXYADENOSINE
| 分子 | 名称: 5'-DEOXYADENOSINE / タイプ: ligand / ID: 7 / コピー数: 1 / 式: 5AD |
|---|---|
| 分子量 | 理論値: 251.242 Da |
| Chemical component information |  ChemComp-5AD: |
-分子 #8: METHIONINE
| 分子 | 名称: METHIONINE / タイプ: ligand / ID: 8 / コピー数: 1 / 式: MET |
|---|---|
| 分子量 | 理論値: 149.211 Da |
| Chemical component information |  ChemComp-MET: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.6 mg/mL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| |||||||||||||||
| グリッド | モデル: Quantifoil R2/1 / 材質: COPPER / メッシュ: 200 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 60 sec. / 前処理 - 雰囲気: AIR / 詳細: 8 mA | |||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV / 詳細: 15 s wait time, blot force 5, 5 s blot time. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.1 µm / 最小 デフォーカス(公称値): 0.9 µm / 倍率(公称値): 105000 Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.1 µm / 最小 デフォーカス(公称値): 0.9 µm / 倍率(公称値): 105000 |
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| ソフトウェア | 名称: EPU (ver. 2.10.0.5REL) |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) デジタル化 - サイズ - 横: 5760 pixel / デジタル化 - サイズ - 縦: 4092 pixel / 実像数: 20720 / 平均電子線量: 41.22 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | Chain - Source name: Other / Chain - Initial model type: in silico model / 詳細: Elp123-tRNA-ACO structure from the same study |
|---|---|
| ソフトウェア | 名称: UCSF ChimeraX |
| 得られたモデル |  PDB-8pu0: |
 ムービー
ムービー コントローラー
コントローラー












 Z
Z Y
Y X
X