+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
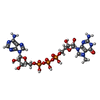
| タイトル | Early transcription elongation state of influenza A/H7N9 polymerase backtracked due to double incoproation of nucleotide analogue T1106 and with singly incoporated T1106 at the +1 position | |||||||||
 マップデータ マップデータ | LocScale filtered | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 キャップスナッチング / viral transcription / symbiont-mediated suppression of host mRNA transcription via inhibition of RNA polymerase II activity / host cell mitochondrion / 7-methylguanosine mRNA capping / キャップスナッチング / viral transcription / symbiont-mediated suppression of host mRNA transcription via inhibition of RNA polymerase II activity / host cell mitochondrion / 7-methylguanosine mRNA capping /  virion component / virion component /  endonuclease activity / host cell cytoplasm / endonuclease activity / host cell cytoplasm /  加水分解酵素; エステル加水分解酵素 / 加水分解酵素; エステル加水分解酵素 /  RNA依存性RNAポリメラーゼ ... RNA依存性RNAポリメラーゼ ... キャップスナッチング / viral transcription / symbiont-mediated suppression of host mRNA transcription via inhibition of RNA polymerase II activity / host cell mitochondrion / 7-methylguanosine mRNA capping / キャップスナッチング / viral transcription / symbiont-mediated suppression of host mRNA transcription via inhibition of RNA polymerase II activity / host cell mitochondrion / 7-methylguanosine mRNA capping /  virion component / virion component /  endonuclease activity / host cell cytoplasm / endonuclease activity / host cell cytoplasm /  加水分解酵素; エステル加水分解酵素 / 加水分解酵素; エステル加水分解酵素 /  RNA依存性RNAポリメラーゼ / viral RNA genome replication / RNA依存性RNAポリメラーゼ / viral RNA genome replication /  RNA-dependent RNA polymerase activity / RNA-dependent RNA polymerase activity /  nucleotide binding / DNA-templated transcription / host cell nucleus / nucleotide binding / DNA-templated transcription / host cell nucleus /  RNA binding / RNA binding /  metal ion binding / metal ion binding /  細胞質 細胞質類似検索 - 分子機能 | |||||||||
| 生物種 |   Influenza A virus (A/Zhejiang/DTID-ZJU01/2013(H7N9)) (A型インフルエンザウイルス) / Influenza A virus (A/Zhejiang/DTID-ZJU01/2013(H7N9)) (A型インフルエンザウイルス) /    Influenza A virus (A型インフルエンザウイルス) Influenza A virus (A型インフルエンザウイルス) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 2.51 Å クライオ電子顕微鏡法 / 解像度: 2.51 Å | |||||||||
 データ登録者 データ登録者 | Cusack S / Kouba T | |||||||||
| 資金援助 |  フランス, 1件 フランス, 1件
| |||||||||
 引用 引用 |  ジャーナル: Cell Rep / 年: 2023 ジャーナル: Cell Rep / 年: 2023タイトル: Direct observation of backtracking by influenza A and B polymerases upon consecutive incorporation of the nucleoside analog T1106. 著者: Tomas Kouba / Anna Dubankova / Petra Drncova / Elisa Donati / Pietro Vidossich / Valentina Speranzini / Alex Pflug / Johanna Huchting / Chris Meier / Marco De Vivo / Stephen Cusack /    要旨: The antiviral pseudo-base T705 and its de-fluoro analog T1106 mimic adenine or guanine and can be competitively incorporated into nascent RNA by viral RNA-dependent RNA polymerases. Although ...The antiviral pseudo-base T705 and its de-fluoro analog T1106 mimic adenine or guanine and can be competitively incorporated into nascent RNA by viral RNA-dependent RNA polymerases. Although dispersed, single pseudo-base incorporation is mutagenic, consecutive incorporation causes polymerase stalling and chain termination. Using a template encoding single and then consecutive T1106 incorporation four nucleotides later, we obtained a cryogenic electron microscopy structure of stalled influenza A/H7N9 polymerase. This shows that the entire product-template duplex backtracks by 5 nt, bringing the singly incorporated T1106 to the +1 position, where it forms an unexpected T1106:U wobble base pair. Similar structures show that influenza B polymerase also backtracks after consecutive T1106 incorporation, regardless of whether prior single incorporation has occurred. These results give insight into the unusual mechanism of chain termination by pyrazinecarboxamide base analogs. Consecutive incorporation destabilizes the proximal end of the product-template duplex, promoting irreversible backtracking to a more energetically favorable overall configuration. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_14222.map.gz emd_14222.map.gz | 159.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-14222-v30.xml emd-14222-v30.xml emd-14222.xml emd-14222.xml | 23.4 KB 23.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_14222_fsc.xml emd_14222_fsc.xml | 15.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_14222.png emd_14222.png | 119.9 KB | ||
| その他 |  emd_14222_additional_1.map.gz emd_14222_additional_1.map.gz emd_14222_half_map_1.map.gz emd_14222_half_map_1.map.gz emd_14222_half_map_2.map.gz emd_14222_half_map_2.map.gz | 302.4 MB 259.5 MB 260.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-14222 http://ftp.pdbj.org/pub/emdb/structures/EMD-14222 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14222 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-14222 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7r0eMC  7qtlC  7r1fC  7zplC  7zpmC  8bdrC  8be0C  8bekC  8bf5C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_14222.map.gz / 形式: CCP4 / 大きさ: 325 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_14222.map.gz / 形式: CCP4 / 大きさ: 325 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | LocScale filtered | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.827 Å | ||||||||||||||||||||
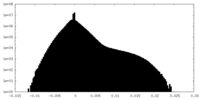
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: RELION post-processed
| ファイル | emd_14222_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | RELION post-processed | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
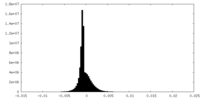
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_14222_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
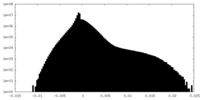
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_14222_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
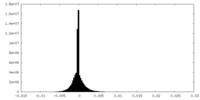
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
+全体 : A/H7N9-Backtracked-T/U+1
+超分子 #1: A/H7N9-Backtracked-T/U+1
+分子 #1: Polymerase acidic protein
+分子 #2: RNA-directed RNA polymerase catalytic subunit
+分子 #3: Polymerase basic protein 2
+分子 #4: mRNA
+分子 #5: 3' vRNA
+分子 #6: 5' vRNA
+分子 #7: MAGNESIUM ION
+分子 #8: P1-7-METHYLGUANOSINE-P3-ADENOSINE-5',5'-TRIPHOSPHATE
+分子 #9: water
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 1.8 µm / 最小 デフォーカス(公称値): 0.5 µm Bright-field microscopy / 最大 デフォーカス(公称値): 1.8 µm / 最小 デフォーカス(公称値): 0.5 µm |
| 撮影 | フィルム・検出器のモデル: GATAN K2 QUANTUM (4k x 4k) 平均電子線量: 33.4 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL |
|---|---|
| 得られたモデル |  PDB-7r0e: |
 ムービー
ムービー コントローラー
コントローラー






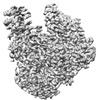
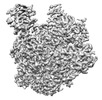
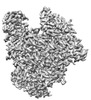

 Z
Z Y
Y X
X