+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-12298 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Ovine (b0,+AT-rBAT)2 hetero-tetramer, consensus refinement, C2-symmetry | |||||||||
 マップデータ マップデータ | Postprocessed map | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |   Ovis aries (ヒツジ) Ovis aries (ヒツジ) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 2.83 Å クライオ電子顕微鏡法 / 解像度: 2.83 Å | |||||||||
 データ登録者 データ登録者 | Lee Y / Kuehlbrandt W | |||||||||
| 資金援助 |  ドイツ, 2件 ドイツ, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2022 ジャーナル: Nat Commun / 年: 2022タイトル: Ca-mediated higher-order assembly of heterodimers in amino acid transport system b biogenesis and cystinuria. 著者: Yongchan Lee / Pattama Wiriyasermkul / Pornparn Kongpracha / Satomi Moriyama / Deryck J Mills / Werner Kühlbrandt / Shushi Nagamori /   要旨: Cystinuria is a genetic disorder characterized by overexcretion of dibasic amino acids and cystine, causing recurrent kidney stones and kidney failure. Mutations of the regulatory glycoprotein rBAT ...Cystinuria is a genetic disorder characterized by overexcretion of dibasic amino acids and cystine, causing recurrent kidney stones and kidney failure. Mutations of the regulatory glycoprotein rBAT and the amino acid transporter bAT, which constitute system b, are linked to type I and non-type I cystinuria respectively and they exhibit distinct phenotypes due to protein trafficking defects or catalytic inactivation. Here, using electron cryo-microscopy and biochemistry, we discover that Ca mediates higher-order assembly of system b. Ca stabilizes the interface between two rBAT molecules, leading to super-dimerization of bAT-rBAT, which in turn facilitates N-glycan maturation and protein trafficking. A cystinuria mutant T216M and mutations of the Ca site of rBAT cause the loss of higher-order assemblies, resulting in protein trapping at the ER and the loss of function. These results provide the molecular basis of system b biogenesis and type I cystinuria and serve as a guide to develop new therapeutic strategies against it. More broadly, our findings reveal an unprecedented link between transporter oligomeric assembly and protein-trafficking diseases. #1:  ジャーナル: Biorxiv / 年: 2021 ジャーナル: Biorxiv / 年: 2021タイトル: Ca 2+ -mediated higher-order assembly of b 0,+ AT-rBAT is a key step for system b 0,+ biogenesis and cystinuria 著者: Lee Y / Wiriyasermkul P / Moriyama S / Mills DJ / Kuhlbrandt W / Nagamori S | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_12298.map.gz emd_12298.map.gz | 14.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-12298-v30.xml emd-12298-v30.xml emd-12298.xml emd-12298.xml | 18.3 KB 18.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_12298_fsc.xml emd_12298_fsc.xml | 11.3 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_12298.png emd_12298.png | 165.2 KB | ||
| マスクデータ |  emd_12298_msk_1.map emd_12298_msk_1.map | 125 MB |  マスクマップ マスクマップ | |
| その他 |  emd_12298_half_map_1.map.gz emd_12298_half_map_1.map.gz emd_12298_half_map_2.map.gz emd_12298_half_map_2.map.gz | 96.8 MB 97 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-12298 http://ftp.pdbj.org/pub/emdb/structures/EMD-12298 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-12298 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-12298 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_12298.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_12298.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Postprocessed map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.09856 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_12298_msk_1.map emd_12298_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map 1
| ファイル | emd_12298_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map 2
| ファイル | emd_12298_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : (b0,+AT-rBAT) hetero-tetramer
| 全体 | 名称: (b0,+AT-rBAT) hetero-tetramer |
|---|---|
| 要素 |
|
-超分子 #1: (b0,+AT-rBAT) hetero-tetramer
| 超分子 | 名称: (b0,+AT-rBAT) hetero-tetramer / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:   Ovis aries (ヒツジ) Ovis aries (ヒツジ) |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 270 kDa/nm |
-分子 #1: B(0,+)-type amino acid transporter 1
| 分子 | 名称: B(0,+)-type amino acid transporter 1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Ovis aries (ヒツジ) Ovis aries (ヒツジ) |
| 分子量 | 理論値: 56.318047 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MEETSLRKRR GDEKSIQSSE PKTTSLQKEL GLFSGTCIIV GTIIGSGIFI SPKSVLSNME AVGPCLIIWA MCGVLATLGA LCFAELGTM ITKSGGEYPY LMEAFGPIPA YLFSWSSLFV IKPSSFAIIC LSFSEYVCAP FYSGCSPPQV VIKTLAAAAI L LISTVNAL ...文字列: MEETSLRKRR GDEKSIQSSE PKTTSLQKEL GLFSGTCIIV GTIIGSGIFI SPKSVLSNME AVGPCLIIWA MCGVLATLGA LCFAELGTM ITKSGGEYPY LMEAFGPIPA YLFSWSSLFV IKPSSFAIIC LSFSEYVCAP FYSGCSPPQV VIKTLAAAAI L LISTVNAL SVRLGSYVQN VFTAAKMVIV VIIIISGLVL LAQGNTRNFE NSFEGASLSV GSISLALYNG LWAYDGWNQL NY ITEELRN PFRNLPLAII IGIPLVTGCY ILMNVSYFTV MTATELLQSQ AVAVTFGDRV LYPASWIVPL FVAFSTIGAA NGS CFTAGR LVFVAGREGH MLKVLSYISV KRLTPAPAIM FHSMIAIIYI IPGDINSLVN YFSFAAWLFY GLTITGLIVM RFTR KELKR PIKVPIFIPI LVTLLSVFLV LAPIISAPAW EYLYCVLFML SGLVFYFLFV HYKFGWAQKI SKPLTMHLQM LMEVV PPEE APEWSHPQFE KGGGSGGGSG GSAWSHPQFE K |
-分子 #2: neutral and basic amino acid transport protein rBAT
| 分子 | 名称: neutral and basic amino acid transport protein rBAT / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Ovis aries (ヒツジ) Ovis aries (ヒツジ) |
| 分子量 | 理論値: 78.754281 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: GSAEDKSKRD SIGLNAKEGQ TNNGFVQNED ILETDLDPSS PAAGPQHNTV DILGPGEPDV KDVRPYAGMP KEVLFQFSGQ ARYRIPREV LFWLTVASVL LLIAATIAII AISPKCLDWW QAGPMYQIYP RSFRDSNKDG DGDLKGIQDK LDYITTLNIK T VWITSFYK ...文字列: GSAEDKSKRD SIGLNAKEGQ TNNGFVQNED ILETDLDPSS PAAGPQHNTV DILGPGEPDV KDVRPYAGMP KEVLFQFSGQ ARYRIPREV LFWLTVASVL LLIAATIAII AISPKCLDWW QAGPMYQIYP RSFRDSNKDG DGDLKGIQDK LDYITTLNIK T VWITSFYK SSLKDFRHAV EDFQEIDPIF GTMKDFENLV AAIHDKGLKL IIDFIPNHTS DKHAWFQWSR NRTGKYTDYY IW HDCNYEN GTTIPPNNWL SVYGNSSWHF DEVRKQCYFH QFMKEQPDLN FRNPDVQEEI KEIIQFWLSK GVDGFSFNAL QYL LEAKHL RDEAQVNKTQ IPDTVTHYSQ LHHDFTTTQV GMHDIVRSFR QTMNQYSREP GRYRFMGTEA HGESITETMV YYGL PFIQE ADFPFNSYLS KLDKPSGNSV SEVITSWLEN MPEGKWPNWM TGGPDNVRLT SRLGEKYVNI MNMLVFTLPG TPITY YGEE IGMRNILAAN LNENYDTGTL FSKSPMQWDN SSNAGFSEGN HTWLPTSSDY HTVNVDVQKT QPRSALKLYQ ELSLLH ANE LLLSRGWFCY LRNDNHSIMY TRELDGINKV FLMVLNFGES SLLNLKEMIS NIPTRVRIRL STSSAYSGRE VDTHAVT LA SGEGLILEYN TGNLLHRQTA FKDRCFVSNR ACYSRVLNIL YSLC |
-分子 #4: CHOLESTEROL
| 分子 | 名称: CHOLESTEROL / タイプ: ligand / ID: 4 / コピー数: 2 / 式: CLR |
|---|---|
| 分子量 | 理論値: 386.654 Da |
| Chemical component information |  ChemComp-CLR: |
-分子 #5: 1-palmitoyl-2-oleoyl-sn-glycero-3-phosphocholine
| 分子 | 名称: 1-palmitoyl-2-oleoyl-sn-glycero-3-phosphocholine / タイプ: ligand / ID: 5 / コピー数: 1 / 式: LBN |
|---|---|
| 分子量 | 理論値: 760.076 Da |
| Chemical component information |  ChemComp-LBN: |
-分子 #6: CALCIUM ION
| 分子 | 名称: CALCIUM ION / タイプ: ligand / ID: 6 / コピー数: 1 / 式: CA |
|---|---|
| 分子量 | 理論値: 40.078 Da |
-分子 #7: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 7 / コピー数: 4 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 50.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
|---|---|
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 再構成 | 想定した対称性 - 点群: C2 (2回回転対称 ) / 解像度のタイプ: BY AUTHOR / 解像度: 2.83 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 581697 ) / 解像度のタイプ: BY AUTHOR / 解像度: 2.83 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 581697 |
FSC曲線 (解像度の算出) | 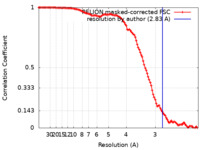 |
-原子モデル構築 1
| 精密化 | プロトコル: RIGID BODY FIT |
|---|---|
| 得られたモデル |  PDB-7nf8: |
 ムービー
ムービー コントローラー
コントローラー


















 Z
Z Y
Y X
X