+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-10590 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of PfMyoA decorated Plasmodium Act1 filament | |||||||||
 マップデータ マップデータ | Global B-factor sharpening | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報plastid inheritance /  分裂 (生物学) / pellicle / glideosome / inner membrane pellicle complex / Platelet degranulation / symbiont-medited actin polymerization-dependent cell-to-cell migration in host / HSP90 chaperone cycle for steroid hormone receptors (SHR) in the presence of ligand / Neutrophil degranulation / entry into host cell by a symbiont-containing vacuole ...plastid inheritance / 分裂 (生物学) / pellicle / glideosome / inner membrane pellicle complex / Platelet degranulation / symbiont-medited actin polymerization-dependent cell-to-cell migration in host / HSP90 chaperone cycle for steroid hormone receptors (SHR) in the presence of ligand / Neutrophil degranulation / entry into host cell by a symbiont-containing vacuole ...plastid inheritance /  分裂 (生物学) / pellicle / glideosome / inner membrane pellicle complex / Platelet degranulation / symbiont-medited actin polymerization-dependent cell-to-cell migration in host / HSP90 chaperone cycle for steroid hormone receptors (SHR) in the presence of ligand / Neutrophil degranulation / entry into host cell by a symbiont-containing vacuole / vesicle transport along actin filament / 分裂 (生物学) / pellicle / glideosome / inner membrane pellicle complex / Platelet degranulation / symbiont-medited actin polymerization-dependent cell-to-cell migration in host / HSP90 chaperone cycle for steroid hormone receptors (SHR) in the presence of ligand / Neutrophil degranulation / entry into host cell by a symbiont-containing vacuole / vesicle transport along actin filament /  myosin complex / microfilament motor activity / cytoskeletal motor activity / cytoskeleton organization / myosin complex / microfilament motor activity / cytoskeletal motor activity / cytoskeleton organization /  マイクロフィラメント / actin filament organization / マイクロフィラメント / actin filament organization /  加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与 / structural constituent of cytoskeleton / 加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与 / structural constituent of cytoskeleton /  actin filament binding / actin filament binding /  マイクロフィラメント / マイクロフィラメント /  actin binding / vesicle / actin binding / vesicle /  ATP hydrolysis activity / ATP hydrolysis activity /  ATP binding / ATP binding /  生体膜 / 生体膜 /  細胞核 / 細胞核 /  細胞膜 / 細胞膜 /  細胞質 細胞質類似検索 - 分子機能 | |||||||||
| 生物種 |   Plasmodium falciparum 3D7 (マラリア病原虫) Plasmodium falciparum 3D7 (マラリア病原虫) | |||||||||
| 手法 | らせん対称体再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.1 Å クライオ電子顕微鏡法 / 解像度: 3.1 Å | |||||||||
 データ登録者 データ登録者 | Vahokoski J / Calder LJ / Lopez AJ / Rosenthal PB / Kursula I | |||||||||
| 資金援助 |  ノルウェー, ノルウェー,  英国, 2件 英国, 2件
| |||||||||
 引用 引用 |  ジャーナル: PLoS Pathog / 年: 2022 ジャーナル: PLoS Pathog / 年: 2022タイトル: High-resolution structures of malaria parasite actomyosin and actin filaments. 著者: Juha Vahokoski / Lesley J Calder / Andrea J Lopez / Justin E Molloy / Inari Kursula / Peter B Rosenthal /    要旨: Malaria is responsible for half a million deaths annually and poses a huge economic burden on the developing world. The mosquito-borne parasites (Plasmodium spp.) that cause the disease depend upon ...Malaria is responsible for half a million deaths annually and poses a huge economic burden on the developing world. The mosquito-borne parasites (Plasmodium spp.) that cause the disease depend upon an unconventional actomyosin motor for both gliding motility and host cell invasion. The motor system, often referred to as the glideosome complex, remains to be understood in molecular terms and is an attractive target for new drugs that might block the infection pathway. Here, we present the high-resolution structure of the actomyosin motor complex from Plasmodium falciparum. The complex includes the malaria parasite actin filament (PfAct1) complexed with the class XIV myosin motor (PfMyoA) and its two associated light-chains. The high-resolution core structure reveals the PfAct1:PfMyoA interface in atomic detail, while at lower-resolution, we visualize the PfMyoA light-chain binding region, including the essential light chain (PfELC) and the myosin tail interacting protein (PfMTIP). Finally, we report a bare PfAct1 filament structure at improved resolution. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_10590.map.gz emd_10590.map.gz | 44.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-10590-v30.xml emd-10590-v30.xml emd-10590.xml emd-10590.xml | 19.9 KB 19.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_10590.png emd_10590.png | 208.6 KB | ||
| マスクデータ |  emd_10590_msk_1.map emd_10590_msk_1.map | 512 MB |  マスクマップ マスクマップ | |
| その他 |  emd_10590_additional_1.map.gz emd_10590_additional_1.map.gz emd_10590_half_map_1.map.gz emd_10590_half_map_1.map.gz emd_10590_half_map_2.map.gz emd_10590_half_map_2.map.gz | 408 MB 409.3 MB 409.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-10590 http://ftp.pdbj.org/pub/emdb/structures/EMD-10590 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10590 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-10590 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_10590.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_10590.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Global B-factor sharpening | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.09 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_10590_msk_1.map emd_10590_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
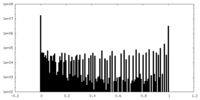
| 密度ヒストグラム |
-追加マップ: #1
| ファイル | emd_10590_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
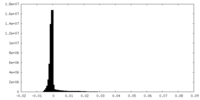
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_10590_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
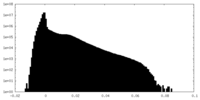
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_10590_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
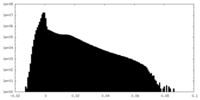
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : PfMyoA decorated PfAct1 filament
| 全体 | 名称: PfMyoA decorated PfAct1 filament |
|---|---|
| 要素 |
|
-超分子 #1: PfMyoA decorated PfAct1 filament
| 超分子 | 名称: PfMyoA decorated PfAct1 filament / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:   Plasmodium falciparum 3D7 (マラリア病原虫) Plasmodium falciparum 3D7 (マラリア病原虫) |
| 組換発現 | 生物種:   Spodoptera frugiperda (ツマジロクサヨトウ) Spodoptera frugiperda (ツマジロクサヨトウ)組換細胞: Sf21 |
-分子 #1: Myosin-A
| 分子 | 名称: Myosin-A / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Plasmodium falciparum 3D7 (マラリア病原虫) Plasmodium falciparum 3D7 (マラリア病原虫) |
| 分子量 | 理論値: 92.675477 KDa |
| 組換発現 | 生物種:   Spodoptera frugiperda (ツマジロクサヨトウ) Spodoptera frugiperda (ツマジロクサヨトウ) |
| 配列 | 文字列: GQFAVTNEEI KTASKIVRRV (SEP)NVEAFDKSG SVFKGYQIWT DISPTIENDP NIMFVKCVVQ QGSKKEKLTV VQIDPP GTG TPYDIDPTHA WNCNSQVDPM SFGDIGLLNH TNIPCVLDFL KHRYLKNQIY TTAVPLIVAI NPYKDLGNTT NEWIRRY RD ...文字列: GQFAVTNEEI KTASKIVRRV (SEP)NVEAFDKSG SVFKGYQIWT DISPTIENDP NIMFVKCVVQ QGSKKEKLTV VQIDPP GTG TPYDIDPTHA WNCNSQVDPM SFGDIGLLNH TNIPCVLDFL KHRYLKNQIY TTAVPLIVAI NPYKDLGNTT NEWIRRY RD TADHTKLPPH VFTCAREALS NLHGVNKSQT IIVSGESGAG KTEATKQIMR YFASSKSGNM DLRIQTAIMA ANPVLEAF G NAKTIRNNNS SRFGRFMQLV ISHEGGIRYG SVVAFLLEKS RIITQDDNER SYHIFYQFLK GANSTMKSKF GLKGVTEYK LLNPNSTEVS GVDDVKDFEE VIESLKNMEL SESDIEVIFS IVAGILTLGN VRLIEKQEAG LSDAAAIMDE DMGVFNKACE LMYLDPELI KREILIKVTV AGGTKIEGRW NKNDAEVLKS SLCKAMYEKL FLWIIRHLNS RIEPEGGFKT FMGMLDIFGF E VFKNNSLE QLFINITNEM LQKNFVDIVF ERESKLYKDE GISTAELKYT SNKEVINVLC EKGKSVLSYL EDQCLAPGGT DE KFVSSCA TNLKENNKFT PAKVASNKNF IIQHTIGPIQ YCAESFLLKN KDVLRGDLVE VIKDSPNPIV QQLFEGQVIE KGK IAKGSL IGSQFLNQLT SLMNLINSTE PHFIRCIKPN ENKKPLEWCE PKILIQLHAL SILEALVLRQ LGYSYRRTFE EFLY QYKFV DIAAAEDSSV ENQNKCVNIL KLSGLSESMY KIGKSMVFLK QEGAKILTKI QREKLVEWEN CVSVIEAAIL KHKYK QKVN KNIPSLLRVQ AHIRKKMVAQ |
-分子 #2: Actin-1
| 分子 | 名称: Actin-1 / タイプ: protein_or_peptide / ID: 2 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Plasmodium falciparum 3D7 (マラリア病原虫) Plasmodium falciparum 3D7 (マラリア病原虫) |
| 分子量 | 理論値: 42.047676 KDa |
| 組換発現 | 生物種:   Spodoptera frugiperda (ツマジロクサヨトウ) Spodoptera frugiperda (ツマジロクサヨトウ) |
| 配列 | 文字列: GAMGEEDVQA LVVDNGSGNV KAGVAGDDAP RSVFPSIVGR PKNPGIMVGM EEKDAFVGDE AQTKRGILTL KYPIEHGIVT NWDDMEKIW HHTFYNELRA APEEHPVLLT EAPLNPKGNR ERMTQIMFES FNVPAMYVAI QAVLSLYSSG RTTGIVLDSG D GVSHTVPI ...文字列: GAMGEEDVQA LVVDNGSGNV KAGVAGDDAP RSVFPSIVGR PKNPGIMVGM EEKDAFVGDE AQTKRGILTL KYPIEHGIVT NWDDMEKIW HHTFYNELRA APEEHPVLLT EAPLNPKGNR ERMTQIMFES FNVPAMYVAI QAVLSLYSSG RTTGIVLDSG D GVSHTVPI YEGYALPHAI MRLDLAGRDL TEYLMKILHE RGYGFSTSAE KEIVRDIKEK LCYIALNFDE EMKTSEQSSD IE KSYELPD GNIITVGNER FRCPEALFQP SFLGKEAAGI HTTTFNSIKK CDVDIRKDLY GNIVLSGGTT MYEGIGERLT RDI TTLAPS TMKIKVVAPP ERKYSVWIGG SILSSLSTFQ QMWITKEEYD ESGPSIVHRK CF |
-分子 #3: ADENOSINE-5'-DIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-DIPHOSPHATE / タイプ: ligand / ID: 3 / コピー数: 4 / 式: ADP |
|---|---|
| 分子量 | 理論値: 427.201 Da |
| Chemical component information |  ChemComp-ADP: |
-分子 #4: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 4 / コピー数: 4 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-分子 #5: Jasplakinolide
| 分子 | 名称: Jasplakinolide / タイプ: ligand / ID: 5 / コピー数: 4 / 式: 9UE |
|---|---|
| 分子量 | 理論値: 709.67 Da |
| Chemical component information |  ChemComp-9UE: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy |
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: COUNTING / 平均電子線量: 49.2 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: OTHER / 詳細: a cylinder |
|---|---|
| 最終 角度割当 | タイプ: NOT APPLICABLE |
| 最終 再構成 | 想定した対称性 - らせんパラメータ - Δz: 28.3417 Å 想定した対称性 - らせんパラメータ - ΔΦ: -166.498 ° 想定した対称性 - らせんパラメータ - 軸対称性: C1 (非対称) 解像度のタイプ: BY AUTHOR / 解像度: 3.1 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: RELION (ver. 3.0 beta) / 使用した粒子像数: 239021 |
 ムービー
ムービー コントローラー
コントローラー















 Z
Z Y
Y X
X