+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-0062 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Transient state structure of CRISPR-Cpf1 (Cas12a). I2 conformation | |||||||||
 マップデータ マップデータ | Transition state CRISPR-Cpf1 (Cas12a)I2 conformation | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | CRISPR genome editing ribonucleoprotein complex /  HYDROLASE (加水分解酵素) HYDROLASE (加水分解酵素) | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 枯草菌リボヌクレアーゼ / 枯草菌リボヌクレアーゼ /  Bacillus subtilis ribonuclease activity / Bacillus subtilis ribonuclease activity /  deoxyribonuclease I / deoxyribonuclease I /  deoxyribonuclease I activity / defense response to virus / deoxyribonuclease I activity / defense response to virus /  lyase activity / lyase activity /  DNA binding / DNA binding /  RNA binding RNA binding類似検索 - 分子機能 | |||||||||
| 生物種 |   Francisella tularensis subsp. novicida U112 (バクテリア) / Francisella tularensis subsp. novicida U112 (バクテリア) /   Francisella tularensis subsp. novicida (strain U112) (バクテリア) Francisella tularensis subsp. novicida (strain U112) (バクテリア) | |||||||||
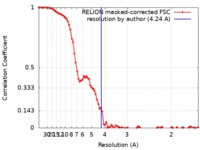
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 4.24 Å クライオ電子顕微鏡法 / 解像度: 4.24 Å | |||||||||
 データ登録者 データ登録者 | Montoya G / Mesa P | |||||||||
| 資金援助 |  デンマーク, 1件 デンマーク, 1件
| |||||||||
 引用 引用 |  ジャーナル: Cell / 年: 2018 ジャーナル: Cell / 年: 2018タイトル: Conformational Activation Promotes CRISPR-Cas12a Catalysis and Resetting of the Endonuclease Activity. 著者: Stefano Stella / Pablo Mesa / Johannes Thomsen / Bijoya Paul / Pablo Alcón / Simon B Jensen / Bhargav Saligram / Matias E Moses / Nikos S Hatzakis / Guillermo Montoya /  要旨: Cas12a, also known as Cpf1, is a type V-A CRISPR-Cas RNA-guided endonuclease that is used for genome editing based on its ability to generate specific dsDNA breaks. Here, we show cryo-EM structures ...Cas12a, also known as Cpf1, is a type V-A CRISPR-Cas RNA-guided endonuclease that is used for genome editing based on its ability to generate specific dsDNA breaks. Here, we show cryo-EM structures of intermediates of the cleavage reaction, thus visualizing three protein regions that sense the crRNA-DNA hybrid assembly triggering the catalytic activation of Cas12a. Single-molecule FRET provides the thermodynamics and kinetics of the conformational activation leading to phosphodiester bond hydrolysis. These findings illustrate why Cas12a cuts its target DNA and unleashes unspecific cleavage activity, degrading ssDNA molecules after activation. In addition, we show that other crRNAs are able to displace the R-loop inside the protein after target DNA cleavage, terminating indiscriminate ssDNA degradation. We propose a model whereby the conformational activation of the enzyme results in indiscriminate ssDNA cleavage. The displacement of the R-loop by a new crRNA molecule will reset Cas12a specificity, targeting new DNAs. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_0062.map.gz emd_0062.map.gz | 34.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-0062-v30.xml emd-0062-v30.xml emd-0062.xml emd-0062.xml | 14.8 KB 14.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_0062_fsc.xml emd_0062_fsc.xml | 8.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_0062.png emd_0062.png | 115 KB | ||
| Filedesc metadata |  emd-0062.cif.gz emd-0062.cif.gz | 6.6 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-0062 http://ftp.pdbj.org/pub/emdb/structures/EMD-0062 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0062 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0062 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_0062.map.gz / 形式: CCP4 / 大きさ: 52.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_0062.map.gz / 形式: CCP4 / 大きさ: 52.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Transition state CRISPR-Cpf1 (Cas12a)I2 conformation | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.832 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : TRansition state complex I2 conformation
| 全体 | 名称: TRansition state complex I2 conformation |
|---|---|
| 要素 |
|
-超分子 #1: TRansition state complex I2 conformation
| 超分子 | 名称: TRansition state complex I2 conformation / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 分子量 | 理論値: 180 KDa |
-超分子 #2: CRISPR-associated endonuclease Cas12a
| 超分子 | 名称: CRISPR-associated endonuclease Cas12a / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:   Francisella tularensis subsp. novicida U112 (バクテリア) Francisella tularensis subsp. novicida U112 (バクテリア) |
-超分子 #3: Nucleic acids
| 超分子 | 名称: Nucleic acids / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #2-#4 |
|---|---|
| 由来(天然) | 生物種:   Francisella tularensis subsp. novicida U112 (バクテリア) Francisella tularensis subsp. novicida U112 (バクテリア) |
-分子 #1: CRISPR-associated endonuclease Cas12a
| 分子 | 名称: CRISPR-associated endonuclease Cas12a / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO / EC番号:  deoxyribonuclease I deoxyribonuclease I |
|---|---|
| 由来(天然) | 生物種:   Francisella tularensis subsp. novicida (strain U112) (バクテリア) Francisella tularensis subsp. novicida (strain U112) (バクテリア)株: U112 |
| 分子量 | 理論値: 155.639828 KDa |
| 組換発現 | 生物種:   Escherichia coli BL21 (大腸菌) Escherichia coli BL21 (大腸菌) |
| 配列 | 文字列: MSIYQEFVNK YSLSKTLRFE LIPQGKTLEN IKARGLILDD EKRAKDYKKA KQIIDKYHQF FIEEILSSVC ISEDLLQNYS DVYFKLKKS DDDNLQKDFK SAKDTIKKQI SEYIKDSEKF KNLFNQNLID AKKGQESDLI LWLKQSKDNG IELFKANSDI T DIDEALEI ...文字列: MSIYQEFVNK YSLSKTLRFE LIPQGKTLEN IKARGLILDD EKRAKDYKKA KQIIDKYHQF FIEEILSSVC ISEDLLQNYS DVYFKLKKS DDDNLQKDFK SAKDTIKKQI SEYIKDSEKF KNLFNQNLID AKKGQESDLI LWLKQSKDNG IELFKANSDI T DIDEALEI IKSFKGWTTY FKGFHENRKN VYSSNDIPTS IIYRIVDDNL PKFLENKAKY ESLKDKAPEA INYEQIKKDL AE ELTFDID YKTSEVNQRV FSLDEVFEIA NFNNYLNQSG ITKFNTIIGG KFVNGENTKR KGINEYINLY SQQINDKTLK KYK MSVLFK QILSDTESKS FVIDKLEDDS DVVTTMQSFY EQIAAFKTVE EKSIKETLSL LFDDLKAQKL DLSKIYFKND KSLT DLSQQ VFDDYSVIGT AVLEYITQQI APKNLDNPSK KEQELIAKKT EKAKYLSLET IKLALEEFNK HRDIDKQCRF EEILA NFAA IPMIFDEIAQ NKDNLAQISI KYQNQGKKDL LQASAEDDVK AIKDLLDQTN NLLHKLKIFH ISQSEDKANI LDKDEH FYL VFEECYFELA NIVPLYNKIR NYITQKPYSD EKFKLNFENS TLANGWDKNK EPDNTAILFI KDDKYYLGVM NKKNNKI FD DKAIKENKGE GYKKIVYKLL PGANKMLPKV FFSAKSIKFY NPSEDILRIR NHSTHTKNGS PQKGYEKFEF NIEDCRKF I DFYKQSISKH PEWKDFGFRF SDTQRYNSID EFYREVENQG YKLTFENISE SYIDSVVNQG KLYLFQIYNK DFSAYSKGR PNLHTLYWKA LFDERNLQDV VYKLNGEAEL FYRKQSIPKK ITHPAKEAIA NKNKDNPKKE SVFEYDLIKD KRFTEDKFFF HCPITINFK SSGANKFNDE INLLLKEKAN DVHILSIDRG ERHLAYYTLV DGKGNIIKQD TFNIIGNDRM KTNYHDKLAA I EKDRDSAR KDWKKINNIK EMKEGYLSQV VHEIAKLVIE YNAIVVFQDL NFGFKRGRFK VEKQVYQKLE KMLIEKLNYL VF KDNEFDK TGGVLRAYQL TAPFETFKKM GKQTGIIYYV PAGFTSKICP VTGFVNQLYP KYESVSKSQE FFSKFDKICY NLD KGYFEF SFDYKNFGDK AAKGKWTIAS FGSRLINFRN SDKNHNWDTR EVYPTKELEK LLKDYSIEYG HGECIKAAIC GESD KKFFA KLTSVLNTIL QMRNSKTGTE LDYLISPVAD VNGNFFDSRQ APKNMPQDAD ANGAYHIGLK GLMLLGRIKN NQEGK KLNL VIKNEEYFEF VQNRNNGSEF ELENLYFQGE LRRQASALEH HHHHH UniProtKB: CRISPR-associated endonuclease Cas12a |
-分子 #2: RNA (28-MER)
| 分子 | 名称: RNA (28-MER) / タイプ: rna / ID: 2 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種:   Francisella tularensis subsp. novicida U112 (バクテリア) Francisella tularensis subsp. novicida U112 (バクテリア) |
| 分子量 | 理論値: 13.749146 KDa |
| 配列 | 文字列: AAUUUCUACU GUUGUAGAUG AGAAGUCAUU UAAUAAGGCC ACU |
-分子 #3: DNA (5'-D(P*TP*GP*AP*CP*TP*TP*CP*TP*CP*TP*AP*AP*CP*AP*AP*GP*CP*TP...
| 分子 | 名称: DNA (5'-D(P*TP*GP*AP*CP*TP*TP*CP*TP*CP*TP*AP*AP*CP*AP*AP*GP*CP*TP*CP*G)-3') タイプ: dna / ID: 3 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:   Francisella tularensis subsp. novicida U112 (バクテリア) Francisella tularensis subsp. novicida U112 (バクテリア) |
| 分子量 | 理論値: 16.898826 KDa |
| 配列 | 文字列: (DA)(DT)(DT)(DG)(DC)(DT)(DT)(DG)(DC)(DT) (DC)(DG)(DA)(DT)(DG)(DC)(DA)(DT)(DG)(DC) (DA)(DG)(DT)(DG)(DG)(DC)(DC)(DT)(DT) (DA)(DT)(DT)(DA)(DA)(DA)(DT)(DG)(DA)(DC) (DT) (DT)(DC)(DT)(DC)(DT) ...文字列: (DA)(DT)(DT)(DG)(DC)(DT)(DT)(DG)(DC)(DT) (DC)(DG)(DA)(DT)(DG)(DC)(DA)(DT)(DG)(DC) (DA)(DG)(DT)(DG)(DG)(DC)(DC)(DT)(DT) (DA)(DT)(DT)(DA)(DA)(DA)(DT)(DG)(DA)(DC) (DT) (DT)(DC)(DT)(DC)(DT)(DA)(DA)(DC) (DG)(DA)(DG)(DC)(DT)(DC)(DG) |
-分子 #4: DNA (5'-D(P*CP*GP*AP*GP*CP*TP*CP*GP*TP*TP*AP*GP*AP*GP*AP*AP*GP*T)-3')
| 分子 | 名称: DNA (5'-D(P*CP*GP*AP*GP*CP*TP*CP*GP*TP*TP*AP*GP*AP*GP*AP*AP*GP*T)-3') タイプ: dna / ID: 4 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:   Francisella tularensis subsp. novicida U112 (バクテリア) Francisella tularensis subsp. novicida U112 (バクテリア) |
| 分子量 | 理論値: 16.992936 KDa |
| 配列 | 文字列: (DC)(DG)(DA)(DG)(DC)(DT)(DC)(DG)(DT)(DT) (DA)(DG)(DA)(DG)(DA)(DA)(DG)(DT)(DC)(DA) (DT)(DT)(DT)(DA)(DA)(DT)(DA)(DA)(DG) (DG)(DC)(DC)(DA)(DC)(DT)(DG)(DC)(DA)(DT) (DG) (DC)(DA)(DT)(DC)(DG) ...文字列: (DC)(DG)(DA)(DG)(DC)(DT)(DC)(DG)(DT)(DT) (DA)(DG)(DA)(DG)(DA)(DA)(DG)(DT)(DC)(DA) (DT)(DT)(DT)(DA)(DA)(DT)(DA)(DA)(DG) (DG)(DC)(DC)(DA)(DC)(DT)(DG)(DC)(DA)(DT) (DG) (DC)(DA)(DT)(DC)(DG)(DA)(DG)(DC) (DA)(DA)(DG)(DC)(DA)(DA)(DT) |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7 |
|---|---|
| グリッド | モデル: C-flat-1.2/1.3 4C / 材質: COPPER |
| 凍結 | 凍結剤: ETHANE / 装置: FEI VITROBOT MARK III |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy |
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: COUNTING / 平均電子線量: 40.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー