+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-8742 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Thermoplasma acidophilum 20S Proteasome using 200keV with image shift | |||||||||
 マップデータ マップデータ | Final sharpened map of T. acidophilum 20S proteasome collected using image shift navigation | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  Proteasome (プロテアソーム) / Proteasome (プロテアソーム) /  hydrolase (加水分解酵素) hydrolase (加水分解酵素) | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 proteasome endopeptidase complex / proteasome core complex, beta-subunit complex / proteasome core complex, alpha-subunit complex / threonine-type endopeptidase activity / proteasomal protein catabolic process / ubiquitin-dependent protein catabolic process / proteasome endopeptidase complex / proteasome core complex, beta-subunit complex / proteasome core complex, alpha-subunit complex / threonine-type endopeptidase activity / proteasomal protein catabolic process / ubiquitin-dependent protein catabolic process /  endopeptidase activity / endopeptidase activity /  細胞質基質 / 細胞質基質 /  細胞質 細胞質類似検索 - 分子機能 | |||||||||
| 生物種 |    Thermoplasma acidophilum (好酸性) Thermoplasma acidophilum (好酸性) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.3 Å クライオ電子顕微鏡法 / 解像度: 3.3 Å | |||||||||
 データ登録者 データ登録者 | Herzik Jr MA / Wu M / Lander GC | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Methods / 年: 2017 ジャーナル: Nat Methods / 年: 2017タイトル: Achieving better-than-3-Å resolution by single-particle cryo-EM at 200 keV. 著者: Mark A Herzik / Mengyu Wu / Gabriel C Lander /  要旨: Nearly all single-particle cryo-EM structures resolved to better than 4-Å resolution have been determined using 300-keV transmission electron microscopes (TEMs). We demonstrate that it is possible ...Nearly all single-particle cryo-EM structures resolved to better than 4-Å resolution have been determined using 300-keV transmission electron microscopes (TEMs). We demonstrate that it is possible to obtain reconstructions of macromolecular complexes of different sizes to better than 3-Å resolution using a 200-keV TEM. These structures are of sufficient quality to unambiguously assign amino acid rotameric conformations and identify ordered water molecules. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_8742.map.gz emd_8742.map.gz | 59.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-8742-v30.xml emd-8742-v30.xml emd-8742.xml emd-8742.xml | 22.5 KB 22.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_8742_fsc.xml emd_8742_fsc.xml | 18 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_8742.png emd_8742.png | 38.3 KB | ||
| Filedesc metadata |  emd-8742.cif.gz emd-8742.cif.gz | 6.6 KB | ||
| その他 |  emd_8742_additional.map.gz emd_8742_additional.map.gz emd_8742_half_map_1.map.gz emd_8742_half_map_1.map.gz emd_8742_half_map_2.map.gz emd_8742_half_map_2.map.gz | 59.1 MB 409.7 MB 409.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-8742 http://ftp.pdbj.org/pub/emdb/structures/EMD-8742 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8742 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8742 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  5vy4MC  8741C  8743C  5vy3C  5vy5C C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10186 (タイトル: T. acidophilum 20S proteasome core movies obtained using Talos Arctica operating at 200 kV equipped with a K2 – image shift used for exposure target navigation EMPIAR-10186 (タイトル: T. acidophilum 20S proteasome core movies obtained using Talos Arctica operating at 200 kV equipped with a K2 – image shift used for exposure target navigationData size: 945.5 Data #1: Raw, unaligned movie stacks of T. acidophilum 20S proteasome core acquired on a Talos Arctica using a K2 direct electron detector - image shift used for exposure navigation [micrographs - multiframe]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_8742.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_8742.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
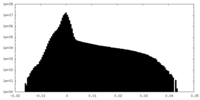
| 注釈 | Final sharpened map of T. acidophilum 20S proteasome collected using image shift navigation | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.91 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: Unsharpened map of T. acidophilum 20S proteasome collected...
| ファイル | emd_8742_additional.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
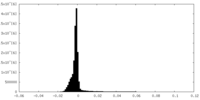
| 注釈 | Unsharpened map of T. acidophilum 20S proteasome collected using image shift navigation | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Thermoplasma acidophilum 20S Proteasome, even half map
| ファイル | emd_8742_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
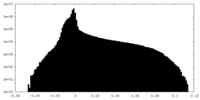
| 注釈 | Thermoplasma acidophilum 20S Proteasome, even half map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Thermoplasma acidophilum 20S Proteasome, odd half map
| ファイル | emd_8742_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
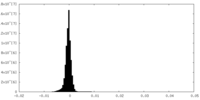
| 注釈 | Thermoplasma acidophilum 20S Proteasome, odd half map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Thermoplasma acidophilum 20S proteasome
| 全体 | 名称: Thermoplasma acidophilum 20S proteasome |
|---|---|
| 要素 |
|
-超分子 #1: Thermoplasma acidophilum 20S proteasome
| 超分子 | 名称: Thermoplasma acidophilum 20S proteasome / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: Thermoplasma acidophilum 20S proteasome purified from Escherichia coli |
|---|---|
| 由来(天然) | 生物種:    Thermoplasma acidophilum (好酸性) Thermoplasma acidophilum (好酸性) |
| 分子量 | 理論値: 700 KDa |
-分子 #1: Proteasome subunit alpha
| 分子 | 名称: Proteasome subunit alpha / タイプ: protein_or_peptide / ID: 1 / コピー数: 14 / 光学異性体: LEVO / EC番号:  proteasome endopeptidase complex proteasome endopeptidase complex |
|---|---|
| 由来(天然) | 生物種:    Thermoplasma acidophilum (好酸性) Thermoplasma acidophilum (好酸性) |
| 分子量 | 理論値: 24.776281 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: RAITVFSPDG RLFQVEYARE AVKKGSTALG MKFANGVLLI SDKKVRSRLI EQNSIEKIQL IDDYVAAVTS GLVADARVLV DFARISAQQ EKVTYGSLVN IENLVKRVAD QMQQYTQYGG VRPYGVSLIF AGIDQIGPRL FDCDPAGTIN EYKATAIGSG K DAVVSFLE ...文字列: RAITVFSPDG RLFQVEYARE AVKKGSTALG MKFANGVLLI SDKKVRSRLI EQNSIEKIQL IDDYVAAVTS GLVADARVLV DFARISAQQ EKVTYGSLVN IENLVKRVAD QMQQYTQYGG VRPYGVSLIF AGIDQIGPRL FDCDPAGTIN EYKATAIGSG K DAVVSFLE REYKENLPEK EAVTLGIKAL KSSLEEGEEL KAPEIASITV GNKYRIYDQE EVKKFL UniProtKB:  Proteasome subunit alpha Proteasome subunit alpha |
-分子 #2: Proteasome subunit beta
| 分子 | 名称: Proteasome subunit beta / タイプ: protein_or_peptide / ID: 2 / コピー数: 14 / 光学異性体: LEVO / EC番号:  proteasome endopeptidase complex proteasome endopeptidase complex |
|---|---|
| 由来(天然) | 生物種:    Thermoplasma acidophilum (好酸性) Thermoplasma acidophilum (好酸性) |
| 分子量 | 理論値: 22.294848 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: TTTVGITLKD AVIMATERRV TMENFIMHKN GKKLFQIDTY TGMTIAGLVG DAQVLVRYMK AELELYRLQR RVNMPIEAVA TLLSNMLNQ VKYMPYMVQL LVGGIDTAPH VFSIDAAGGS VEDIYASTGS GSPFVYGVLE SQYSEKMTVD EGVDLVIRAI S AAKQRDSA ...文字列: TTTVGITLKD AVIMATERRV TMENFIMHKN GKKLFQIDTY TGMTIAGLVG DAQVLVRYMK AELELYRLQR RVNMPIEAVA TLLSNMLNQ VKYMPYMVQL LVGGIDTAPH VFSIDAAGGS VEDIYASTGS GSPFVYGVLE SQYSEKMTVD EGVDLVIRAI S AAKQRDSA SGGMIDVAVI TRKDGYVQLP TDQIESRIRK LGLIL UniProtKB:  Proteasome subunit beta Proteasome subunit beta |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.5 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.4 構成要素:
| |||||||||
| グリッド | モデル: Quantifoil, UltrAuFoil, R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 前処理 - タイプ: PLASMA CLEANING / 前処理 - 時間: 7 sec. / 前処理 - 雰囲気: OTHER / 前処理 - 気圧: 0.009000000000000001 kPa / 詳細: 15 Watts | |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.15 K / 装置: HOMEMADE PLUNGER 詳細: 3 uL of sample/grid was manually blotted for 4 seconds prior to immediate plunge-freezing in liquid nitrogen-cooled ethane.. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm Bright-field microscopy / Cs: 2.7 mm最大 デフォーカス(公称値): 2.8000000000000003 µm 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 45000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / デジタル化 - サイズ - 横: 7420 pixel / デジタル化 - サイズ - 縦: 7676 pixel / デジタル化 - 画像ごとのフレーム数: 1-68 / 撮影したグリッド数: 1 / 実像数: 394 / 平均露光時間: 17.0 sec. / 平均電子線量: 65.0 e/Å2 詳細: Images were collected using image shift navigation to target exposure. |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
- 画像解析
画像解析
| 粒子像選択 | 選択した数: 111184 詳細: Template-based particle picking was performed using templates generated from reference-free 2D classification of an initial set of automated particle picks. |
|---|---|
| 初期モデル | モデルのタイプ: EMDB MAP EMDB ID:  6283 詳細: Initial model was low-passed filtered to 60 Angstrom resolution |
| 初期 角度割当 | タイプ: PROJECTION MATCHING / ソフトウェア - 名称: RELION (ver. 2.0) |
| 最終 角度割当 | タイプ: PROJECTION MATCHING / ソフトウェア - 名称: RELION (ver. 2.0) |
| 最終 再構成 | 想定した対称性 - 点群: D7 (2回x7回 2面回転対称 ) )アルゴリズム: BACK PROJECTION / 解像度のタイプ: BY AUTHOR / 解像度: 3.3 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: RELION (ver. 2.0) / 使用した粒子像数: 96254 |
| 詳細 | Movies were collected in super-resolution mode and Fourier-binned by two prior to motion correction and dose weighting using MotionCor2 program. |
FSC曲線 (解像度の算出) | 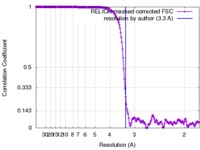 |
-原子モデル構築 1
| 詳細 | Starting model was generated by stripping PDB entry 1YAR of all ligands and alternate conformations, then refining into the EM density using imposed symmetry while adjusting weighting/scoring according to estimated map resolution. The top 10 generated models (ranked based on quality metrics) were real-space refined using Phenix software. |
|---|---|
| 精密化 | 空間: REAL / プロトコル: OTHER / 温度因子: 99 / 当てはまり具合の基準: Maximum Likelihood |
| 得られたモデル |  PDB-5vy4: |
 ムービー
ムービー コントローラー
コントローラー















 Z
Z Y
Y X
X