+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-8397 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | In situ structures of the genome and genome-delivery apparatus in ssRNA bacteriophage MS2 | |||||||||
 マップデータ マップデータ | Asymmetric cryoEM reconstruction of Enterobacteria Phage MS2 at 3.6 Angstroms resolution. | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報viral genome circularization / virion attachment to host cell pilus / negative regulation of viral translation / T=3 icosahedral viral capsid /  virion component / virion component /  regulation of translation / symbiont entry into host cell / structural molecule activity / regulation of translation / symbiont entry into host cell / structural molecule activity /  RNA binding / identical protein binding RNA binding / identical protein binding類似検索 - 分子機能 | |||||||||
| 生物種 |   Enterobacteria phage MS2 (ファージ) Enterobacteria phage MS2 (ファージ) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.6 Å クライオ電子顕微鏡法 / 解像度: 3.6 Å | |||||||||
 データ登録者 データ登録者 | Dai XH / Li ZH / Lai M / Shu S / Du YS / Zhou ZH / Sun R | |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2017 ジャーナル: Nature / 年: 2017タイトル: In situ structures of the genome and genome-delivery apparatus in a single-stranded RNA virus. 著者: Xinghong Dai / Zhihai Li / Mason Lai / Sara Shu / Yushen Du / Z Hong Zhou / Ren Sun /   要旨: Packaging of the genome into a protein capsid and its subsequent delivery into a host cell are two fundamental processes in the life cycle of a virus. Unlike double-stranded DNA viruses, which pump ...Packaging of the genome into a protein capsid and its subsequent delivery into a host cell are two fundamental processes in the life cycle of a virus. Unlike double-stranded DNA viruses, which pump their genome into a preformed capsid, single-stranded RNA (ssRNA) viruses, such as bacteriophage MS2, co-assemble their capsid with the genome; however, the structural basis of this co-assembly is poorly understood. MS2 infects Escherichia coli via the host 'sex pilus' (F-pilus); it was the first fully sequenced organism and is a model system for studies of translational gene regulation, RNA-protein interactions, and RNA virus assembly. Its positive-sense ssRNA genome of 3,569 bases is enclosed in a capsid with one maturation protein monomer and 89 coat protein dimers arranged in a T = 3 icosahedral lattice. The maturation protein is responsible for attaching the virus to an F-pilus and delivering the viral genome into the host during infection, but how the genome is organized and delivered is not known. Here we describe the MS2 structure at 3.6 Å resolution, determined by electron-counting cryo-electron microscopy (cryoEM) and asymmetric reconstruction. We traced approximately 80% of the backbone of the viral genome, built atomic models for 16 RNA stem-loops, and identified three conserved motifs of RNA-coat protein interactions among 15 of these stem-loops with diverse sequences. The stem-loop at the 3' end of the genome interacts extensively with the maturation protein, which, with just a six-helix bundle and a six-stranded β-sheet, forms a genome-delivery apparatus and joins 89 coat protein dimers to form a capsid. This atomic description of genome-capsid interactions in a spherical ssRNA virus provides insight into genome delivery via the host sex pilus and mechanisms underlying ssRNA-capsid co-assembly, and inspires speculation about the links between nucleoprotein complexes and the origins of viruses. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
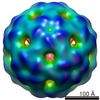
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_8397.map.gz emd_8397.map.gz | 109 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-8397-v30.xml emd-8397-v30.xml emd-8397.xml emd-8397.xml | 28.2 KB 28.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_8397.png emd_8397.png | 281.1 KB | ||
| その他 |  emd_8397_additional.map.gz emd_8397_additional.map.gz | 58 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-8397 http://ftp.pdbj.org/pub/emdb/structures/EMD-8397 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8397 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8397 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_8397.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_8397.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Asymmetric cryoEM reconstruction of Enterobacteria Phage MS2 at 3.6 Angstroms resolution. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.06 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: Asymmetric cryoEM reconstruction of Enterobacteria Phage MS2 low-pass...
| ファイル | emd_8397_additional.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Asymmetric cryoEM reconstruction of Enterobacteria Phage MS2 low-pass filtered to 6 Angstroms resolution to visualize the ssRNA genome. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
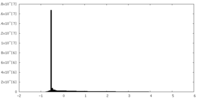
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Enterobacteria phage MS2
| 全体 | 名称:   Enterobacteria phage MS2 (ファージ) Enterobacteria phage MS2 (ファージ) |
|---|---|
| 要素 |
|
-超分子 #1: Enterobacteria phage MS2
| 超分子 | 名称: Enterobacteria phage MS2 / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: The viral stock was obtained from ATCC (ATCC number 15597-B1) and cultured in Escherichia coli strain C-3000 (ATCC 15597). NCBI-ID: 329852 / 生物種: Enterobacteria phage MS2 / ウイルスタイプ: VIRION / ウイルス・単離状態: SPECIES / ウイルス・エンベロープ: No / ウイルス・中空状態: No |
|---|---|
| 宿主 | 生物種:   Escherichia coli (大腸菌) / 株: C-3000 Escherichia coli (大腸菌) / 株: C-3000 |
| 分子量 | 理論値: 3.6 MDa |
| ウイルス殻 | Shell ID: 1 / 名称: capsid / 直径: 270.0 Å / T番号(三角分割数): 3 |
-超分子 #2: capsid shell of Enterobacteria Phage MS2
| 超分子 | 名称: capsid shell of Enterobacteria Phage MS2 / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1-#2 詳細: The capsid of MS2 is composed of 178 copies of the coat protein and one single copy of the maturation protein. |
|---|---|
| 由来(天然) | 生物種:   Enterobacteria phage MS2 (ファージ) Enterobacteria phage MS2 (ファージ) |
| 分子量 | 理論値: 2.5 MDa |
-超分子 #3: coat protein of Enterobacteria Phage MS2
| 超分子 | 名称: coat protein of Enterobacteria Phage MS2 / タイプ: complex / ID: 3 / 親要素: 2 / 含まれる分子: #1 詳細: The 178 copies of the coat protein are organized as 89 dimers in a T=3 icosahedral lattice. |
|---|---|
| 由来(天然) | 生物種:   Enterobacteria phage MS2 (ファージ) Enterobacteria phage MS2 (ファージ) |
| 分子量 | 理論値: 14 KDa |
-超分子 #4: maturation protein of Enterobacteria Phage MS2
| 超分子 | 名称: maturation protein of Enterobacteria Phage MS2 / タイプ: complex / ID: 4 / 親要素: 2 / 含まれる分子: #2 詳細: The single copy of maturation protein in the capsid shell of MS2 is located at one of the 2-fold symmetry axes and it replaces a coat protein dimer at this position. |
|---|---|
| 由来(天然) | 生物種:   Enterobacteria phage MS2 (ファージ) Enterobacteria phage MS2 (ファージ) |
| 分子量 | 理論値: 44 KDa |
-超分子 #5: the ssRNA genome of Enterobacteria Phage MS2
| 超分子 | 名称: the ssRNA genome of Enterobacteria Phage MS2 / タイプ: complex / ID: 5 / 親要素: 1 / 含まれる分子: #3 詳細: The genome of MS2 is a single-stranded RNA with 3569 bases. Our asymmetric cryoEM reconstruction of the MS2 virion shows that its ssRNA genome is well organized and has multiple contacts with ...詳細: The genome of MS2 is a single-stranded RNA with 3569 bases. Our asymmetric cryoEM reconstruction of the MS2 virion shows that its ssRNA genome is well organized and has multiple contacts with the capsid shell via tens of RNA stem-loop structures. |
|---|---|
| 由来(天然) | 生物種:   Enterobacteria phage MS2 (ファージ) Enterobacteria phage MS2 (ファージ) |
| 分子量 | 理論値: 1.1 MDa |
-分子 #1: Capsid protein
| 分子 | 名称: Capsid protein / タイプ: protein_or_peptide / ID: 1 詳細: The capsid of MS2 contains 178 copies of the coat protein arranged as 89 dimers in a T=3 icosahedral lattice. Structure of the capsid has been solved by crystallography with icosahedral ...詳細: The capsid of MS2 contains 178 copies of the coat protein arranged as 89 dimers in a T=3 icosahedral lattice. Structure of the capsid has been solved by crystallography with icosahedral symmetry applied (PDB ID 2MS2, also included as chains A, B, C in this model). Chains D, E, F, G, H are coat proteins that have slightly different structures in the asymmetric cryoEM reconstruction compared to the crystallographic structure. Structures of the other 170 copies of the coat protein are the same with 2MS2, and thus are not included due to the limited number of chain IDs. コピー数: 8 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Enterobacteria phage MS2 (ファージ) Enterobacteria phage MS2 (ファージ) |
| 分子量 | 理論値: 13.869659 KDa |
| 配列 | 文字列: MASNFTQFVL VDNGGTGDVT VAPSNFANGV AEWISSNSRS QAYKVTCSVR QSSAQNRKYT IKVEVPKVAT QTVGGVELPV AAWRSYLNM ELTIPIFATN SDCELIVKAM QGLLKDGNPI PSAIAANSGI Y |
-分子 #2: Maturation protein
| 分子 | 名称: Maturation protein / タイプ: protein_or_peptide / ID: 2 詳細: The capsid of MS2 contains a single copy of the maturation protein. Our structure shows that it replaces a coat protein dimer at one of the 2-fold icosahedral symmetry axes. Function of the ...詳細: The capsid of MS2 contains a single copy of the maturation protein. Our structure shows that it replaces a coat protein dimer at one of the 2-fold icosahedral symmetry axes. Function of the maturation protein is to attach the MS2 virion to the host (E. coli) F-pili and deliver the ssRNA viral genome into the host during infection. of the icosahedral capsid. 178 copies of the coat protein arranged as 89 dimers in a T=3 icosahedral lattice. Structure of the capsid has been solved by crystallography with icosahedral symmetry applied (PDB ID 2MS2, also included as chains A, B, C in this model). Chains D, E, F, G, H are coat proteins that have slightly different structures in the asymmetric cryoEM reconstruction compared to the crystallographic structure. Structures of the other 170 copies of the coat protein are the same with 2MS2, and thus are not included due to the limited number of chain IDs. コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Enterobacteria phage MS2 (ファージ) Enterobacteria phage MS2 (ファージ) |
| 分子量 | 理論値: 44.030934 KDa |
| 配列 | 文字列: MRAFSTLDRE NETFVPSVRV YADGETEDNS FSLKYRSNWT PGRFNSTGAK TKQWHYPSPY SRGALSVTSI DQGAYKRSGS SWGRPYEEK AGFGFSLDAR SCYSLFPVSQ NLTYIEVPQN VANRASTEVL QKVTQGNFNL GVALAEARST ASQLATQTIA L VKAYTAAR ...文字列: MRAFSTLDRE NETFVPSVRV YADGETEDNS FSLKYRSNWT PGRFNSTGAK TKQWHYPSPY SRGALSVTSI DQGAYKRSGS SWGRPYEEK AGFGFSLDAR SCYSLFPVSQ NLTYIEVPQN VANRASTEVL QKVTQGNFNL GVALAEARST ASQLATQTIA L VKAYTAAR RGNWRQALRY LALNEDRKFR SKHVAGRWLE LQFGWLPLMS DIQGAYEMLT KVHLQEFLPM RAVRQVGTNI KL DGRLSYP AANFQTTCNI SRRIVIWFYI NDARLAWLSS LGILNPLGIV WEKVPFSFVV DWLLPVGNML EGLTAPVGCS YMS GTVTDV ITGESIISVD APYGWTVERQ GTAKAQISAM HRGVQSVWPT TGAYVKSPFS MVHTLDALAL IRQRLSR |
-分子 #3: phage MS2 genome
| 分子 | 名称: phage MS2 genome / タイプ: rna / ID: 3 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種:   Enterobacteria phage MS2 (ファージ) Enterobacteria phage MS2 (ファージ) |
| 分子量 | 理論値: 1.14720075 MDa |
| 配列 | 文字列: GGGUGGGACC CCUUUCGGGG UCCUGCUCAA CUUCCUGUCG AGCUAAUGCC AUUUUUAAUG UCUUUAGCGA GACGCUACCA UGGCUAUCG CUGUAGGUAG CCGGAAUUCC AUUCCUAGGA GGUUUGACCU GUGCGAGCUU UUAGUACCCU UGAUAGGGAG A ACGAGACC ...文字列: GGGUGGGACC CCUUUCGGGG UCCUGCUCAA CUUCCUGUCG AGCUAAUGCC AUUUUUAAUG UCUUUAGCGA GACGCUACCA UGGCUAUCG CUGUAGGUAG CCGGAAUUCC AUUCCUAGGA GGUUUGACCU GUGCGAGCUU UUAGUACCCU UGAUAGGGAG A ACGAGACC UUCGUCCCCU CCGUUCGCGU UUACGCGGAC GGUGAGACUG AAGAUAACUC AUUCUCUUUA AAAUAUCGUU CG AACUGGA CUCCCGGUCG UUUUAACUCG ACUGGGGCCA AAACGAAACA GUGGCACUAC CCCUCUCCGU AUUCACGGGG GGC GUUAAG UGUCACAUCG AUAGAUCAAG GUGCCUACAA GCGAAGUGGG UCAUCGUGGG GUCGCCCGUA CGAGGAGAAA GCCG GUUUC GGCUUCUCCC UCGACGCACG CUCCUGCUAC AGCCUCUUCC CUGUAAGCCA AAACUUGACU UACAUCGAAG UGCCG CAGA ACGUUGCGAA CCGGGCGUCG ACCGAAGUCC UGCAAAAGGU CACCCAGGGU AAUUUUAACC UUGGUGUUGC UUUAGC AGA GGCCAGGUCG ACAGCCUCAC AACUCGCGAC GCAAACCAUU GCGCUCGUGA AGGCGUACAC UGCCGCUCGU CGCGGUA AU UGGCGCCAGG CGCUCCGCUA CCUUGCCCUA AACGAAGAUC GAAAGUUUCG AUCAAAACAC GUGGCCGGCA GGUGGUUG G AGUUGCAGUU CGGUUGGUUA CCACUAAUGA GUGAUAUCCA GGGUGCAUAU GAGAUGCUUA CGAAGGUUCA CCUUCAAGA GUUUCUUCCU AUGAGAGCCG UACGUCAGGU CGGUACUAAC AUCAAGUUAG AUGGCCGUCU GUCGUAUCCA GCUGCAAACU UCCAGACAA CGUGCAACAU AUCGCGACGU AUCGUGAUAU GGUUUUACAU AAACGAUGCA CGUUUGGCAU GGUUGUCGUC U CUAGGUAU CUUGAACCCA CUAGGUAUAG UGUGGGAAAA GGUGCCUUUC UCAUUCGUUG UCGACUGGCU CCUACCUGUA GG UAACAUG CUCGAGGGCC UUACGGCCCC CGUGGGAUGC UCCUACAUGU CAGGAACAGU UACUGACGUA AUAACGGGUG AGU CCAUCA UAAGCGUUGA CGCUCCCUAC GGGUGGACUG UGGAGAGACA GGGCACUGCU AAGGCCCAAA UCUCAGCCAU GCAU CGAGG GGUACAAUCC GUAUGGCCAA CAACUGGCGC GUACGUAAAG UCUCCUUUCU CGAUGGUCCA UACCUUAGAU GCGUU AGCA UUAAUCAGGC AACGGCUCUC UAGAUAGAGC CCUCAACCGG AGUUUGAAGC AUGGCUUCUA ACUUUACUCA GUUCGU UCU CGUCGACAAU GGCGGAACUG GCGACGUGAC UGUCGCCCCA AGCAACUUCG CUAACGGGGU CGCUGAAUGG AUCAGCU CU AACUCGCGUU CACAGGCUUA CAAAGUAACC UGUAGCGUUC GUCAGAGCUC UGCGCAGAAU CGCAAAUACA CCAUCAAA G UCGAGGUGCC UAAAGUGGCA ACCCAGACUG UUGGUGGUGU AGAGCUUCCU GUAGCCGCAU GGCGUUCGUA CUUAAAUAU GGAACUAACC AUUCCAAUUU UCGCUACGAA UUCCGACUGC GAGCUUAUUG UUAAGGCAAU GCAAGGUCUC CUAAAAGAUG GAAACCCGA UUCCCUCAGC AAUCGCAGCA AACUCCGGCA UCUACUAAUA GACGCCGGCC AUUCAAACAU GAGGAUUACC C AUGUCGAA GACAACAAAG AAGUUCAACU CUUUAUGUAU UGAUCUUCCU CGCGAUCUUU CUCUCGAAAU UUACCAAUCA AU UGCUUCU GUCGCUACUG GAAGCGGUGA UCCGCACAGU GACGACUUUA CAGCAAUUGC UUACUUAAGG GACGAAUUGC UCA CAAAGC AUCCGACCUU AGGUUCUGGU AAUGACGAGG CGACCCGUCG UACCUUAGCU AUCGCUAAGC UACGGGAGGC GAAU GGUGA UCGCGGUCAG AUAAAUAGAG AAGGUUUCUU ACAUGACAAA UCCUUGUCAU GGGAUCCGGA UGUUUUACAA ACCAG CAUC CGUAGCCUUA UUGGCAACCU CCUCUCUGGC UACCGAUCGU CGUUGUUUGG GCAAUGCACG UUCUCCAACG GUGCUC CUA UGGGGCACAA GUUGCAGGAU GCAGCGCCUU ACAAGAAGUU CGCUGAACAA GCAACCGUUA CCCCCCGCGC UCUGAGA GC GGCUCUAUUG GUCCGAGACC AAUGUGCGCC GUGGAUCAGA CACGCGGUCC GCUAUAACGA GUCAUAUGAA UUUAGGCU C GUUGUAGGGA ACGGAGUGUU UACAGUUCCG AAGAAUAAUA AAAUAGAUCG GGCUGCCUGU AAGGAGCCUG AUAUGAAUA UGUACCUCCA GAAAGGGGUC GGUGCUUUCA UCAGACGCCG GCUCAAAUCC GUUGGUAUAG ACCUGAAUGA UCAAUCGAUC AACCAGCGU CUGGCUCAGC AGGGCAGCGU AGAUGGUUCG CUUGCGACGA UAGACUUAUC GUCUGCAUCC GAUUCCAUCU C CGAUCGCC UGGUGUGGAG UUUUCUCCCA CCAGAGCUAU AUUCAUAUCU CGAUCGUAUC CGCUCACACU ACGGAAUCGU AG AUGGCGA GACGAUACGA UGGGAACUAU UUUCCACAAU GGGAAAUGGG UUCACAUUUG AGCUAGAGUC CAUGAUAUUC UGG GCAAUA GUCAAAGCGA CCCAAAUCCA UUUUGGUAAC GCCGGAACCA UAGGCAUCUA CGGGGACGAU AUUAUAUGUC CCAG UGAGA UUGCACCCCG UGUGCUAGAG GCACUUGCCU ACUACGGUUU UAAACCGAAU CUUCGUAAAA CGUUCGUGUC CGGGC UCUU UCGCGAGAGC UGCGGCGCGC ACUUUUACCG UGGUGUCGAU GUCAAACCGU UUUACAUCAA GAAACCUGUU GACAAU CUC UUCGCCCUGA UGCUGAUAUU AAAUCGGCUA CGGGGUUGGG GAGUUGUCGG AGGUAUGUCA GAUCCACGCC UCUAUAA GG UGUGGGUACG GCUCUCCUCC CAGGUGCCUU CGAUGUUCUU CGGUGGGACG GACCUCGCUG CCGACUACUA CGUAGUCA G CCCGCCUACG GCAGUCUCGG UAUACACCAA GACUCCGUAC GGGCGGCUGC UCGCGGAUAC CCGUACCUCG GGUUUCCGU CUUGCUCGUA UCGCUCGAGA ACGCAAGUUC UUCAGCGAAA AGCACGACAG UGGUCGCUAC AUAGCGUGGU UCCAUACUGG AGGUGAAAU CACCGACAGC AUGAAGUCCG CCGGCGUGCG CGUUAUACGC ACUUCGGAGU GGCUAACGCC GGUUCCCACA U UCCCUCAG GAGUGUGGGC CAGCGAGCUC UCCUCGGUAG CUGACCGAGG GACCCCCGUA AACGGGGUGG GUGUGCUCGA AA GAGCACG GGUGCGAAAG CGGUCCGGCU CCACCGAAAG GUGGGCGGGC UUCGGCCCAG GGACCUCCCC CUAAAGAGAG GAC CCGGGA UUCUCCCGAU UUGGUAACUA GCUGCUUGGC UAGUUACCAC CCA |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 / 詳細: pH7.4 PBS |
|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: OTHER |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 295 K / 装置: FEI VITROBOT MARK II |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 倍率(補正後): 47170 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 倍率(公称値): 130000 Bright-field microscopy / Cs: 2.7 mm / 倍率(公称値): 130000 |
| 特殊光学系 | エネルギーフィルター - 名称: GIF エネルギーフィルター - エネルギー下限: 20 eV エネルギーフィルター - エネルギー上限: 20 eV |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 温度 | 最低: 79.0 K |
| 詳細 | EFTEM mode with Gatan GIF energy filter. |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / デジタル化 - サイズ - 横: 7676 pixel / デジタル化 - サイズ - 縦: 7420 pixel / デジタル化 - サンプリング間隔: 5.0 µm / デジタル化 - 画像ごとのフレーム数: 1-14 / 撮影したグリッド数: 2 / 実像数: 6080 / 平均露光時間: 5.8 sec. / 平均電子線量: 50.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 粒子像選択 | 選択した数: 360000 |
|---|---|
| CTF補正 | ソフトウェア - 名称: CTFFIND3 |
| 初期モデル | モデルのタイプ: INSILICO MODEL In silico モデル: A subset of particle images were processed with refine2d.py of EMAN1.9. The resulted 2D averages were processed with starticos of EMAN1.9 to produce an initial model with ...In silico モデル: A subset of particle images were processed with refine2d.py of EMAN1.9. The resulted 2D averages were processed with starticos of EMAN1.9 to produce an initial model with icosahedral symmetry at roughly 50 Angstroms resolution. 詳細: This initial model was used to do 3D classification of the dataset with C1 symmetry using Relion. One of the resulted 3D classes showed prominent genome densities was used as initial model ...詳細: This initial model was used to do 3D classification of the dataset with C1 symmetry using Relion. One of the resulted 3D classes showed prominent genome densities was used as initial model for asymmetric structure refinement with Frealign. |
| 初期 角度割当 | タイプ: PROJECTION MATCHING Projection matching processing - Angular sampling: 5.0 degrees ソフトウェア - 名称: FREALIGN (ver. 9.11) / 詳細: Frealign grid search (mode 3) |
| 最終 3次元分類 | クラス数: 10 / 平均メンバー数/クラス: 30000 / ソフトウェア - 名称: RELION (ver. 1.4) |
| 最終 角度割当 | タイプ: PROJECTION MATCHING / ソフトウェア - 名称: FREALIGN (ver. 9.11) / 詳細: Frealign local refinement (mode 1) |
| 最終 再構成 | 使用したクラス数: 1 / 想定した対称性 - 点群: C1 (非対称) / アルゴリズム: FOURIER SPACE / 解像度のタイプ: BY AUTHOR / 解像度: 3.6 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: FREALIGN (ver. 9.11) / 使用した粒子像数: 339718 |
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL |
|---|---|
| 得られたモデル |  PDB-5tc1: |
 ムービー
ムービー コントローラー
コントローラー














 Z
Z Y
Y X
X