+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-7774 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | 3.9A Cryo-EM structure of murine antibody bound at a novel epitope of respiratory syncytial virus fusion protein | |||||||||
 マップデータ マップデータ | 3.9A density map of R4.C6 in complex with RSV F710 | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報positive regulation of syncytium formation by virus / Assembly and release of respiratory syncytial virus (RSV) virions / Translation of respiratory syncytial virus mRNAs / Respiratory syncytial virus (RSV) attachment and entry / RSV-host interactions / Maturation of hRSV A proteins / host cell Golgi membrane / entry receptor-mediated virion attachment to host cell / symbiont entry into host cell / fusion of virus membrane with host plasma membrane ...positive regulation of syncytium formation by virus / Assembly and release of respiratory syncytial virus (RSV) virions / Translation of respiratory syncytial virus mRNAs / Respiratory syncytial virus (RSV) attachment and entry / RSV-host interactions / Maturation of hRSV A proteins / host cell Golgi membrane / entry receptor-mediated virion attachment to host cell / symbiont entry into host cell / fusion of virus membrane with host plasma membrane /  エンベロープ (ウイルス) / host cell plasma membrane / virion membrane / identical protein binding / エンベロープ (ウイルス) / host cell plasma membrane / virion membrane / identical protein binding /  細胞膜 細胞膜類似検索 - 分子機能 | |||||||||
| 生物種 |   Mus musculus (ハツカネズミ) / Mus musculus (ハツカネズミ) /   Human respiratory syncytial virus A2 (RSウイルス) / Human respiratory syncytial virus A2 (RSウイルス) /    Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.9 Å クライオ電子顕微鏡法 / 解像度: 3.9 Å | |||||||||
 データ登録者 データ登録者 | Xie Q / Wang Z / Chen X / Ni F / Ma J / Wang Q | |||||||||
 引用 引用 |  ジャーナル: PLoS One / 年: 2019 ジャーナル: PLoS One / 年: 2019タイトル: Structure basis of neutralization by a novel site II/IV antibody against respiratory syncytial virus fusion protein. 著者: Qingqing Xie / Zhao Wang / Fengyun Ni / Xiaorui Chen / Jianpeng Ma / Nita Patel / Hanxin Lu / Ye Liu / Jing-Hui Tian / David Flyer / Michael J Massare / Larry Ellingsworth / Gregory Glenn / ...著者: Qingqing Xie / Zhao Wang / Fengyun Ni / Xiaorui Chen / Jianpeng Ma / Nita Patel / Hanxin Lu / Ye Liu / Jing-Hui Tian / David Flyer / Michael J Massare / Larry Ellingsworth / Gregory Glenn / Gale Smith / Qinghua Wang /  要旨: Globally, human respiratory syncytial virus (RSV) is a leading cause of lower respiratory tract infections in newborns, young children, and the elderly for which there is no vaccine. The RSV fusion ...Globally, human respiratory syncytial virus (RSV) is a leading cause of lower respiratory tract infections in newborns, young children, and the elderly for which there is no vaccine. The RSV fusion (F) glycoprotein is a major target for vaccine development. Here, we describe a novel monoclonal antibody (designated as R4.C6) that recognizes both pre-fusion and post-fusion RSV F, and binds with nanomole affinity to a unique neutralizing site comprised of antigenic sites II and IV on the globular head. A 3.9 Å-resolution structure of RSV F-R4.C6 Fab complex was obtained by single particle cryo-electron microscopy and 3D reconstruction. The structure unraveled detailed interactions of R4.C6 with antigenic site II on one protomer and site IV on a neighboring protomer of post-fusion RSV F protein. These findings significantly further our understanding of the antigenic complexity of the F protein and provide new insights into RSV vaccine design. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_7774.map.gz emd_7774.map.gz | 59.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-7774-v30.xml emd-7774-v30.xml emd-7774.xml emd-7774.xml | 16.1 KB 16.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
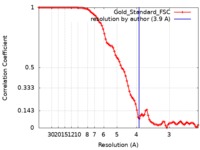
| FSC (解像度算出) |  emd_7774_fsc.xml emd_7774_fsc.xml | 9.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_7774.png emd_7774.png | 47.2 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-7774 http://ftp.pdbj.org/pub/emdb/structures/EMD-7774 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7774 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7774 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_7774.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_7774.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | 3.9A density map of R4.C6 in complex with RSV F710 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.2546 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Complex of R4.C6 Fab with RSV F protein
| 全体 | 名称: Complex of R4.C6 Fab with RSV F protein |
|---|---|
| 要素 |
|
-超分子 #1: Complex of R4.C6 Fab with RSV F protein
| 超分子 | 名称: Complex of R4.C6 Fab with RSV F protein / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 |
|---|
-超分子 #2: R4.C6 Fab
| 超分子 | 名称: R4.C6 Fab / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:   Mus musculus (ハツカネズミ) Mus musculus (ハツカネズミ) |
| 組換発現 | 生物種:   Mus musculus (ハツカネズミ) Mus musculus (ハツカネズミ) |
-超分子 #3: RSV F protein
| 超分子 | 名称: RSV F protein / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #3 |
|---|---|
| 由来(天然) | 生物種:   Human respiratory syncytial virus A2 (RSウイルス) Human respiratory syncytial virus A2 (RSウイルス) |
| 組換発現 | 生物種:   Spodoptera frugiperda (ツマジロクサヨトウ) Spodoptera frugiperda (ツマジロクサヨトウ) |
-分子 #1: R4.C6 Fab Heavy Chain
| 分子 | 名称: R4.C6 Fab Heavy Chain / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Mus musculus (ハツカネズミ) Mus musculus (ハツカネズミ) |
| 分子量 | 理論値: 12.831376 KDa |
| 組換発現 | 生物種:   Mus musculus (ハツカネズミ) Mus musculus (ハツカネズミ) |
| 配列 | 文字列: MAEVQLQQSG PELVKPGASV KISCKASGYA FSNSWMSWVK QRPGKGLEWI GRLFPADGDI TYNGHFKDKA ALTADKSSNT AYIQLSSLT SEDSAVYFCA RMDNSEVFWG QGTLVTVSA |
-分子 #2: R4.C6 Fab Light Chain
| 分子 | 名称: R4.C6 Fab Light Chain / タイプ: protein_or_peptide / ID: 2 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Mus musculus (ハツカネズミ) Mus musculus (ハツカネズミ) |
| 分子量 | 理論値: 13.457151 KDa |
| 組換発現 | 生物種:   Mus musculus (ハツカネズミ) Mus musculus (ハツカネズミ) |
| 配列 | 文字列: MADILLTQSQ KFMSTSVGDR VSITCKASQN VRTGVSWYQR KPGQSPKALI YLASNRHTGV PDRFTGRGSG TDFTLTISEV QSEDLADYF CLQHWTVPYT FGGGTKLEIK RTAAAPSRAN SFK |
-分子 #3: Fusion glycoprotein F0, Envelope glycoprotein chimera
| 分子 | 名称: Fusion glycoprotein F0, Envelope glycoprotein chimera タイプ: protein_or_peptide / ID: 3 / コピー数: 6 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:    Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
| 分子量 | 理論値: 60.728047 KDa |
| 組換発現 | 生物種:   Spodoptera frugiperda (ツマジロクサヨトウ) Spodoptera frugiperda (ツマジロクサヨトウ) |
| 配列 | 文字列: QNITEEFYQS TCSAVSKGYL SALRTGWYTS VITIELSNIK ENKCNGTDAK VKLIKQELDK YKNAVTELQL LMQSTPATNN RARRELPRF MNYTLNNAKK TNVTLSKKQK QQFLGFLLGV GSAIASGVAV SKVLHLEGEV NKIKSALLST NKAVVSLSNG V SVLTSKVL ...文字列: QNITEEFYQS TCSAVSKGYL SALRTGWYTS VITIELSNIK ENKCNGTDAK VKLIKQELDK YKNAVTELQL LMQSTPATNN RARRELPRF MNYTLNNAKK TNVTLSKKQK QQFLGFLLGV GSAIASGVAV SKVLHLEGEV NKIKSALLST NKAVVSLSNG V SVLTSKVL DLKNYIDKQL LPIVNKQSCS ISNIETVIEF QQKNNRLLEI TREFSVNAGV TTPVSTYMLT NSELLSLIND MP ITNDQKK LMSNNVQIVR QQSYSIMSII KEEVLAYVVQ LPLYGVIDTP CWKLHTSPLC TTNTKEGSNI CLTRTDRGWY CDN AGSVSF FPQAETCKVQ SNRVFCDTMN SLTLPSEVNL CNVDIFNPKY DCKIMTSKTD VSSSVITSLG AIVSCYGKTK CTAS NKNRG IIKTFSNGCD YVSNKGVDTV SVGNTLYYVN KQEGKSLYVK GEPIINFYDP LVFPSDEFDA SISQVNEKIN QSLAF IRKS DELLHNVNAG KSTTNIMGAL VPRGSPGSGY IPEAPRDGQA YVRKDGEWVL LSTFLGSSHH HHHH |
-分子 #4: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 4 / コピー数: 6 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7 |
|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 200 / 支持フィルム - 材質: GRAPHENE OXIDE |
| 凍結 | 凍結剤: NITROGEN / チャンバー内湿度: 98 % / チャンバー内温度: 295 K / 装置: LEICA EM GP |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | JEOL 3200FSC |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 4.7 mm Bright-field microscopy / Cs: 4.7 mm |
| 試料ステージ | 試料ホルダーモデル: JEOL 3200FSC CRYOHOLDER / ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / 平均露光時間: 10.0 sec. / 平均電子線量: 50.0 e/Å2 |
- 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
|---|---|
| 得られたモデル |  PDB-6cxc: |
 ムービー
ムービー コントローラー
コントローラー