+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CryoEM structure of an inward-facing MelBSt at a Na(+)-bound and sugar low-affinity conformation | |||||||||
 マップデータ マップデータ | The model was refined against this map. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  Sugar transporter / Cation-coupled symporter / Sugar transporter / Cation-coupled symporter /  Na(+) binding (ナトリウム) / Protein conformation / Na(+) binding (ナトリウム) / Protein conformation /  Nanobodies (ナノボディ) / NabFab / Nanobodies (ナノボディ) / NabFab /  CryoEM (低温電子顕微鏡法) / CryoEM (低温電子顕微鏡法) /  Membrane proteins (膜タンパク質) / Membrane proteins (膜タンパク質) /  protein-protein interaction (タンパク質間相互作用) / protein-protein interaction (タンパク質間相互作用) /  TRANSPORT PROTEIN (運搬体タンパク質) TRANSPORT PROTEIN (運搬体タンパク質) | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |   Escherichia coli (大腸菌) / Escherichia coli (大腸菌) /   Salmonella enterica subsp. enterica serovar Typhimurium (サルモネラ菌) / synthetic construct (人工物) Salmonella enterica subsp. enterica serovar Typhimurium (サルモネラ菌) / synthetic construct (人工物) | |||||||||
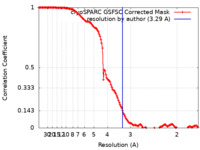
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.29 Å クライオ電子顕微鏡法 / 解像度: 3.29 Å | |||||||||
 データ登録者 データ登録者 | Guan L | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2024 ジャーナル: Elife / 年: 2024タイトル: Mobile barrier mechanisms for Na-coupled symport in an MFS sugar transporter. 著者: Parameswaran Hariharan / Yuqi Shi / Satoshi Katsube / Katleen Willibal / Nathan D Burrows / Patrick Mitchell / Amirhossein Bakhtiiari / Samantha Stanfield / Els Pardon / H Ronald Kaback / ...著者: Parameswaran Hariharan / Yuqi Shi / Satoshi Katsube / Katleen Willibal / Nathan D Burrows / Patrick Mitchell / Amirhossein Bakhtiiari / Samantha Stanfield / Els Pardon / H Ronald Kaback / Ruibin Liang / Jan Steyaert / Rosa Viner / Lan Guan /   要旨: While many 3D structures of cation-coupled transporters have been determined, the mechanistic details governing the obligatory coupling and functional regulations still remain elusive. The bacterial ...While many 3D structures of cation-coupled transporters have been determined, the mechanistic details governing the obligatory coupling and functional regulations still remain elusive. The bacterial melibiose transporter (MelB) is a prototype of major facilitator superfamily transporters. With a conformation-selective nanobody, we determined a low-sugar affinity inward-facing Na-bound cryoEM structure. The available outward-facing sugar-bound structures showed that the N- and C-terminal residues of the inner barrier contribute to the sugar selectivity. The inward-open conformation shows that the sugar selectivity pocket is also broken when the inner barrier is broken. Isothermal titration calorimetry measurements revealed that this inward-facing conformation trapped by this nanobody exhibited a greatly decreased sugar-binding affinity, suggesting the mechanisms for substrate intracellular release and accumulation. While the inner/outer barrier shift directly regulates the sugar-binding affinity, it has little or no effect on the cation binding, which is supported by molecular dynamics simulations. Furthermore, the hydron/deuterium exchange mass spectrometry analyses allowed us to identify dynamic regions; some regions are involved in the functionally important inner barrier-specific salt-bridge network, which indicates their critical roles in the barrier switching mechanisms for transport. These complementary results provided structural and dynamic insights into the mobile barrier mechanism for cation-coupled symport. #1:  ジャーナル: Elife / 年: 2024 ジャーナル: Elife / 年: 2024タイトル: Mobile barrier mechanisms for Na+-coupled symport in an MFS sugar transporter 著者: Hariharan P / Shi Y / Katsube S / Willibal K / Burrows ND / Mitchell P / Bakhtiiari A / Stanfield S / Pardon E / Kaback HR / Liang R / Steyaert J / Viner R / Gran L | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_41062.map.gz emd_41062.map.gz | 198.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-41062-v30.xml emd-41062-v30.xml emd-41062.xml emd-41062.xml | 24.4 KB 24.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_41062_fsc.xml emd_41062_fsc.xml | 14.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_41062.png emd_41062.png | 92.3 KB | ||
| マスクデータ |  emd_41062_msk_1.map emd_41062_msk_1.map | 216 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-41062.cif.gz emd-41062.cif.gz | 7.4 KB | ||
| その他 |  emd_41062_additional_1.map.gz emd_41062_additional_1.map.gz emd_41062_half_map_1.map.gz emd_41062_half_map_1.map.gz emd_41062_half_map_2.map.gz emd_41062_half_map_2.map.gz | 107.5 MB 200.5 MB 200.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-41062 http://ftp.pdbj.org/pub/emdb/structures/EMD-41062 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41062 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41062 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8t60MC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_41062.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_41062.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | The model was refined against this map. | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.86 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_41062_msk_1.map emd_41062_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: #1
| ファイル | emd_41062_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_41062_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_41062_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : MelBSt bound with Nb725_4-NabFab complex
| 全体 | 名称: MelBSt bound with Nb725_4-NabFab complex |
|---|---|
| 要素 |
|
-超分子 #1: MelBSt bound with Nb725_4-NabFab complex
| 超分子 | 名称: MelBSt bound with Nb725_4-NabFab complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 分子量 | 理論値: 120 KDa |
-分子 #1: Melibiose permease
| 分子 | 名称: Melibiose permease / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Salmonella enterica subsp. enterica serovar Typhimurium (サルモネラ菌) Salmonella enterica subsp. enterica serovar Typhimurium (サルモネラ菌)株: LT2 |
| 分子量 | 理論値: 54.104438 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: SISMTTKLSY GFGAFGKDFA IGIVYMYLMY YYTDVVGLSV GLVGTLFLVA RIWDAINDPI MGWIVNATRS RWGKFKPWIL IGTLTNSLV LFLLFSAHLF EGTAQVVFVC VTYILWGMTY TIMDIPFWSL VPTITLDKRE REQLVPFPRF FASLAGFVTA G ITLPFVSY ...文字列: SISMTTKLSY GFGAFGKDFA IGIVYMYLMY YYTDVVGLSV GLVGTLFLVA RIWDAINDPI MGWIVNATRS RWGKFKPWIL IGTLTNSLV LFLLFSAHLF EGTAQVVFVC VTYILWGMTY TIMDIPFWSL VPTITLDKRE REQLVPFPRF FASLAGFVTA G ITLPFVSY VGGADRGFGF QMFTLVLIAF FIASTIVTLR NVHEVYSSDN GVTAGRPHLT LKTIVGLIYK NDQLSCLLGM AL AYNIASN IINGFAIYYF TYVIGDADLF PYYLSYAGAA NLLTLIVFPR LVKMLSRRIL WAGASVMPVL SCAGLFAMAL ADI HNAALI VAAGIFLNIG TALFWVLQVI MVADTVDYGE FKLNIRCESI AYSVQTMVVK GGSAFAAFFI ALVLGLIGYT PNVA QSAQT LQGMQFIMIV LPVLFFMMTL VLYFRYYRLN GDMLRKIQIH LLDKYRKTPP FVEQPDSPAI SVVATSDVKA HHHHH HHHH H UniProtKB: Melibiose permease |
-分子 #2: Nb725_4
| 分子 | 名称: Nb725_4 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 14.817506 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: MSQRQLVESG GGLVQPGGSL RLSCAVSGII FRDNAMGWYR QAPGKEREWV ATITDLGYTA YADSVKGRFT ISRDNAKDTV YLQMNSLEP EDTAVYYCHL PGTAAGDYWG KGTPVTVSSL EVLFQGPHHH HHHHH |
-分子 #3: NabFab_H Chain
| 分子 | 名称: NabFab_H Chain / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 25.684463 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: EISEVQLVES GGGLVQPGGS LRLSCAASGF NFSYYSIHWV RQAPGKGLEW VAYISSSSSY TSYADSVKGR FTISADTSKN TAYLQMNSL RAEDTAVYYC ARGYQYWQYH ASWYWNGGLD YWGQGTLVTV SSASTKGPSV FPLAPSSKST SGGTAALGCL V KDYFPEPV ...文字列: EISEVQLVES GGGLVQPGGS LRLSCAASGF NFSYYSIHWV RQAPGKGLEW VAYISSSSSY TSYADSVKGR FTISADTSKN TAYLQMNSL RAEDTAVYYC ARGYQYWQYH ASWYWNGGLD YWGQGTLVTV SSASTKGPSV FPLAPSSKST SGGTAALGCL V KDYFPEPV TVSWNSGALT SGVHTFPAVL QSSGLYSLSS VVTVPSSSLG TQTYICNVNH KPSNTKVDKK VEPKSCDKTH T |
-分子 #4: NabFab_L Chain
| 分子 | 名称: NabFab_L Chain / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 23.258783 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: SDIQMTQSPS SLSASVGDRV TITCRASQSV SSAVAWYQQK PGKAPKLLIY SASSLYSGVP SRFSGSRSGT DFTLTISSLQ PEDFATYYC QQSSSSLITF GQGTKVEIKR TVAAPSVFIF PPSDSQLKSG TASVVCLLNN FYPREAKVQW KVDNALQSGN S QESVTEQD ...文字列: SDIQMTQSPS SLSASVGDRV TITCRASQSV SSAVAWYQQK PGKAPKLLIY SASSLYSGVP SRFSGSRSGT DFTLTISSLQ PEDFATYYC QQSSSSLITF GQGTKVEIKR TVAAPSVFIF PPSDSQLKSG TASVVCLLNN FYPREAKVQW KVDNALQSGN S QESVTEQD SKDSTYSLSS TLTLSKADYE KHKVYACEVT HQGLSSPVTK SFNRGEC |
-分子 #5: SODIUM ION
| 分子 | 名称: SODIUM ION / タイプ: ligand / ID: 5 / コピー数: 1 |
|---|---|
| 分子量 | 理論値: 22.99 Da |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 |
| 凍結 | 凍結剤: ETHANE / 装置: FEI VITROBOT MARK II |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 1.8 µm / 最小 デフォーカス(公称値): 0.8 µm Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 1.8 µm / 最小 デフォーカス(公称値): 0.8 µm |
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: FEI CETA (4k x 4k) / 実像数: 20778 / 平均露光時間: 0.0535 sec. / 平均電子線量: 1.28 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 初期モデル |
| ||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | プロトコル: AB INITIO MODEL | ||||||||||
| 得られたモデル |  PDB-8t60: |
 ムービー
ムービー コントローラー
コントローラー







 Z
Z Y
Y X
X