+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CryoEM structure of yeast recombination mediator Rad52 | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Recombination mediator protein /  DNA repair (DNA修復) / Apo structure / DNA repair (DNA修復) / Apo structure /  Decamer (オリゴマー) / Decamer (オリゴマー) /  RECOMBINATION (遺伝的組換え) RECOMBINATION (遺伝的組換え) | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報HDR through Single Strand Annealing (SSA) / meiotic joint molecule formation / double-strand break repair via single-strand annealing /  DNA amplification / DNA/DNA annealing activity / SUMOylation of DNA damage response and repair proteins / telomere maintenance via recombination / DNA recombinase assembly / DNA amplification / DNA/DNA annealing activity / SUMOylation of DNA damage response and repair proteins / telomere maintenance via recombination / DNA recombinase assembly /  mitotic recombination / double-strand break repair via break-induced replication ...HDR through Single Strand Annealing (SSA) / meiotic joint molecule formation / double-strand break repair via single-strand annealing / mitotic recombination / double-strand break repair via break-induced replication ...HDR through Single Strand Annealing (SSA) / meiotic joint molecule formation / double-strand break repair via single-strand annealing /  DNA amplification / DNA/DNA annealing activity / SUMOylation of DNA damage response and repair proteins / telomere maintenance via recombination / DNA recombinase assembly / DNA amplification / DNA/DNA annealing activity / SUMOylation of DNA damage response and repair proteins / telomere maintenance via recombination / DNA recombinase assembly /  mitotic recombination / double-strand break repair via break-induced replication / DNA strand exchange activity / mitotic recombination / double-strand break repair via break-induced replication / DNA strand exchange activity /  postreplication repair / mitochondrial DNA repair / nuclear chromosome / double-strand break repair via homologous recombination / postreplication repair / mitochondrial DNA repair / nuclear chromosome / double-strand break repair via homologous recombination /  ミトコンドリア / ミトコンドリア /  細胞核 細胞核類似検索 - 分子機能 | |||||||||
| 生物種 |   Saccharomyces cerevisiae S288C (パン酵母) Saccharomyces cerevisiae S288C (パン酵母) | |||||||||
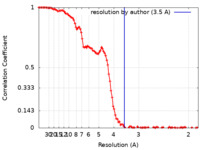
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.5 Å クライオ電子顕微鏡法 / 解像度: 3.5 Å | |||||||||
 データ登録者 データ登録者 | Deveryshetty J / Basore K / Rau M / Fitzpatrick JAJ / Antony E | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2023 ジャーナル: Nat Commun / 年: 2023タイトル: Yeast Rad52 is a homodecamer and possesses BRCA2-like bipartite Rad51 binding modes. 著者: Jaigeeth Deveryshetty / Rahul Chadda / Jenna R Mattice / Simrithaa Karunakaran / Michael J Rau / Katherine Basore / Nilisha Pokhrel / Noah Englander / James A J Fitzpatrick / Brian Bothner / Edwin Antony /  要旨: Homologous recombination (HR) is an essential double-stranded DNA break repair pathway. In HR, Rad52 facilitates the formation of Rad51 nucleoprotein filaments on RPA-coated ssDNA. Here, we decipher ...Homologous recombination (HR) is an essential double-stranded DNA break repair pathway. In HR, Rad52 facilitates the formation of Rad51 nucleoprotein filaments on RPA-coated ssDNA. Here, we decipher how Rad52 functions using single-particle cryo-electron microscopy and biophysical approaches. We report that Rad52 is a homodecameric ring and each subunit possesses an ordered N-terminal and disordered C-terminal half. An intrinsic structural asymmetry is observed where a few of the C-terminal halves interact with the ordered ring. We describe two conserved charged patches in the C-terminal half that harbor Rad51 and RPA interacting motifs. Interactions between these patches regulate ssDNA binding. Surprisingly, Rad51 interacts with Rad52 at two different bindings sites: one within the positive patch in the disordered C-terminus and the other in the ordered ring. We propose that these features drive Rad51 nucleation onto a single position on the DNA to promote formation of uniform pre-synaptic Rad51 filaments in HR. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_29695.map.gz emd_29695.map.gz | 96.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-29695-v30.xml emd-29695-v30.xml emd-29695.xml emd-29695.xml | 17.4 KB 17.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_29695_fsc.xml emd_29695_fsc.xml | 13.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_29695.png emd_29695.png | 65.3 KB | ||
| Filedesc metadata |  emd-29695.cif.gz emd-29695.cif.gz | 6.4 KB | ||
| その他 |  emd_29695_half_map_1.map.gz emd_29695_half_map_1.map.gz emd_29695_half_map_2.map.gz emd_29695_half_map_2.map.gz | 95.7 MB 95.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-29695 http://ftp.pdbj.org/pub/emdb/structures/EMD-29695 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-29695 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-29695 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8g3gMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_29695.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_29695.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 0.94 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_29695_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
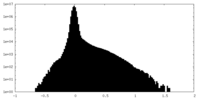
| 投影像・断面図 |
| ||||||||||||
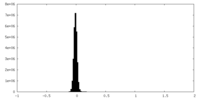
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_29695_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
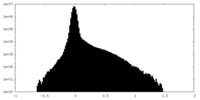
| 投影像・断面図 |
| ||||||||||||
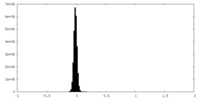
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Rad52 decamer
| 全体 | 名称: Rad52 decamer |
|---|---|
| 要素 |
|
-超分子 #1: Rad52 decamer
| 超分子 | 名称: Rad52 decamer / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: Full length yeast Rad52 expressed with chitin binding domain (CBD) tag. CBD was removed by thiol induced cleavage by intein. |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae S288C (パン酵母) / 細胞中の位置: Nucleus Saccharomyces cerevisiae S288C (パン酵母) / 細胞中の位置: Nucleus |
| 分子量 | 理論値: 520 kDa/nm |
-分子 #1: DNA repair and recombination protein RAD52
| 分子 | 名称: DNA repair and recombination protein RAD52 / タイプ: protein_or_peptide / ID: 1 / コピー数: 10 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae S288C (パン酵母) Saccharomyces cerevisiae S288C (パン酵母) |
| 分子量 | 理論値: 52.476496 KDa |
| 組換発現 | 生物種:   Escherichia coli BL21(DE3) (大腸菌) Escherichia coli BL21(DE3) (大腸菌) |
| 配列 | 文字列: MNEIMDMDEK KPVFGNHSED IQTKLDKKLG PEYISKRVGF GTSRIAYIEG WRVINLANQI FGYNGWSTEV KSVVIDFLDE RQGKFSIGC TAIVRVTLTS GTYREDIGYG TVENERRKPA AFERAKKSAV TDALKRSLRG FGNALGNCLY DKDFLAKIDK V KFDPPDFD ...文字列: MNEIMDMDEK KPVFGNHSED IQTKLDKKLG PEYISKRVGF GTSRIAYIEG WRVINLANQI FGYNGWSTEV KSVVIDFLDE RQGKFSIGC TAIVRVTLTS GTYREDIGYG TVENERRKPA AFERAKKSAV TDALKRSLRG FGNALGNCLY DKDFLAKIDK V KFDPPDFD ENNLFRPTDE ISESSRTNTL HENQEQQQYP NKRRQLTKVT NTNPDSTKNL VKIENTVSRG TPMMAAPAEA NS KNSSNKD TDLKSLDASK QDQDDLLDDS LMFSDDFQDD DLINMGNTNS NVLTTEKDPV VAKQSPTASS NPEAEQITFV TAK AATSVQ NERYIGEESI FDPKYQAQSI RHTVDQTTSK HIPASVLKDK TMTTARDSVY EKFAPKGKQL SMKNNDKELG PHML EGAGN QVPRETTPIK TNATAFPPAA APRFAPPSKV VHPNGNGAVP AVPQQRSTRR EVGRPKINPL HARKPT UniProtKB: DNA repair and recombination protein RAD52 |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.3 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.8 構成要素:
| ||||||||||||
| グリッド | モデル: Quantifoil R2/2 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: PLASMA CLEANING / 前処理 - 時間: 60 sec. | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 97 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS GLACIOS |
|---|---|
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 150000 Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 150000 |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) 平均電子線量: 50.7 e/Å2 |
- 画像解析
画像解析
-原子モデル構築 1
| 詳細 | Initial model was built de novo in ModelAngelo tool. the best-looking subunit was real space refined in Phenix, followed by manual building in Coot. Later C10 symmetry was applied, and real space refined in Phenix. |
|---|---|
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL |
| 得られたモデル |  PDB-8g3g: |
 ムービー
ムービー コントローラー
コントローラー




 Z
Z Y
Y X
X