+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CryoEM structure of cytoplasmic GAPDH under 24h Oxidative Stress | |||||||||
 マップデータ マップデータ | GAPDH, cyto, oxidative stress 24h, overall, d2 Glyceraldehyde 3-phosphate dehydrogenase Glyceraldehyde 3-phosphate dehydrogenase | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  GAPDH / GAPDH /  energy (エネルギー) / energy (エネルギー) /  cytosolic protein (細胞質基質) / cytosolic protein (細胞質基質) /  TRANSFERASE (転移酵素) TRANSFERASE (転移酵素) | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 転移酵素; 含窒素の基を移すもの; その他の含窒素の基を移すもの / peptidyl-cysteine S-nitrosylase activity / peptidyl-cysteine S-trans-nitrosylation / 転移酵素; 含窒素の基を移すもの; その他の含窒素の基を移すもの / peptidyl-cysteine S-nitrosylase activity / peptidyl-cysteine S-trans-nitrosylation /  グリセルアルデヒド-3-リン酸デヒドロゲナーゼ (リン酸化) / killing by host of symbiont cells / glyceraldehyde-3-phosphate dehydrogenase (NAD+) (phosphorylating) activity / negative regulation of endopeptidase activity / aspartic-type endopeptidase inhibitor activity / グリセルアルデヒド-3-リン酸デヒドロゲナーゼ (リン酸化) / killing by host of symbiont cells / glyceraldehyde-3-phosphate dehydrogenase (NAD+) (phosphorylating) activity / negative regulation of endopeptidase activity / aspartic-type endopeptidase inhibitor activity /  糖新生 / 糖新生 /  GAIT complex ... GAIT complex ... 転移酵素; 含窒素の基を移すもの; その他の含窒素の基を移すもの / peptidyl-cysteine S-nitrosylase activity / peptidyl-cysteine S-trans-nitrosylation / 転移酵素; 含窒素の基を移すもの; その他の含窒素の基を移すもの / peptidyl-cysteine S-nitrosylase activity / peptidyl-cysteine S-trans-nitrosylation /  グリセルアルデヒド-3-リン酸デヒドロゲナーゼ (リン酸化) / killing by host of symbiont cells / glyceraldehyde-3-phosphate dehydrogenase (NAD+) (phosphorylating) activity / negative regulation of endopeptidase activity / aspartic-type endopeptidase inhibitor activity / グリセルアルデヒド-3-リン酸デヒドロゲナーゼ (リン酸化) / killing by host of symbiont cells / glyceraldehyde-3-phosphate dehydrogenase (NAD+) (phosphorylating) activity / negative regulation of endopeptidase activity / aspartic-type endopeptidase inhibitor activity /  糖新生 / 糖新生 /  GAIT complex / GAIT complex /  解糖系 / positive regulation of type I interferon production / 解糖系 / positive regulation of type I interferon production /  regulation of macroautophagy / defense response to fungus / regulation of macroautophagy / defense response to fungus /  lipid droplet / positive regulation of cytokine production / glycolytic process / microtubule cytoskeleton organization / cellular response to type II interferon / glucose metabolic process / NAD binding / microtubule cytoskeleton / disordered domain specific binding / antimicrobial humoral immune response mediated by antimicrobial peptide / lipid droplet / positive regulation of cytokine production / glycolytic process / microtubule cytoskeleton organization / cellular response to type II interferon / glucose metabolic process / NAD binding / microtubule cytoskeleton / disordered domain specific binding / antimicrobial humoral immune response mediated by antimicrobial peptide /  NADP binding / NADP binding /  microtubule binding / neuron apoptotic process / positive regulation of canonical NF-kappaB signal transduction / microtubule binding / neuron apoptotic process / positive regulation of canonical NF-kappaB signal transduction /  核膜 / killing of cells of another organism / vesicle / negative regulation of translation / protein stabilization / 核膜 / killing of cells of another organism / vesicle / negative regulation of translation / protein stabilization /  ribonucleoprotein complex / intracellular membrane-bounded organelle / perinuclear region of cytoplasm / extracellular exosome / ribonucleoprotein complex / intracellular membrane-bounded organelle / perinuclear region of cytoplasm / extracellular exosome /  生体膜 / identical protein binding / 生体膜 / identical protein binding /  細胞核 / 細胞核 /  細胞膜 / 細胞膜 /  細胞質基質 / 細胞質基質 /  細胞質 細胞質類似検索 - 分子機能 | |||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 2.07 Å クライオ電子顕微鏡法 / 解像度: 2.07 Å | |||||||||
 データ登録者 データ登録者 | Choi WY / Wu H / Cheng YF / Manglik A | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2023 ジャーナル: Proc Natl Acad Sci U S A / 年: 2023タイトル: Efficient tagging of endogenous proteins in human cell lines for structural studies by single-particle cryo-EM. 著者: Wooyoung Choi / Hao Wu / Klaus Yserentant / Bo Huang / Yifan Cheng /  要旨: CRISPR/Cas9-based genome engineering has revolutionized our ability to manipulate biological systems, particularly in higher organisms. Here, we designed a set of homology-directed repair donor ...CRISPR/Cas9-based genome engineering has revolutionized our ability to manipulate biological systems, particularly in higher organisms. Here, we designed a set of homology-directed repair donor templates that enable efficient tagging of endogenous proteins with affinity tags by transient transfection and selection of genome-edited cells in various human cell lines. Combined with technological advancements in single-particle cryogenic electron microscopy, this strategy allows efficient structural studies of endogenous proteins captured in their native cellular environment and during different cellular processes. We demonstrated this strategy by tagging six different human proteins in both HEK293T and Jurkat cells. Moreover, analysis of endogenous glyceraldehyde 3-phosphate dehydrogenase (GAPDH) in HEK293T cells allowed us to follow its behavior spatially and temporally in response to prolonged oxidative stress, correlating the increased number of oxidation-induced inactive catalytic sites in GAPDH with its translocation from cytosol to nucleus. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_29663.map.gz emd_29663.map.gz | 39.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-29663-v30.xml emd-29663-v30.xml emd-29663.xml emd-29663.xml | 24.1 KB 24.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_29663.png emd_29663.png | 94.7 KB | ||
| Filedesc metadata |  emd-29663.cif.gz emd-29663.cif.gz | 5.4 KB | ||
| その他 |  emd_29663_additional_1.map.gz emd_29663_additional_1.map.gz emd_29663_additional_2.map.gz emd_29663_additional_2.map.gz emd_29663_additional_3.map.gz emd_29663_additional_3.map.gz emd_29663_additional_4.map.gz emd_29663_additional_4.map.gz emd_29663_additional_5.map.gz emd_29663_additional_5.map.gz emd_29663_half_map_1.map.gz emd_29663_half_map_1.map.gz emd_29663_half_map_2.map.gz emd_29663_half_map_2.map.gz | 1.2 MB 1.3 MB 1.3 MB 1.3 MB 1.2 MB 39.7 MB 39.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-29663 http://ftp.pdbj.org/pub/emdb/structures/EMD-29663 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-29663 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-29663 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_29663.map.gz / 形式: CCP4 / 大きさ: 52.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_29663.map.gz / 形式: CCP4 / 大きさ: 52.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | GAPDH, cyto, oxidative stress 24h, overall, d2 | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.835 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: GAPDH, cyto, oxidative stress 24h, active, single subunit...
| ファイル | emd_29663_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | GAPDH, cyto, oxidative stress 24h, active, single subunit analysis, class4 | ||||||||||||
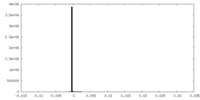
| 投影像・断面図 |
| ||||||||||||
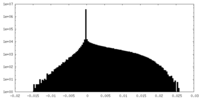
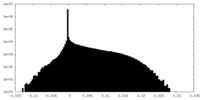
| 密度ヒストグラム |
-追加マップ: GAPDH, cyto, oxidative stress 24h, inactive, single subunit...
| ファイル | emd_29663_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | GAPDH, cyto, oxidative stress 24h, inactive, single subunit analysis, class1 | ||||||||||||
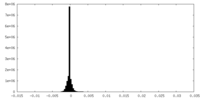
| 投影像・断面図 |
| ||||||||||||
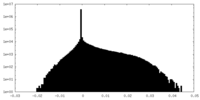
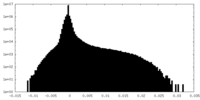
| 密度ヒストグラム |
-追加マップ: GAPDH, cyto, oxidative stress 24h, nondefined, single subunit...
| ファイル | emd_29663_additional_3.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | GAPDH, cyto, oxidative stress 24h, nondefined, single subunit analysis, class2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
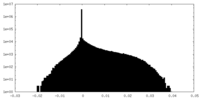
| 密度ヒストグラム |
-追加マップ: GAPDH, cyto, oxidative stress 24h, nondefined, single subunit...
| ファイル | emd_29663_additional_4.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | GAPDH, cyto, oxidative stress 24h, nondefined, single subunit analysis, class3 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
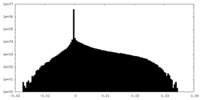
| 密度ヒストグラム |
-追加マップ: GAPDH, cyto, oxidative stress 24h, nondefined, single subunit...
| ファイル | emd_29663_additional_5.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | GAPDH, cyto, oxidative stress 24h, nondefined, single subunit analysis, class5 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: GAPDH, cyto, oxidative stress 24h, halfmap1, d2
| ファイル | emd_29663_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | GAPDH, cyto, oxidative stress 24h, halfmap1, d2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: GAPDH, cyto, oxidative stress 24h, halfmap2, d2
| ファイル | emd_29663_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | GAPDH, cyto, oxidative stress 24h, halfmap2, d2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : GAPDH
| 全体 | 名称: GAPDH Glyceraldehyde 3-phosphate dehydrogenase Glyceraldehyde 3-phosphate dehydrogenase |
|---|---|
| 要素 |
|
-超分子 #1: GAPDH
| 超分子 | 名称: GAPDH / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Glyceraldehyde-3-phosphate dehydrogenase
| 分子 | 名称: Glyceraldehyde-3-phosphate dehydrogenase / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO EC番号:  グリセルアルデヒド-3-リン酸デヒドロゲナーゼ (リン酸化) グリセルアルデヒド-3-リン酸デヒドロゲナーゼ (リン酸化) |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 35.967969 KDa |
| 配列 | 文字列: GKVKVGVNGF GRIGRLVTRA AFNSGKVDIV AINDPFIDLN YMVYMFQYDS THGKFHGTVK AENGKLVING NPITIFQERD PSKIKWGDA GAEYVVESTG VFTTMEKAGA HLQGGAKRVI ISAPSADAPM FVMGVNHEKY DNSLKIISNA SCTTNCLAPL A KVIHDNFG ...文字列: GKVKVGVNGF GRIGRLVTRA AFNSGKVDIV AINDPFIDLN YMVYMFQYDS THGKFHGTVK AENGKLVING NPITIFQERD PSKIKWGDA GAEYVVESTG VFTTMEKAGA HLQGGAKRVI ISAPSADAPM FVMGVNHEKY DNSLKIISNA SCTTNCLAPL A KVIHDNFG IVEGLMTTVH AITATQKTVD GPSGKLWRDG RGALQNIIPA STGAAKAVGK VIPELNGKLT GMAFRVPTAN VS VVDLTCR LEKPAKYDDI KKVVKQASEG PLKGILGYTE HQVVSSDFNS DTHSSTFDAG AGIALNDHFV KLISWYDNEF GYS NRVVDL MAHMASKE UniProtKB:  Glyceraldehyde-3-phosphate dehydrogenase Glyceraldehyde-3-phosphate dehydrogenase |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | cell |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| 凍結 | 凍結剤: OTHER |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 1.5 µm / 最小 デフォーカス(公称値): 0.8 µm Bright-field microscopy / 最大 デフォーカス(公称値): 1.5 µm / 最小 デフォーカス(公称値): 0.8 µm |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 45.8 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: OTHER |
|---|---|
| 初期 角度割当 | タイプ: ANGULAR RECONSTITUTION |
| 最終 角度割当 | タイプ: ANGULAR RECONSTITUTION |
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 2.07 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 598578 |
 ムービー
ムービー コントローラー
コントローラー

















 Z
Z Y
Y X
X