+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of STING oligomer bound to cGAMP, NVS-STG2 and C53 | |||||||||||||||
 マップデータ マップデータ | ||||||||||||||||
 試料 試料 |
| |||||||||||||||
 キーワード キーワード | STING /  innate immunity (自然免疫系) / innate immunity (自然免疫系) /  molecular glue / molecular glue /  IMMUNE SYSTEM (免疫系) IMMUNE SYSTEM (免疫系) | |||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報STING complex / STAT6-mediated induction of chemokines / protein localization to endoplasmic reticulum /  serine/threonine protein kinase complex / 2',3'-cyclic GMP-AMP binding / serine/threonine protein kinase complex / 2',3'-cyclic GMP-AMP binding /  proton channel activity / cyclic-di-GMP binding / STING mediated induction of host immune responses / IRF3-mediated induction of type I IFN / positive regulation of type I interferon-mediated signaling pathway ...STING complex / STAT6-mediated induction of chemokines / protein localization to endoplasmic reticulum / proton channel activity / cyclic-di-GMP binding / STING mediated induction of host immune responses / IRF3-mediated induction of type I IFN / positive regulation of type I interferon-mediated signaling pathway ...STING complex / STAT6-mediated induction of chemokines / protein localization to endoplasmic reticulum /  serine/threonine protein kinase complex / 2',3'-cyclic GMP-AMP binding / serine/threonine protein kinase complex / 2',3'-cyclic GMP-AMP binding /  proton channel activity / cyclic-di-GMP binding / STING mediated induction of host immune responses / IRF3-mediated induction of type I IFN / positive regulation of type I interferon-mediated signaling pathway / cGAS/STING signaling pathway / reticulophagy / pattern recognition receptor signaling pathway / cytoplasmic pattern recognition receptor signaling pathway / cellular response to exogenous dsRNA / autophagosome membrane / antiviral innate immune response / positive regulation of macroautophagy / proton channel activity / cyclic-di-GMP binding / STING mediated induction of host immune responses / IRF3-mediated induction of type I IFN / positive regulation of type I interferon-mediated signaling pathway / cGAS/STING signaling pathway / reticulophagy / pattern recognition receptor signaling pathway / cytoplasmic pattern recognition receptor signaling pathway / cellular response to exogenous dsRNA / autophagosome membrane / antiviral innate immune response / positive regulation of macroautophagy /  autophagosome assembly / cellular response to organic cyclic compound / autophagosome assembly / cellular response to organic cyclic compound /  オートファゴソーム / positive regulation of type I interferon production / cellular response to interferon-beta / signaling adaptor activity / positive regulation of defense response to virus by host / Regulation of innate immune responses to cytosolic DNA / activation of innate immune response / endoplasmic reticulum-Golgi intermediate compartment membrane / positive regulation of interferon-beta production / secretory granule membrane / cytoplasmic vesicle membrane / positive regulation of DNA-binding transcription factor activity / オートファゴソーム / positive regulation of type I interferon production / cellular response to interferon-beta / signaling adaptor activity / positive regulation of defense response to virus by host / Regulation of innate immune responses to cytosolic DNA / activation of innate immune response / endoplasmic reticulum-Golgi intermediate compartment membrane / positive regulation of interferon-beta production / secretory granule membrane / cytoplasmic vesicle membrane / positive regulation of DNA-binding transcription factor activity /  ペルオキシソーム / SARS-CoV-1 activates/modulates innate immune responses / positive regulation of protein binding / protein complex oligomerization / ペルオキシソーム / SARS-CoV-1 activates/modulates innate immune responses / positive regulation of protein binding / protein complex oligomerization /  regulation of inflammatory response / defense response to virus / RNA polymerase II-specific DNA-binding transcription factor binding / mitochondrial outer membrane / regulation of inflammatory response / defense response to virus / RNA polymerase II-specific DNA-binding transcription factor binding / mitochondrial outer membrane /  エンドソーム / エンドソーム /  ゴルジ体 / ゴルジ体 /  自然免疫系 / 自然免疫系 /  ubiquitin protein ligase binding / Neutrophil degranulation / endoplasmic reticulum membrane / ubiquitin protein ligase binding / Neutrophil degranulation / endoplasmic reticulum membrane /  protein kinase binding / SARS-CoV-2 activates/modulates innate and adaptive immune responses / perinuclear region of cytoplasm / protein homodimerization activity / positive regulation of transcription by RNA polymerase II / protein kinase binding / SARS-CoV-2 activates/modulates innate and adaptive immune responses / perinuclear region of cytoplasm / protein homodimerization activity / positive regulation of transcription by RNA polymerase II /  核質 / identical protein binding / 核質 / identical protein binding /  細胞膜 / 細胞膜 /  細胞質基質 細胞質基質類似検索 - 分子機能 | |||||||||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 2.9 Å クライオ電子顕微鏡法 / 解像度: 2.9 Å | |||||||||||||||
 データ登録者 データ登録者 | Li J / Canham SM / Zhang X / Bai X / Feng Y | |||||||||||||||
| 資金援助 |  米国, 4件 米国, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Nat Chem Biol / 年: 2024 ジャーナル: Nat Chem Biol / 年: 2024タイトル: Activation of human STING by a molecular glue-like compound. 著者: Jie Li / Stephen M Canham / Hua Wu / Martin Henault / Lihao Chen / Guoxun Liu / Yu Chen / Gary Yu / Howard R Miller / Viktor Hornak / Scott M Brittain / Gregory A Michaud / Antonin Tutter / ...著者: Jie Li / Stephen M Canham / Hua Wu / Martin Henault / Lihao Chen / Guoxun Liu / Yu Chen / Gary Yu / Howard R Miller / Viktor Hornak / Scott M Brittain / Gregory A Michaud / Antonin Tutter / Wendy Broom / Mary Ellen Digan / Sarah M McWhirter / Kelsey E Sivick / Helen T Pham / Christine H Chen / George S Tria / Jeffery M McKenna / Markus Schirle / Xiaohong Mao / Thomas B Nicholson / Yuan Wang / Jeremy L Jenkins / Rishi K Jain / John A Tallarico / Sejal J Patel / Lianxing Zheng / Nathan T Ross / Charles Y Cho / Xuewu Zhang / Xiao-Chen Bai / Yan Feng /  要旨: Stimulator of interferon genes (STING) is a dimeric transmembrane adapter protein that plays a key role in the human innate immune response to infection and has been therapeutically exploited for its ...Stimulator of interferon genes (STING) is a dimeric transmembrane adapter protein that plays a key role in the human innate immune response to infection and has been therapeutically exploited for its antitumor activity. The activation of STING requires its high-order oligomerization, which could be induced by binding of the endogenous ligand, cGAMP, to the cytosolic ligand-binding domain. Here we report the discovery through functional screens of a class of compounds, named NVS-STGs, that activate human STING. Our cryo-EM structures show that NVS-STG2 induces the high-order oligomerization of human STING by binding to a pocket between the transmembrane domains of the neighboring STING dimers, effectively acting as a molecular glue. Our functional assays showed that NVS-STG2 could elicit potent STING-mediated immune responses in cells and antitumor activities in animal models. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_29282.map.gz emd_29282.map.gz | 4.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-29282-v30.xml emd-29282-v30.xml emd-29282.xml emd-29282.xml | 17.8 KB 17.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_29282_fsc.xml emd_29282_fsc.xml | 7.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_29282.png emd_29282.png | 100 KB | ||
| Filedesc metadata |  emd-29282.cif.gz emd-29282.cif.gz | 6.1 KB | ||
| その他 |  emd_29282_half_map_1.map.gz emd_29282_half_map_1.map.gz emd_29282_half_map_2.map.gz emd_29282_half_map_2.map.gz | 28.4 MB 28.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-29282 http://ftp.pdbj.org/pub/emdb/structures/EMD-29282 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-29282 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-29282 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8flmMC  8flkC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_29282.map.gz / 形式: CCP4 / 大きさ: 38.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_29282.map.gz / 形式: CCP4 / 大きさ: 38.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.08 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #1
| ファイル | emd_29282_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
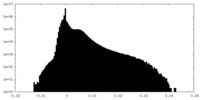
| 投影像・断面図 |
| ||||||||||||
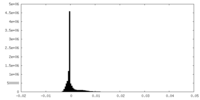
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_29282_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
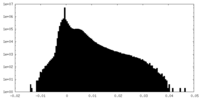
| 投影像・断面図 |
| ||||||||||||
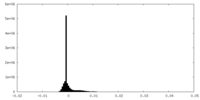
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : STING tetramer
| 全体 | 名称: STING tetramer |
|---|---|
| 要素 |
|
-超分子 #1: STING tetramer
| 超分子 | 名称: STING tetramer / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 詳細: STING tetramer, which can further assemblies into larger oligomers, induced by cGAMP, NVS-STG2 and C53 |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 300 KDa |
-分子 #1: Stimulator of interferon genes protein
| 分子 | 名称: Stimulator of interferon genes protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 39.553398 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MPHSSLHPSI PCPRGHGAQK AALVLLSACL VTLWGLGEPP EHTLRYLVLH LASLQLGLLL NGVCSLAEEL RHIHSRYRGS YWRTVRACL GCPLRRGALL LLSIYFYYSL PNAVGPPFTW MLALLGLSQA LNILLGLKGL APAEISAVCE KGNFNVAHGL A WSYYIGYL ...文字列: MPHSSLHPSI PCPRGHGAQK AALVLLSACL VTLWGLGEPP EHTLRYLVLH LASLQLGLLL NGVCSLAEEL RHIHSRYRGS YWRTVRACL GCPLRRGALL LLSIYFYYSL PNAVGPPFTW MLALLGLSQA LNILLGLKGL APAEISAVCE KGNFNVAHGL A WSYYIGYL RLILPELQAR IRTYNQHYNN LLRGAVSQRL YILLPLDCGV PDNLSMADPN IRFLDKLPQQ TGDRAGIKDR VY SNSIYEL LENGQRAGTC VLEYATPLQT LFAMSQYSQA GFSREDRLEQ AKLFCRTLED ILADAPESQN NCRLIAYQEP ADD SSFSLS QEVLRHLRQE EKEEVTVGTS SGLEVLFQ UniProtKB:  Stimulator of interferon genes protein Stimulator of interferon genes protein |
-分子 #2: 4-({[4-(2-tert-butyl-5,5-dimethyl-1,3-dioxan-2-yl)phenyl]methyl}a...
| 分子 | 名称: 4-({[4-(2-tert-butyl-5,5-dimethyl-1,3-dioxan-2-yl)phenyl]methyl}amino)-3-methoxybenzoic acid タイプ: ligand / ID: 2 / コピー数: 2 / 式: Y6H |
|---|---|
| 分子量 | 理論値: 427.533 Da |
| Chemical component information |  ChemComp-Y6H: |
-分子 #3: cGAMP
| 分子 | 名称: cGAMP / タイプ: ligand / ID: 3 / コピー数: 2 / 式: 1SY |
|---|---|
| 分子量 | 理論値: 674.411 Da |
| Chemical component information | 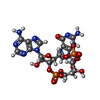 ChemComp-1SY: |
-分子 #4: 1-[(2-chloro-6-fluorophenyl)methyl]-3,3-dimethyl-2-oxo-N-[(2,4,6-...
| 分子 | 名称: 1-[(2-chloro-6-fluorophenyl)methyl]-3,3-dimethyl-2-oxo-N-[(2,4,6-trifluorophenyl)methyl]-2,3-dihydro-1H-indole-6-carboxamide タイプ: ligand / ID: 4 / コピー数: 2 / 式: 9IM |
|---|---|
| 分子量 | 理論値: 490.877 Da |
| Chemical component information | 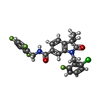 ChemComp-9IM: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: NITROGEN |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.5 µm Bright-field microscopy / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.5 µm |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 60.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: EMDB MAP EMDB ID: |
|---|---|
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 2.9 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 292483 |
FSC曲線 (解像度の算出) | 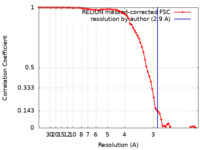 |
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL |
|---|---|
| 得られたモデル |  PDB-8flm: |
 ムービー
ムービー コントローラー
コントローラー









 Z
Z Y
Y X
X