+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of LRP2 at pH 7.5 | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Transport of RCbl within the body / endocytic hemoglobin import into cell / diol metabolic process / chemoattraction of axon / positive regulation of lipoprotein transport / pulmonary artery morphogenesis / secondary heart field specification / Retinoid metabolism and transport / positive regulation of oligodendrocyte progenitor proliferation / folate import across plasma membrane ...Transport of RCbl within the body / endocytic hemoglobin import into cell / diol metabolic process / chemoattraction of axon / positive regulation of lipoprotein transport / pulmonary artery morphogenesis / secondary heart field specification / Retinoid metabolism and transport / positive regulation of oligodendrocyte progenitor proliferation / folate import across plasma membrane / metal ion transport / response to leptin / ventricular compact myocardium morphogenesis / protein transporter activity /  hormone binding / Cargo recognition for clathrin-mediated endocytosis / vitamin D metabolic process / neuron projection arborization / hormone binding / Cargo recognition for clathrin-mediated endocytosis / vitamin D metabolic process / neuron projection arborization /  Clathrin-mediated endocytosis / hormone secretion / coronary artery morphogenesis / outflow tract septum morphogenesis / Clathrin-mediated endocytosis / hormone secretion / coronary artery morphogenesis / outflow tract septum morphogenesis /  insulin-like growth factor I binding / insulin-like growth factor I binding /  transcytosis / protein import / coronary vasculature development / cargo receptor activity / aorta development / positive regulation of neurogenesis / ventricular septum development / endosomal transport / low-density lipoprotein particle receptor binding / transcytosis / protein import / coronary vasculature development / cargo receptor activity / aorta development / positive regulation of neurogenesis / ventricular septum development / endosomal transport / low-density lipoprotein particle receptor binding /  hemoglobin binding / amyloid-beta clearance / positive regulation of endocytosis / vagina development / hemoglobin binding / amyloid-beta clearance / positive regulation of endocytosis / vagina development /  刷子縁 / negative regulation of BMP signaling pathway / response to X-ray / endocytic vesicle / axonal growth cone / 刷子縁 / negative regulation of BMP signaling pathway / response to X-ray / endocytic vesicle / axonal growth cone /  クラスリン / forebrain development / クラスリン / forebrain development /  receptor-mediated endocytosis / receptor-mediated endocytosis /  kidney development / neural tube closure / endosome lumen / kidney development / neural tube closure / endosome lumen /  PDZ domain binding / PDZ domain binding /  nuclear receptor binding / brush border membrane / sensory perception of sound / cellular response to growth factor stimulus / nuclear receptor binding / brush border membrane / sensory perception of sound / cellular response to growth factor stimulus /  SH3 domain binding / SH3 domain binding /  エンドサイトーシス / male gonad development / エンドサイトーシス / male gonad development /  protein transport / apical part of cell / protein transport / apical part of cell /  heart development / protein-folding chaperone binding / cell population proliferation / positive regulation of phosphatidylinositol 3-kinase/protein kinase B signal transduction / heart development / protein-folding chaperone binding / cell population proliferation / positive regulation of phosphatidylinositol 3-kinase/protein kinase B signal transduction /  receptor complex / receptor complex /  エンドソーム / エンドソーム /  脂質ラフト / apical plasma membrane / external side of plasma membrane / 脂質ラフト / apical plasma membrane / external side of plasma membrane /  神経繊維 / 神経繊維 /  樹状突起 / 樹状突起 /  calcium ion binding / protein-containing complex binding / negative regulation of apoptotic process / calcium ion binding / protein-containing complex binding / negative regulation of apoptotic process /  ゴルジ体 / ゴルジ体 /  細胞膜 / 細胞膜 /  小胞体 / protein-containing complex / 小胞体 / protein-containing complex /  extracellular space / extracellular space /  生体膜 / 生体膜 /  細胞膜 / 細胞膜 /  細胞質 細胞質類似検索 - 分子機能 | |||||||||
| 生物種 |   Mus musculus (ハツカネズミ) / Mus musculus (ハツカネズミ) /   house mouse (ハツカネズミ) house mouse (ハツカネズミ) | |||||||||
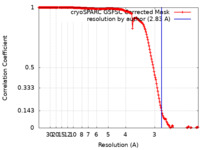
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 2.83 Å クライオ電子顕微鏡法 / 解像度: 2.83 Å | |||||||||
 データ登録者 データ登録者 | Beenken A / Cerutti G / Brasch J / Fitzpatrick AW / Barasch J / Shapiro L | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Cell / 年: 2023 ジャーナル: Cell / 年: 2023タイトル: Structures of LRP2 reveal a molecular machine for endocytosis. 著者: Andrew Beenken / Gabriele Cerutti / Julia Brasch / Yicheng Guo / Zizhang Sheng / Hediye Erdjument-Bromage / Zainab Aziz / Shelief Y Robbins-Juarez / Estefania Y Chavez / Goran Ahlsen / ...著者: Andrew Beenken / Gabriele Cerutti / Julia Brasch / Yicheng Guo / Zizhang Sheng / Hediye Erdjument-Bromage / Zainab Aziz / Shelief Y Robbins-Juarez / Estefania Y Chavez / Goran Ahlsen / Phinikoula S Katsamba / Thomas A Neubert / Anthony W P Fitzpatrick / Jonathan Barasch / Lawrence Shapiro /  要旨: The low-density lipoprotein (LDL) receptor-related protein 2 (LRP2 or megalin) is representative of the phylogenetically conserved subfamily of giant LDL receptor-related proteins, which function in ...The low-density lipoprotein (LDL) receptor-related protein 2 (LRP2 or megalin) is representative of the phylogenetically conserved subfamily of giant LDL receptor-related proteins, which function in endocytosis and are implicated in diseases of the kidney and brain. Here, we report high-resolution cryoelectron microscopy structures of LRP2 isolated from mouse kidney, at extracellular and endosomal pH. The structures reveal LRP2 to be a molecular machine that adopts a conformation for ligand binding at the cell surface and for ligand shedding in the endosome. LRP2 forms a homodimer, the conformational transformation of which is governed by pH-sensitive sites at both homodimer and intra-protomer interfaces. A subset of LRP2 deleterious missense variants in humans appears to impair homodimer assembly. These observations lay the foundation for further understanding the function and mechanism of LDL receptors and implicate homodimerization as a conserved feature of the LRP receptor subfamily. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_28233.map.gz emd_28233.map.gz | 254.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-28233-v30.xml emd-28233-v30.xml emd-28233.xml emd-28233.xml | 24.9 KB 24.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_28233_fsc.xml emd_28233_fsc.xml | 16.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_28233.png emd_28233.png | 73.9 KB | ||
| その他 |  emd_28233_additional_1.map.gz emd_28233_additional_1.map.gz emd_28233_additional_2.map.gz emd_28233_additional_2.map.gz emd_28233_half_map_1.map.gz emd_28233_half_map_1.map.gz emd_28233_half_map_2.map.gz emd_28233_half_map_2.map.gz | 99.2 MB 241.7 MB 474.9 MB 474.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-28233 http://ftp.pdbj.org/pub/emdb/structures/EMD-28233 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-28233 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-28233 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8em4MC  8em7C C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_28233.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_28233.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.08 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: Composite of unsharpened local refinement maps
| ファイル | emd_28233_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Composite of unsharpened local refinement maps | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Composite map of the C2 global map with...
| ファイル | emd_28233_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Composite map of the C2 global map with deepEMhancer density-modified local refinements | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_28233_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_28233_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : LRP2 at neutral pH
| 全体 | 名称: LRP2 at neutral pH |
|---|---|
| 要素 |
|
-超分子 #1: LRP2 at neutral pH
| 超分子 | 名称: LRP2 at neutral pH / タイプ: complex / ID: 1 / キメラ: Yes / 親要素: 0 / 含まれる分子: #1 / 詳細: Endogenously purified from mouse kidney |
|---|---|
| 由来(天然) | 生物種:   Mus musculus (ハツカネズミ) Mus musculus (ハツカネズミ) |
-分子 #1: Low-density lipoprotein receptor-related protein 2
| 分子 | 名称: Low-density lipoprotein receptor-related protein 2 / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   house mouse (ハツカネズミ) house mouse (ハツカネズミ) |
| 分子量 | 理論値: 519.7465 KDa |
| 配列 | 文字列: MERGAAAAAW MLLLAIAACL APVSGQECGS GNFRCDNGYC IPASWRCDGT RDCLDDTDEI GCPPRSCGSG FFLCPAEGTC IPSSWVCDQ DKDCSDGADE QQNCPGTTCS SQQLTCSNGQ CVPIEYRCDH VSDCPDGSDE RNCYYPTCDQ LTCANGACYN T SQKCDHKV ...文字列: MERGAAAAAW MLLLAIAACL APVSGQECGS GNFRCDNGYC IPASWRCDGT RDCLDDTDEI GCPPRSCGSG FFLCPAEGTC IPSSWVCDQ DKDCSDGADE QQNCPGTTCS SQQLTCSNGQ CVPIEYRCDH VSDCPDGSDE RNCYYPTCDQ LTCANGACYN T SQKCDHKV DCRDSSDEAN CTTLCSQKEF QCGSGECILR AYVCDHDNDC EDNSDEHNCN YDTCGGHQFT CSNGQCINQN WV CDGDDDC QDSGDEDGCE SNQRHHTCYP REWACPGSGR CISMDKVCDG VPDCPEGEDE NNATSGRYCG TGLCSILNCE YQC HQTPYG GECFCPPGHI INSNDSRTCI DFDDCQIWGI CDQKCESRQG RHQCLCEEGY ILERGQHCKS NDSFSAASII FSNG RDLLV GDLHGRNFRI LAESKNRGIV MGVDFHYQKH RVFWTDPMQA KVFSTDINGL NTQEILNVSI DAPENLAVDW INNKL YLVE TRVNRIDVVN LEGNQRVTLI TENLGHPRGI ALDPTVGYLF FSDWGSLSGQ PKVERAFMDG SNRKDLVTTK LGWPAG ITL DLVSKRVYWV DSRYDYIETV TYDGIQRKTV ARGGSLVPHP FGISLFEEHV FFTDWTKMAV MKANKFTDTN PQVYHQS SL TPFGVTVYHA LRQPNATNPC GNNNGGCAQI CVLSHRTDNG GLGYRCKCEF GFELDADEHH CVAVKNFLLF SSQTAVRG I PFTLSTQEDV MVPVTGSPSF FVGIDFDAQH STIFYSDLSK NIIYQQKIDG TGKEVITANR LQNVECLSFD WISRNLYWT DGGSKSVTVM KLADKSRRQI ISNLNNPRSI VVHPAAGYMF LSDWFRPAKI MRAWSDGSHL MPIVNTSLGW PNGLAIDWST SRLYWVDAF FDKIEHSNLD GLDRKRLGHV DQMTHPFGLT VFKDNVFLTD WRLGAIIRVR KSDGGDMTVV RRGISSIMHV K AYDADLQT GTNYCSQTTH PNGDCSHFCF PVPNFQRVCG CPYGMKLQRD QMTCEGDPAR EPPTQQCGSS SFPCNNGKCV PS IFRCDGV DDCHDNSDEH QCGALNNTCS SSAFTCVHGG QCIPGQWRCD KQNDCLDGSD EQNCPTRSPS STCPPTSFTC DNH MCIPKE WVCDTDNDCS DGSDEKNCQA SGTCHPTQFR CPDHRCISPL YVCDGDKDCV DGSDEAGCVL NCTSSQFKCA DGSS CINSR YRCDGVYDCK DNSDEAGCPT RPPGMCHPDE FQCQGDGTCI PNTWECDGHP DCIQGSDEHN GCVPKTCSPS HFLCD NGNC IYNSWVCDGD NDCRDMSDEK DCPTQPFHCP SSQWQCPGYS ICVNLSALCD GVFDCPNGTD ESPLCNQDSC LHFNGG CTH RCIQGPFGAT CVCPIGYQLA NDTKTCEDVN ECDIPGFCSQ HCVNMRGSFR CACDPEYTLE SDGRTCKVTA SENLLLV VA SRDKIIMDNI TAHTHNIYSL VQDVSFVVAL DFDSVTGRVF WSDLLEGKTW SAFQNGTDKR VVHDSGLSLT EMIAVDWI G RNIYWTDYTL ETIEVSKIDG SHRTVLISKN VTKPRGLALD PRMGDNVMFW SDWGHHPRIE RASMDGTMRT VIVQEKIYW PCGLSIDYPN RLIYFMDAYL DYIEFCDYDG QNRRQVIASD LVLHHPHALT LFEDSVFWTD RGTHQVMQAN KWHGRNQSVV MYSVPQPLG IIAIHPSRQP SSPNPCASAT CSHLCLLSAQ EPRHYSCACP SGWNLSDDSV NCVRGDQPFL ISVRENVIFG I SLDPEVKS NDAMVPISGI QHGYDVEFDD SEQFIYWVEN PGEIHRVKTD GSNRTAFAPL SLLGSSLGLA LDWVSRNIYY TT PASRSIE VLTLRGDTRY GKTLITNDGT PLGVGFPVGI AVDPARGKLY WSDHGTDSGV PAKIASANMD GTSLKILFTG NME HLEVVT LDIQEQKLYW AVTSRGVIER GNVDGTERMI LVHHLAHPWG LVVHGSFLYY SDEQYEVIER VDKSSGSNKV VFRD NIPYL RGLRVYHHRN AADSSNGCSN NPNACQQICL PVPGGMFSCA CASGFKLSPD GRSCSPYNSF IVVSMLPAVR GFSLE LSDH SEAMVPVAGQ GRNVLHADVD VANGFIYWCD FSSSVRSSNG IRRIKPNGSN FTNIVTYGIG ANGIRGVAVD WVAGNL YFT NAFVYETLIE VIRINTTYRR VLLKVSVDMP RHIVVDPKHR YLFWADYGQK PKIERSFLDC TNRTVLVSEG IVTPRGL AV DHDTGYIYWV DDSLDIIARI HRDGGESQVV RYGSRYPTPY GITVFGESII WVDRNLRKVF QASKQPGNTD PPTVIRDS I NLLRDVTIFD EHVQPLSPAE LNNNPCLQSN GGCSHFCFAL PELPTPKCGC AFGTLEDDGK NCATSREDFL IYSLNNSLR SLHFDPQDHN LPFQAISVEG MAIALDYDRR NNRIFFTQKL NPIRGQISYV NLYSGASSPT ILLSNIGVTD GIAFDWINRR IYYSDFSNQ TINSMAEDGS NRAVIARVSK PRAIVLDPCR GYMYWTDWGT NAKIERATLG GNFRVPIVNT SLVWPNGLTL D LETDLLYW ADASLQKIER STLTGSNREV VISTAFHSFG LTVYGQYIYW TDFYTKKIYR ANKYDGSDLI AMTTRLPTQP SG ISTVVKT QQQQCSNPCD QFNGGCSHIC APGPNGAECQ CPHEGSWYLA NDNKYCVVDT GARCNQFQFT CLNGRCISQD WKC DNDNDC GDGSDELPTV CAFHTCRSTA FTCANGRCVP YHYRCDFYND CGDNSDEAGC LFRSCNSTTE FTCSNGRCIP LSYV CNGIN NCHDNDTSDE KNCPPITCQP DFAKCQTTNI CVPRAFLCDG DNDCGDGSDE NPIYCASHTC RSNEFQCVSP HRCIP SYWF CDGEADCVDS SDEPDTCGHS LNSCSANQFH CDNGRCISSS WVCDGDNDCG DMSDEDQRHH CELQNCSSTE FTCINS RPP NRRCIPQHWV CDGDADCADA LDELQNCTMR ACSTGEFSCA NGRCIRQSFR CDRRNDCGDY SDERGCSYPP CRDDQFT CQ NGQCITKLYV CDEDNDCGDG SDEQEHLCHT PEPTCPPHQF RCDNGHCIEM GTVCNHVDDC SDNSDEKGCG INECQDSS I SHCDHNCTDT ITSFYCSCLP GYKLMSDKRT CVDIDECKET PQLCSQKCEN VIGSYICKCA PGYIREPDGK SCRQNSNIE PYLVFSNRYY IRNLTIDGTS YSLILQGLGN VVALDFDRVE ERLYWIDAEK QIIERMFLNK TNQETIISHR LRRAESLAVD WVSRKLYWL DAILDCLFVS DLEGRQRKML AQHCVDANNT FCFENPRGIV LHPQRGYVYW ADWGDHAYIA RIGMDGTNKT V IISTKIEW PNAITIDYTN DLLYWADAHL GYIEFSDLEG HHRHTVYDGT LPHPFALTIF EDTVFWTDWN TRTVEKGNKY DG SGRVVLV NTTHKPFDIH VLHPYRQPIM SNPCATNNGG CSHLCLIKAG GRGFTCECPD DFQTVQLRDR TLCMPMCSST QFL CGNNEK CIPIWWKCDG QKDCSDGSDE SDLCPHRFCR LGQFQCRDGN CTSPQALCNA RQDCADGSDE DRVLCEHHRC EANE WQCAN KRCIPEYWQC DSVDDCLDNS DEDPSHCASR TCRPGQFKCN NGRCIPQSWK CDVDNDCGDY SDEPIHECMT AAYNC DNHT EFSCKTNYRC IPQWAVCNGF DDCRDNSDEQ GCESVPCHPS GDFRCGNHHC IPLRWKCDGI DDCGDNSDEE SCVPRE CTE SEFRCADQQC IPSRWVCDQE NDCGDNSDER DCEMKTCHPE HFQCTSGHCV PKALACDGRA DCLDASDESA CPTRFPN GT YCPAAMFECK NHVCIQSFWI CDGENDCVDG SDEEIHLCFN VPCESPQRFR CDNSRCIYGH QLCNGVDDCG DGSDEKEE H CRKPTHKPCT DTEYKCSNGN CVSQHYVCDN VDDCGDLSDE TGCNLGENRT CAEKICEQNC TQLSNGGFIC SCRPGFKPS TLDKNSCQDI NECEEFGICP QSCRNSKGSY ECFCVDGFKS MSTHYGERCA ADGSPPLLLL PENVRIRKYN ISSEKFSEYL EEEEHIQAI DYDWDPEGIG LSVVYYTVLS QGSQFGAIKR AYLPDFESGS NNPVREVDLG LKYLMQPDGL AVDWVGRHIY W SDAKSQRI EVATLDGRYR KWLITTQLDQ PAAIAVNPKL GLMFWTDQGK QPKIESAWMN GEHRSVLASA NLGWPNGLSI DY LNGDRIY WSDSKEDVIE SIKYDGTDRR LIINDAMKPF SLDIFEDQLY WVAKEKGEVW RQNKFGKGNK EKLLVVNPWL TQV RIFHQL RYNQSVSNPC KQVCSHLCLL RPGGYSCACP QGSDFVTGST VECDAASELP ITMPSPCRCM HGGSCYFDEN DLPK CKCSS GYSGEYCEIG LSRGIPPGTT MALLLTFAMV IIVGALVLVG FFHYRKTGSL LPSLPKLPSL SSLAKPSENG NGVTF RSGA DVNMDIGVSP FGPETIIDRS MAMNEQFVME VGKQPVIFEN PMYAAKDSTS KVGLAVQGPS VSSQVTVPEN VENQNY GRS IDPSEIVPEP KPASPGADET QGTKWNIFKR KPKQTTNFEN PIYAEMDTEQ KEAVAVAPPP SPSLPAKASK RSSTPGY TA TEDTFKDTAN LVKEDSDV |
-分子 #3: 2-acetamido-2-deoxy-beta-D-galactopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-galactopyranose / タイプ: ligand / ID: 3 / コピー数: 32 / 式: NGA |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information | 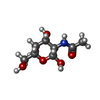 ChemComp-NGA: |
-分子 #4: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 4 / コピー数: 58 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-分子 #5: CALCIUM ION
| 分子 | 名称: CALCIUM ION / タイプ: ligand / ID: 5 / コピー数: 84 / 式: CA |
|---|---|
| 分子量 | 理論値: 40.078 Da |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 1.5 µm / 最小 デフォーカス(公称値): 0.5 µm Bright-field microscopy / 最大 デフォーカス(公称値): 1.5 µm / 最小 デフォーカス(公称値): 0.5 µm |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 54.87 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 精密化 | プロトコル: FLEXIBLE FIT |
|---|---|
| 得られたモデル |  PDB-8em4: |
 ムービー
ムービー コントローラー
コントローラー

























 Z
Z Y
Y X
X