+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of Lates calcarifer DNA polymerase theta polymerase domain with long duplex DNA, complex Ia | |||||||||
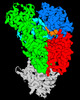
 マップデータ マップデータ | Structure of Lates calcarifer DNA polymerase theta polymerase domain with long duplex DNA, complex Ia | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |   Lates calcarifer (あかめ) Lates calcarifer (あかめ) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 2.4 Å クライオ電子顕微鏡法 / 解像度: 2.4 Å | |||||||||
 データ登録者 データ登録者 | Li C / Zhu H / Sun J / Gao Y | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nucleic Acids Res / 年: 2023 ジャーナル: Nucleic Acids Res / 年: 2023タイトル: Structural basis of DNA polymerase θ mediated DNA end joining. 著者: Chuxuan Li / Hanwen Zhu / Shikai Jin / Leora M Maksoud / Nikhil Jain / Ji Sun / Yang Gao /  要旨: DNA polymerase θ (Pol θ) plays an essential role in the microhomology-mediated end joining (MMEJ) pathway for repairing DNA double-strand breaks. However, the mechanisms by which Pol θ recognizes ...DNA polymerase θ (Pol θ) plays an essential role in the microhomology-mediated end joining (MMEJ) pathway for repairing DNA double-strand breaks. However, the mechanisms by which Pol θ recognizes microhomologous DNA ends and performs low-fidelity DNA synthesis remain unclear. Here, we present cryo-electron microscope structures of the polymerase domain of Lates calcarifer Pol θ with long and short duplex DNA at up to 2.4 Å resolution. Interestingly, Pol θ binds to long and short DNA substrates similarly, with extensive interactions around the active site. Moreover, Pol θ shares a similar active site as high-fidelity A-family polymerases with its finger domain well-closed but differs in having hydrophilic residues surrounding the nascent base pair. Computational simulations and mutagenesis studies suggest that the unique insertion loops of Pol θ help to stabilize short DNA binding and assemble the active site for MMEJ repair. Taken together, our results illustrate the structural basis of Pol θ-mediated MMEJ. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_28075.map.gz emd_28075.map.gz | 59.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-28075-v30.xml emd-28075-v30.xml emd-28075.xml emd-28075.xml | 17.5 KB 17.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_28075.png emd_28075.png | 103.1 KB | ||
| その他 |  emd_28075_half_map_1.map.gz emd_28075_half_map_1.map.gz emd_28075_half_map_2.map.gz emd_28075_half_map_2.map.gz | 59.3 MB 59.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-28075 http://ftp.pdbj.org/pub/emdb/structures/EMD-28075 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-28075 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-28075 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_28075.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_28075.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Structure of Lates calcarifer DNA polymerase theta polymerase domain with long duplex DNA, complex Ia | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.06 Å | ||||||||||||||||||||
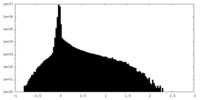
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: Structure of Lates calcarifer DNA polymerase theta polymerase...
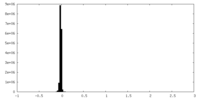
| ファイル | emd_28075_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Structure of Lates calcarifer DNA polymerase theta polymerase domain with long duplex DNA, complex Ia | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Structure of Lates calcarifer DNA polymerase theta polymerase...
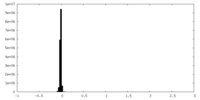
| ファイル | emd_28075_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Structure of Lates calcarifer DNA polymerase theta polymerase domain with long duplex DNA, complex Ia | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Ternary complex of Lates calcarifer DNA polymerase Theta with dup...
| 全体 | 名称: Ternary complex of Lates calcarifer DNA polymerase Theta with duplex DNA and incoming nucleotide |
|---|---|
| 要素 |
|
-超分子 #1: Ternary complex of Lates calcarifer DNA polymerase Theta with dup...
| 超分子 | 名称: Ternary complex of Lates calcarifer DNA polymerase Theta with duplex DNA and incoming nucleotide タイプ: complex / ID: 1 / キメラ: Yes / 親要素: 0 / 含まれる分子: #1-#3 |
|---|---|
| 由来(天然) | 生物種:   Lates calcarifer (あかめ) Lates calcarifer (あかめ) |
-分子 #1: DNA polymerase theta
| 分子 | 名称: DNA polymerase theta / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Lates calcarifer (あかめ) Lates calcarifer (あかめ) |
| 分子量 | 理論値: 95.674117 KDa |
| 組換発現 | 生物種:   Escherichia coli 'BL21-Gold(DE3)pLysS AG' (大腸菌) Escherichia coli 'BL21-Gold(DE3)pLysS AG' (大腸菌) |
| 配列 | 文字列: MGHHHHHHPS GVKTENNDHI NLKVAGQDGS VVQFKIKRHT PLSKLMKAYC ERQGLSMRQI RFRFDGQPIN ETDTPAQLEM EDEDTIDVF QQQTGGCMDP PSDAESPVTD DGFTLQLSQD ASLCPSNSGT FSIIDVASDR RLFNTFIKEW KTKERYSLAL A CEKREHIQ ...文字列: MGHHHHHHPS GVKTENNDHI NLKVAGQDGS VVQFKIKRHT PLSKLMKAYC ERQGLSMRQI RFRFDGQPIN ETDTPAQLEM EDEDTIDVF QQQTGGCMDP PSDAESPVTD DGFTLQLSQD ASLCPSNSGT FSIIDVASDR RLFNTFIKEW KTKERYSLAL A CEKREHIQ QPEGEIGGKH KRAPAARQKL NRTDGFPVRD SDGLVLIGLS VCWGARDSYY ISLQQEQSKG LSSSLAPPPL DD DLPVSER LGQVRSCLSR PSAGLRGGVV VTYDIIQVYK TLVLSCGISL AGNCEDPKVA CWLLDPGSEE RTLPNMVTVY CPE ELPLLD GLGSAHAHCP RVRAATKSVL VHAVMNHLTG LLEKDSMLDL FRSIEMPSQV CLALLELNGV GFSVEECERQ KHVM QAKLT ALESQAYNLA GHSFSLTSID DIAQVLFLEL HLPPNGDVGG SKSKKTLGYT RRGGGRVRLG KQFSTTKDIL EKLRP LHPL PGVILEWRRI TNALTKVVFP LQREKQYHPT LAMDRIYPIA QTHTATGRVS FTEPNIQNVP KDFEICMPTV VGESPP SQN GCQMTTKPGK NRRSVAPSVT GGAAEQGPAF SVSMRHAFVP FSGGMILAAD YSQLELRVLA HLSKDQRLLQ VLNGGAD VF RCIAAEWKGV DPETVNDSLR QQAKQICYGI IYGMGAKSLG EQMGVEENDA ACYIESFKAR YKGINAFLKE TVKNCIKN G YVQTLMGRRR YLPGISNTNT HIKAHAERQA VNTTVQGSAA DIVKLATVNI QKRLRKTYPT APLSHQHTHS GTSQYRAGT SHLRGAFFVL QLHDELIYET REEDLIQVAQ IVKREMESAV KLYVKLKAKV KVGPSWGNLQ DLDL |
-分子 #2: DNA (5'-D(*AP*GP*CP*TP*CP*TP*AP*CP*GP*GP*AP*TP*GP*C)-3')
| 分子 | 名称: DNA (5'-D(*AP*GP*CP*TP*CP*TP*AP*CP*GP*GP*AP*TP*GP*C)-3') タイプ: dna / ID: 2 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:   Lates calcarifer (あかめ) Lates calcarifer (あかめ) |
| 分子量 | 理論値: 8.864712 KDa |
| 配列 | 文字列: (DG)(DC)(DA)(DG)(DT)(DC)(DA)(DG)(DC)(DT) (DC)(DT)(DA)(DC)(DG)(DG)(DA)(DT)(DG)(DC) (DC)(DT)(DC)(DA)(DC)(DA)(DG)(DC)(DA) |
-分子 #3: DNA (5'-D(*AP*GP*CP*AP*TP*CP*CP*GP*TP*AP*GP*(2DA))-3')
| 分子 | 名称: DNA (5'-D(*AP*GP*CP*AP*TP*CP*CP*GP*TP*AP*GP*(2DA))-3') タイプ: dna / ID: 3 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:   Lates calcarifer (あかめ) Lates calcarifer (あかめ) |
| 分子量 | 理論値: 5.844796 KDa |
| 配列 | 文字列: (DT)(DG)(DC)(DT)(DG)(DT)(DG)(DA)(DG)(DC) (DA)(DT)(DC)(DC)(DG)(DT)(DA)(DG)(2DA) |
-分子 #4: 2'-DEOXYGUANOSINE-5'-TRIPHOSPHATE
| 分子 | 名称: 2'-DEOXYGUANOSINE-5'-TRIPHOSPHATE / タイプ: ligand / ID: 4 / コピー数: 1 / 式: DGT |
|---|---|
| 分子量 | 理論値: 507.181 Da |
| Chemical component information | 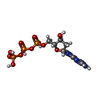 ChemComp-DGT: |
-分子 #5: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 5 / コピー数: 1 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.8 mg/mL |
|---|---|
| 緩衝液 | pH: 7.6 |
| グリッド | モデル: UltrAuFoil R1.2/1.3 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 60 sec. |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 295 K |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 1.6 µm / 最小 デフォーカス(公称値): 0.8 µm Bright-field microscopy / 最大 デフォーカス(公称値): 1.6 µm / 最小 デフォーカス(公称値): 0.8 µm |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 1.2 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
|---|---|
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 2.4 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 825204 |
 ムービー
ムービー コントローラー
コントローラー








 Z
Z Y
Y X
X