+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-23816 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the VRC310 clinical trial, vaccine-elicited, human antibody 310-030-1D06 Fab in complex with an H1 NC99 HA trimer | |||||||||
 マップデータ マップデータ | sharpened map | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 viral budding from plasma membrane / clathrin-dependent endocytosis of virus by host cell / host cell surface receptor binding / apical plasma membrane / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane / viral budding from plasma membrane / clathrin-dependent endocytosis of virus by host cell / host cell surface receptor binding / apical plasma membrane / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane /  エンベロープ (ウイルス) / virion attachment to host cell / host cell plasma membrane / virion membrane / エンベロープ (ウイルス) / virion attachment to host cell / host cell plasma membrane / virion membrane /  生体膜 生体膜類似検索 - 分子機能 | |||||||||
| 生物種 |    Influenza A virus (A型インフルエンザウイルス) / Influenza A virus (A型インフルエンザウイルス) /   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.87 Å クライオ電子顕微鏡法 / 解像度: 3.87 Å | |||||||||
 データ登録者 データ登録者 | Gorman J / Kwong PD | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Med / 年: 2022 ジャーナル: Nat Med / 年: 2022タイトル: A single residue in influenza virus H2 hemagglutinin enhances the breadth of the B cell response elicited by H2 vaccination. 著者: Sarah F Andrews / Julie E Raab / Jason Gorman / Rebecca A Gillespie / Crystal S F Cheung / Reda Rawi / Lauren Y Cominsky / Jeffrey C Boyington / Adrian Creanga / Chen-Hsiang Shen / Darcy R ...著者: Sarah F Andrews / Julie E Raab / Jason Gorman / Rebecca A Gillespie / Crystal S F Cheung / Reda Rawi / Lauren Y Cominsky / Jeffrey C Boyington / Adrian Creanga / Chen-Hsiang Shen / Darcy R Harris / Adam S Olia / Alexandra F Nazzari / Tongqing Zhou / Katherine V Houser / Grace L Chen / John R Mascola / Barney S Graham / Masaru Kanekiyo / Julie E Ledgerwood / Peter D Kwong / Adrian B McDermott /  要旨: Conserved epitopes on the influenza hemagglutinin (HA) stem are an attractive target for universal vaccine strategies as they elicit broadly neutralizing antibodies. Such antibody responses to stem- ...Conserved epitopes on the influenza hemagglutinin (HA) stem are an attractive target for universal vaccine strategies as they elicit broadly neutralizing antibodies. Such antibody responses to stem-specific epitopes have been extensively characterized for HA subtypes H1 and H5 in humans. H2N2 influenza virus circulated 50 years ago and represents a pandemic threat due to the lack of widespread immunity, but, unlike H1 and H5, the H2 HA stem contains Phe45 predicted to sterically clash with HA stem-binding antibodies characterized to date. To understand the effect of Phe45, we compared the HA stem-specific B cell response in post hoc analyses of two phase 1 clinical trials, one testing vaccination with an H2 ferritin nanoparticle immunogen ( NCT03186781 ) and one with an inactivated H5N1 vaccine ( NCT01086657 ). In H2-naive individuals, the magnitude of the B cell response was equivalent, but H2-elicited HA stem-binding B cells displayed greater cross-reactivity than those elicited by H5. However, in individuals with childhood H2 exposure, H5-elicited HA stem-binding B cells also displayed high cross-reactivity, suggesting recall of memory B cells formed 50 years ago. Overall, we propose that a one-residue difference on an HA immunogen can alter establishment and expansion of broadly neutralizing memory B cells. These data have implications for stem-based universal influenza vaccination strategies. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
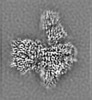
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_23816.map.gz emd_23816.map.gz | 108.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-23816-v30.xml emd-23816-v30.xml emd-23816.xml emd-23816.xml | 23.3 KB 23.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_23816.png emd_23816.png | 107.8 KB | ||
| マスクデータ |  emd_23816_msk_1.map emd_23816_msk_1.map | 115.9 MB |  マスクマップ マスクマップ | |
| その他 |  emd_23816_additional_1.map.gz emd_23816_additional_1.map.gz emd_23816_additional_2.map.gz emd_23816_additional_2.map.gz emd_23816_half_map_1.map.gz emd_23816_half_map_1.map.gz emd_23816_half_map_2.map.gz emd_23816_half_map_2.map.gz | 24.5 MB 22.5 MB 107.7 MB 107.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-23816 http://ftp.pdbj.org/pub/emdb/structures/EMD-23816 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23816 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23816 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_23816.map.gz / 形式: CCP4 / 大きさ: 115.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_23816.map.gz / 形式: CCP4 / 大きさ: 115.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | sharpened map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.07325 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_23816_msk_1.map emd_23816_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
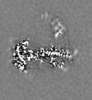
| 投影像・断面図 |
| ||||||||||||
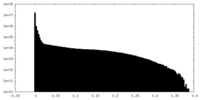
| 密度ヒストグラム |
-追加マップ: unsharpened map
| ファイル | emd_23816_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | unsharpened map | ||||||||||||
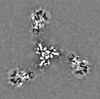
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: density modified with resolve
| ファイル | emd_23816_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | density modified with resolve | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half map A
| ファイル | emd_23816_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half map B
| ファイル | emd_23816_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : 310-030-1D06 Fab in complex with an H1 NC99 HA trimer
| 全体 | 名称: 310-030-1D06 Fab in complex with an H1 NC99 HA trimer |
|---|---|
| 要素 |
|
-超分子 #1: 310-030-1D06 Fab in complex with an H1 NC99 HA trimer
| 超分子 | 名称: 310-030-1D06 Fab in complex with an H1 NC99 HA trimer タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 |
|---|---|
| 由来(天然) | 生物種:    Influenza A virus (A型インフルエンザウイルス) Influenza A virus (A型インフルエンザウイルス) |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Hemagglutinin HA1 chain
| 分子 | 名称: Hemagglutinin HA1 chain / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:    Influenza A virus (A型インフルエンザウイルス) Influenza A virus (A型インフルエンザウイルス) |
| 分子量 | 理論値: 36.407852 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: DTICIGYHAN NSTDTVDTVC EKNVTVTHSV NLLEDSHNGK LCLLKGIAPL QLGNCSVAGW ILGNPECELL ISKESWSYIV ETPNPENGT CYPGYFADYE ELREQLSSVS SFERFEIFPK ESSWPNHTVT GVSASCSHNG KSSFYRNLLW LTGKNGLYPN L SKSYVNNK ...文字列: DTICIGYHAN NSTDTVDTVC EKNVTVTHSV NLLEDSHNGK LCLLKGIAPL QLGNCSVAGW ILGNPECELL ISKESWSYIV ETPNPENGT CYPGYFADYE ELREQLSSVS SFERFEIFPK ESSWPNHTVT GVSASCSHNG KSSFYRNLLW LTGKNGLYPN L SKSYVNNK EKEVLVLWGV HHPPNIGNQR ALYHTENAYV SVVSSHYSRR FTPEIAKRPK VRDQEGRINY YWTLLEPGDT II FEANGNL IAPWYAFALS RGFGSGIITS NAPMDECDAK CQTPQGAINS SLPFQNVHPV TIGECPKYVR SAKLRMVTGL RNI PSIQSR |
-分子 #2: Hemagglutinin HA2 chain
| 分子 | 名称: Hemagglutinin HA2 chain / タイプ: protein_or_peptide / ID: 2 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:    Influenza A virus (A型インフルエンザウイルス) Influenza A virus (A型インフルエンザウイルス) |
| 分子量 | 理論値: 25.263104 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: GLFGAIAGFI EGGWTGMVDG WYGYHHQNEQ GSGYAADQKS TQNAINCITN KVNSVIEKMN TQFTAVGKEF NKLERRMENL NKKVDDGFL DIWTYNAELL VLLENERTLD FHDSNVKNLY EKVKSQLKNN AKEIGNGCFE FYHKCNNECM ESVKNGTYDY P KYSEESKL ...文字列: GLFGAIAGFI EGGWTGMVDG WYGYHHQNEQ GSGYAADQKS TQNAINCITN KVNSVIEKMN TQFTAVGKEF NKLERRMENL NKKVDDGFL DIWTYNAELL VLLENERTLD FHDSNVKNLY EKVKSQLKNN AKEIGNGCFE FYHKCNNECM ESVKNGTYDY P KYSEESKL NREKIDGVSG RLVPRGSPGS GYIPEAPRDG QAYVRKDGEW VLLSTFLGHH HHHH |
-分子 #3: 310-030-1D06 Heavy
| 分子 | 名称: 310-030-1D06 Heavy / タイプ: protein_or_peptide / ID: 3 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 13.000457 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: QVQLVQSGAE VKKPGSSVKV SCKASGGTFS SYGISWVRQA PGQGLEWMGG IIGMFGTTNY AQKFQGRVTI TADEFTSTAY MELSSLRSE DTAVYYCARG GSYYVDYFHH WGQGTLVTVS S |
-分子 #4: 310-030-1D06 Light
| 分子 | 名称: 310-030-1D06 Light / タイプ: protein_or_peptide / ID: 4 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 11.658904 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: EIVLTQSPGT LSLFSGERAT LSCRASQSVS SSSLAWYQQK HGQGPRLIMY GASSRATGIP DRFSGSGFGT DFTITISRLE PEDFAVYYC QQYGSSSGTF GQGTKLEMK |
-分子 #6: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 6 / コピー数: 21 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 / 構成要素 - 式: PBS |
| 凍結 | 凍結剤: ETHANE |
| 詳細 | 310-030-1D06 Fab in complex with an H1 NC99 HA trimer |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 70.13 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
|---|---|
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 再構成 | アルゴリズム: FOURIER SPACE / 解像度のタイプ: BY AUTHOR / 解像度: 3.87 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: cryoSPARC / 使用した粒子像数: 36235 |
-原子モデル構築 1
| 精密化 | プロトコル: FLEXIBLE FIT |
|---|---|
| 得られたモデル |  PDB-7mfg: |
 ムービー
ムービー コントローラー
コントローラー






















 Z
Z Y
Y X
X